Übermittlung onkologischer Daten auf der Basis von CDA R2
Foemig (Diskussion | Beiträge) K |
Foemig (Diskussion | Beiträge) K |
||
| (7 dazwischenliegende Versionen desselben Benutzers werden nicht angezeigt) | |||
| Zeile 11: | Zeile 11: | ||
|Version = 0.9 | |Version = 0.9 | ||
|Submitted = . | |Submitted = . | ||
| − | |Date = | + | |Date = 12. Februar 2012 |
|Copyright = 2010-2013 | |Copyright = 2010-2013 | ||
|Status = Entwurf | |Status = Entwurf | ||
| Zeile 21: | Zeile 21: | ||
{{Infobox Contributors Begin}} | {{Infobox Contributors Begin}} | ||
{{Contributor | Logo = Logo-Dkg.jpg | Name = Deutsche Krebsgesellschaft | Location = Berlin }} | {{Contributor | Logo = Logo-Dkg.jpg | Name = Deutsche Krebsgesellschaft | Location = Berlin }} | ||
| − | {{Contributor | Logo = Logo-Uni-giessen.gif | Name = | + | {{Contributor | Logo = Logo-Uni-giessen.gif | Name = Institut für Medizinische Informatik der Universität Gießen | Location = Gießen }} |
{{Contributor | Logo = Logo-Agfa.jpg | Name = Agfa HealthCare GmbH | Location = Bonn }} | {{Contributor | Logo = Logo-Agfa.jpg | Name = Agfa HealthCare GmbH | Location = Bonn }} | ||
{{Contributor | Logo = Logo-Gkd.jpg | Name = GKD Gesellschaft für klinische Dienstleistungen Düsseldorf mbH | Location = Düsseldorf }} | {{Contributor | Logo = Logo-Gkd.jpg | Name = GKD Gesellschaft für klinische Dienstleistungen Düsseldorf mbH | Location = Düsseldorf }} | ||
| Zeile 42: | Zeile 42: | ||
{{HL7transclude|cdaonk:DynamischesModell}} | {{HL7transclude|cdaonk:DynamischesModell}} | ||
| − | {{HL7transclude|cdaonk: | + | {{HL7transclude|cdaonk:Statisches Modell}} |
| − | {{HL7transclude|cdaonk:Header}} | + | {{HL7transclude|cdaonk:Krebsregistermeldung}} |
| + | |||
| + | =Beteiligte (Header)= | ||
| + | {{HL7transclude|cdaonk:Patient (recordTarget) (Template)}} | ||
| + | {{HL7transclude|cdaonk:Sendendes System (author) (Template)}} | ||
| + | {{HL7transclude|cdaonk:Medizinischer Dokumentar (dataEnterer) (Template)}} | ||
| + | {{HL7transclude|cdaonk:Melder (informant) (Template)}} | ||
| + | {{HL7transclude|cdaonk:Verwaltende Organisation (custodian) (Template)}} | ||
| + | {{HL7transclude|cdaonk:Empfänger (informationRecipient) (Template)}} | ||
| + | {{HL7transclude|cdaonk:Kostenträger (participant) (Template)}} | ||
| − | + | =Abschnitte= | |
{{HL7transclude|cdaonk:ergänzende Patientendaten-Section (Template)}} | {{HL7transclude|cdaonk:ergänzende Patientendaten-Section (Template)}} | ||
{{HL7transclude|cdaonk:Meldebegründung-Section (Template)}} | {{HL7transclude|cdaonk:Meldebegründung-Section (Template)}} | ||
| − | |||
{{HL7transclude|cdaonk:Diagnostik-Section (Template)}} | {{HL7transclude|cdaonk:Diagnostik-Section (Template)}} | ||
{{HL7transclude|cdaonk:Morphologie-Entry (Template)}} | {{HL7transclude|cdaonk:Morphologie-Entry (Template)}} | ||
{{HL7transclude|cdaonk:Frühere Tumorerkrankung-Entry (Template)}} | {{HL7transclude|cdaonk:Frühere Tumorerkrankung-Entry (Template)}} | ||
{{HL7transclude|cdaonk:Ann-Arbor-Stadium-Entry (Template)}} | {{HL7transclude|cdaonk:Ann-Arbor-Stadium-Entry (Template)}} | ||
| − | {{HL7transclude| | + | {{HL7transclude|cdaab2:Residualtumor-Entry (Template)}} |
| + | {{HL7transclude|cdaab2:TNM-Klassifikation-Entry (Template)}} | ||
{{HL7transclude|cdaonk:TNM-Klassifikation-Entry (Template)}} | {{HL7transclude|cdaonk:TNM-Klassifikation-Entry (Template)}} | ||
{{HL7transclude|cdaonk:Generische Observation mit Messwert-Entry (Template)}} | {{HL7transclude|cdaonk:Generische Observation mit Messwert-Entry (Template)}} | ||
| Zeile 61: | Zeile 70: | ||
{{HL7transclude|cdaonk:Generische Observation mit codierter Angabe-Entry (Template)}} | {{HL7transclude|cdaonk:Generische Observation mit codierter Angabe-Entry (Template)}} | ||
{{HL7transclude|cdaonk:Gleason-Score-Entry (Template)}} | {{HL7transclude|cdaonk:Gleason-Score-Entry (Template)}} | ||
| + | {{HL7transclude|cdaab2:Elston-Ellis-Score-Entry (Template)}} | ||
| + | {{HL7transclude|cdaonk:Breslow-Score-Entry (Template)}} | ||
{{HL7transclude|cdaonk:Erkrankungsdaten-Section (Template)}} | {{HL7transclude|cdaonk:Erkrankungsdaten-Section (Template)}} | ||
Aktuelle Version vom 28. November 2013, 15:57 Uhr
|
|
Dieses Dokument gibt wieder:
Implementierungsleitfaden Übermittlung onkologischer Daten auf der Basis von CDA R2 (0.9). Die Teilmaterialien gehören der Kategorie cdaonk an. |
HL7 Clinical Document Architecture Release 2
für das deutsche Gesundheitswesen
.
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
 |
HL7-Benutzergruppe in Deutschland e. V. |
Copyright © 2012/2013: HL7 Benutzergruppe in Deutschland e.V.
HL7-Benutzergruppe in Deutschland e.V.
Geschäftsstelle Köln
An der Schanz 1
50735 Köln
Abstimmung durch:
Mitglieder der HL7-Benutzergruppe e.V.
Ansprechpartner:
Dr. Frank Oemig, Agfa HealthCare GmbH (Bonn)
Inhaltsverzeichnis
- 1 Dokumentenhistorie
- 2 Editor
- 3 Autoren
- 4 Mit Beiträgen von
- 5 Nachnutzungs- bzw. Veröffentlichungsansprüche
- 6 Disclaimer
- 7 Einleitung
- 8 Szenarien
- 9 Domänen-Analyse Modell
- 10 Dynamisches Modell
- 11 Statisches Modell
- 11.1 Einleitung
- 11.2 Grundsätzliche Anforderungen an die Dokumentstruktur
- 11.3 Beispiel für groben Aufbau
- 11.4 Identifikation von Informationseinheiten
- 11.5 Referenzen auf andere Informationseinheiten
- 11.6 Referenzen auf Beteiligte
- 11.7 Handhabung von Negationen
- 11.8 Handhabung von Korrekturen und Folgemeldungen
- 11.9 Meldeanlässe und Inhalte
- 11.10 offene Punkte
- 12 Dokumentenstruktur Krebsregistermeldung
- 13 Beteiligte (Header)
- 13.1 Participant: Patient (recordTarget)
- 13.2 Participant: Sendendes System (author)
- 13.3 Participant: medizinischer Dokumentar (dataEnterer)
- 13.4 Participant: Arzt, Melder (informant)
- 13.5 Participant: verwaltende Organisation (custodian)
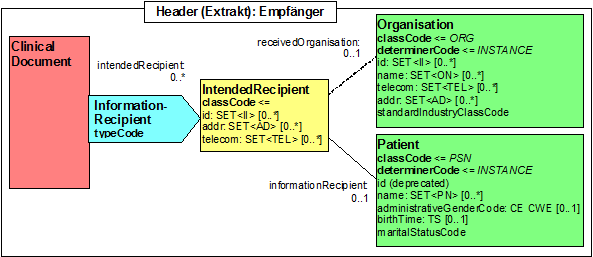
- 13.6 Participant: Empfänger (informationRecipient)
- 13.7 Versicherungsnehmer und Kostenträger
- 14 Abschnitte
- 14.1 Section: Ergänzende Patientendaten
- 14.2 Section: Meldebegründung
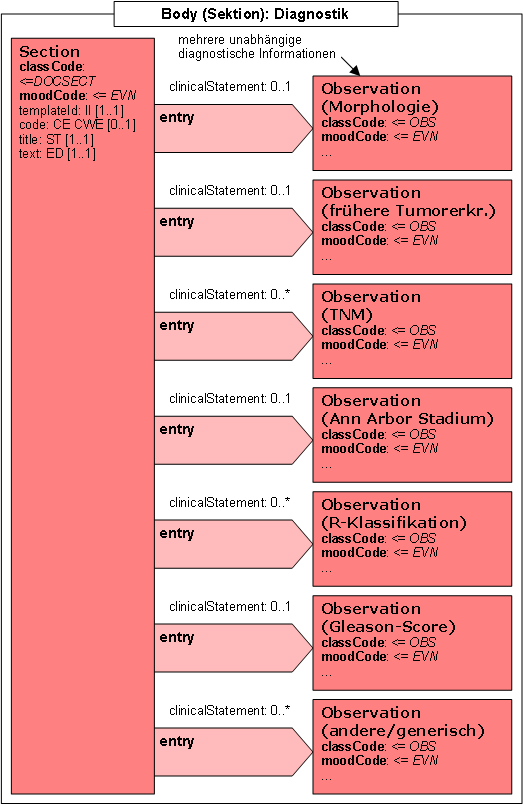
- 14.3 Section: Diagnostik
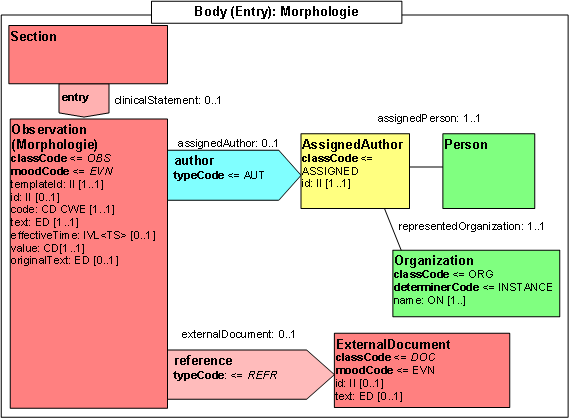
- 14.4 Entry: Morphologie
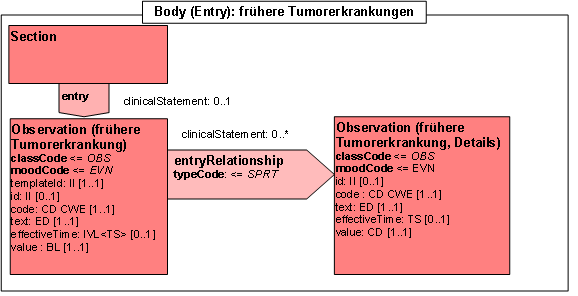
- 14.5 Entry: frühere Tumorerkrankungen
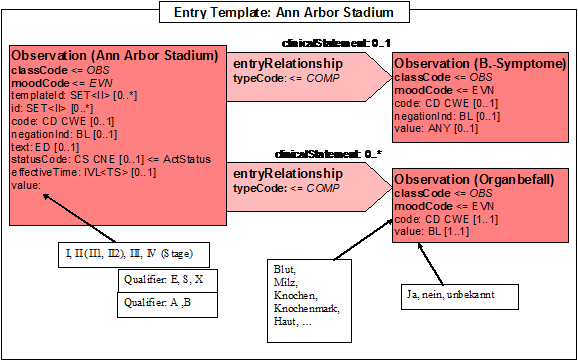
- 14.6 Entry: Ann Arbor Stadium
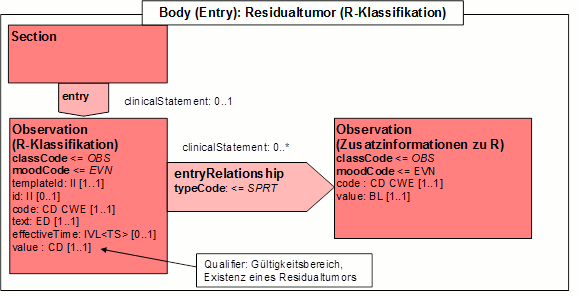
- 14.7 Entry: R-Klassifikation
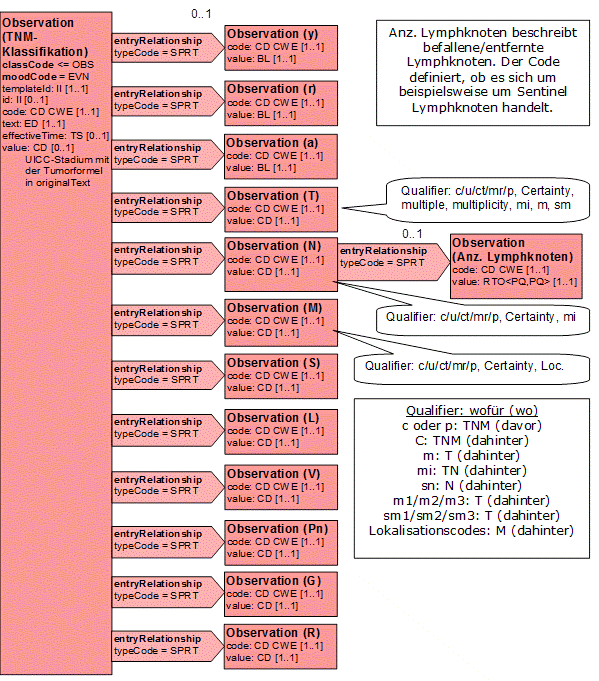
- 14.8 Entry: TNM-Klassifikation
- 14.8.1 Darstellung der TNM-Klassifikation in HL7
- 14.8.2 Erläuterung
- 14.8.2.1 TNM-Klassifikation UICC-Stadien)
- 14.8.2.2 TNM y-Symbol (Präfix)
- 14.8.2.3 TNM r-Symbol (Präfix)
- 14.8.2.4 TNM a-Symbol (Präfix)
- 14.8.2.5 T: Tumor
- 14.8.2.6 Art der Sicherung (qualifier)
- 14.8.2.7 C-Faktor (Certainty) (qualifier)
- 14.8.2.8 m-Symbol: Multiplizität (multiple tumor) (qualifier)
- 14.8.2.9 Multiplizität (numerisch): Anzahl der Tumorherde (qualifier)
- 14.8.2.10 Mucosainfiltration (qualifier)
- 14.8.2.11 Submucosainfiltration (qualifier)
- 14.8.2.12 N: Nodalstatus
- 14.8.2.13 Art der Sicherung (qualifier)
- 14.8.2.14 C-Faktor (Certainty)
- 14.8.2.15 i (Suffix): ITC durch Immunhistochemie (qualifier)
- 14.8.2.16 mol (Suffix): ITC durch Molekularpathologie
- 14.8.2.17 Anzahl Lymphknoten
- 14.8.2.18 M: Metastasen
- 14.8.2.19 Art der Sicherung (qualifier)
- 14.8.2.20 C-Faktor (Certainty)
- 14.8.2.21 i (Suffix): ITC durch Immunhistochemie (qualifier)
- 14.8.2.22 mol (Suffix): RT-PCR (ITC durch Molekularpathologie) (qualifier)
- 14.8.2.23 S: Serumtumormarker
- 14.8.2.24 L: Lymphgefäßinvasion
- 14.8.2.25 V: Veneninvasion
- 14.8.2.26 Pn: Perineuralinvasion
- 14.8.2.27 G: histopathologisches Grading
- 14.8.2.28 R: Residualtumor
- 14.8.3 Beispiel
- 14.8.4 verwendete Terminologie
- 14.8.4.1 Stadiengruppierung nach UICC
- 14.8.4.2 Ausdehnung des Primärtumors (T)
- 14.8.4.3 Nodalstatus (regionäre Lymphknoten) (N)
- 14.8.4.4 Fernmetastasen (M)
- 14.8.4.5 Veneninvasion
- 14.8.4.6 Lymphgefäßinvasion
- 14.8.4.7 Perineuralscheideninvasion
- 14.8.4.8 Unterscheidung autoptisch/klinisch/pathologisch
- 14.8.4.9 Beschränkung auf Mukosa
- 14.8.4.10 Beschränkung auf Submukosa
- 14.8.4.11 C-Faktor (Certainty, Diagnosesicherheit)
- 14.8.4.12 Lokalisation von Fernmetastasen
- 14.9 Entry: TNM-Klassifikation (zur Übermittlung onkolog. Daten)
- 14.10 Entry: Generische Observation mit Messwert
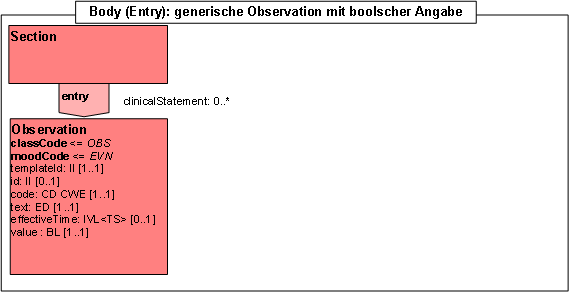
- 14.11 Entry: Generische Observation mit boolscher Angabe
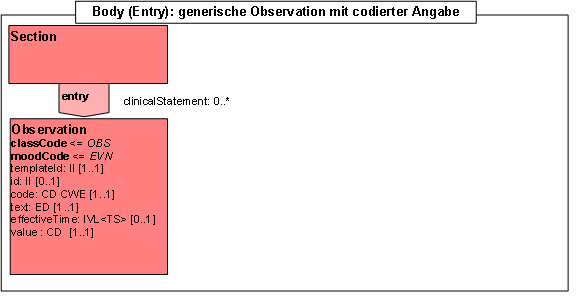
- 14.12 Entry: Generische Observation mit codierter Angabe
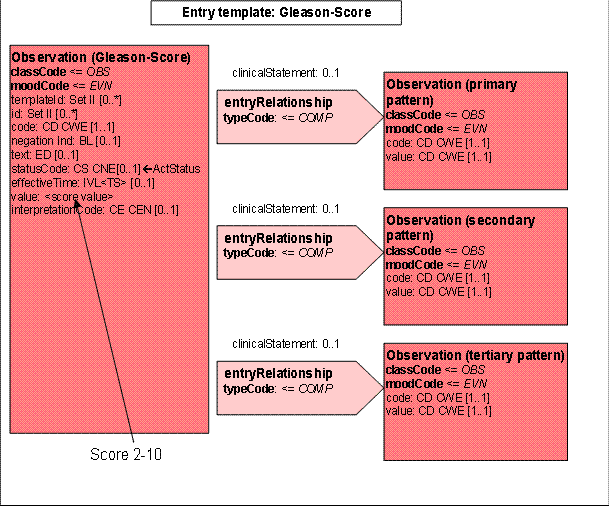
- 14.13 Entry: Gleason-Score
- 14.14 Elston-Ellis-Score Entry (Template) (Nottingham-Grading)
- 14.15 Breslow-Index Entry (Template)
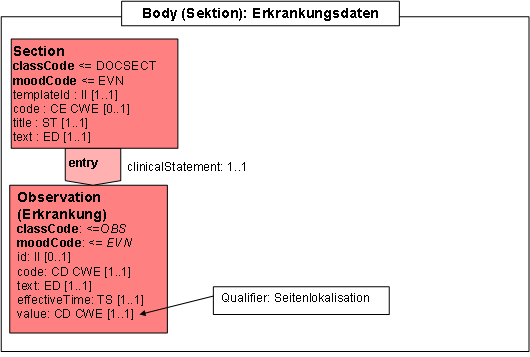
- 14.16 Section: Erkrankungsdaten
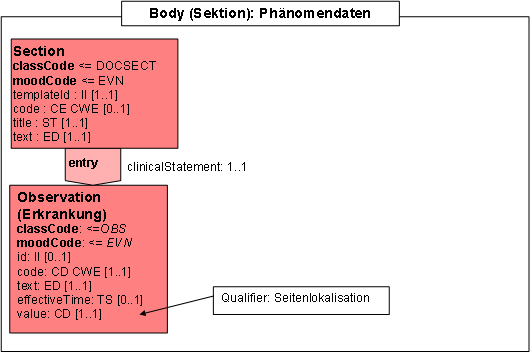
- 14.17 Section: Phänomendaten
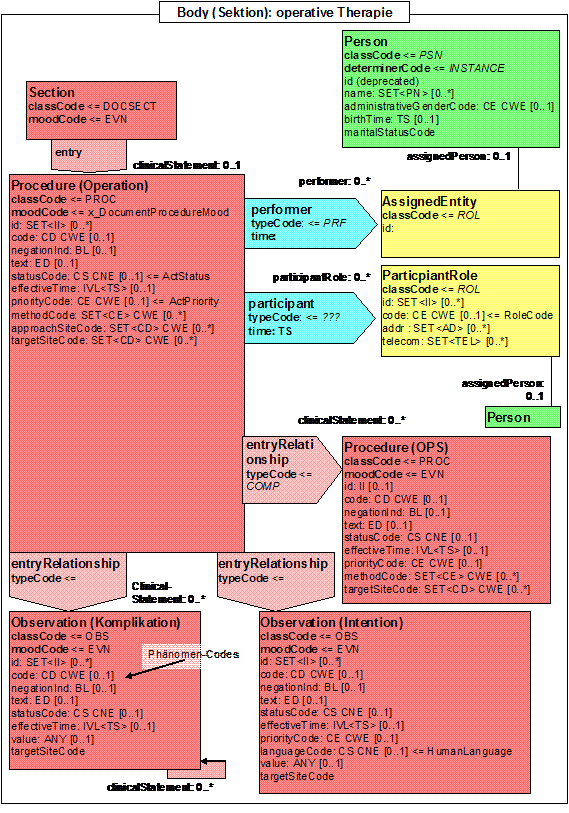
- 14.18 Section: Operation (operative Therapie)
- 14.19 Section: Bestrahlung
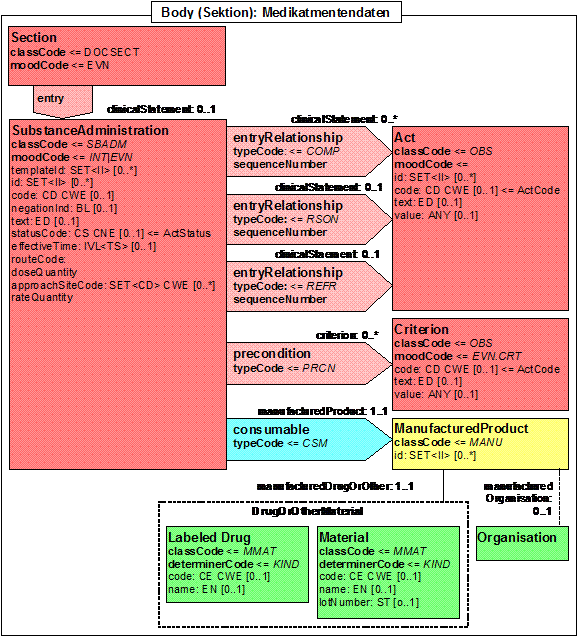
- 14.20 Section: Medikation
- 14.21 Section: Studiendaten
- 14.22 Section: Abschlussdaten
- 14.23 Section: Todesursache
- 14.24 Section: Planung
- 15 Anhang A: Diverses
- 16 Anhang B: Verzeichnisse
- 16.1 Codesysteme
- 16.1.1 DKG-Labels
- 16.1.2 GEKID Meldebegründung
- 16.1.3 GEKID Diagnosesicherung
- 16.1.4 GEKID Diagnoseanlass
- 16.1.5 GEKID Grobstadium
- 16.1.6 Art des Krankenhausaufenthalts
- 16.1.7 Allgemeiner Leistungszustand, ECOG
- 16.1.8 TLS - Tumorlokalisationsschlüssel (ICD-O-2 deutsche Erweiterung)
- 16.1.9 Untersuchungsanlass
- 16.1.10 Remissionsstatus
- 16.1.11 Status Primärtumor
- 16.1.12 Status regionär
- 16.1.13 Status Fernmetastasen
- 16.1.14 Therapiekategorien
- 16.1.15 Komplikationen
- 16.1.16 Zielgebiet der Strahlentherapie (Zielgebietsschlüssel, BDT 5. Aufl. - Version B4)
- 16.1.17 Applikationsart der Strahlentherapie
- 16.1.18 Therapieintention
- 16.1.19 Beendigungsstatus der Therapie
- 16.1.20 WHO-ART 1999 (Basisdokumentation für Tumorkranke, 5. Auflage)
- 16.1.21 RTOG Toxicity Criteria
- 16.1.22 WHO-ART 1999 (Basisdokumentation für Tumorkranke, 5. Auflage) - Grad
- 16.1.23 RTOG Toxicity Criteria - Grad
- 16.1.24 Risikokategorien bei trophoblastären Schwangerschaftstumoren (C58)
- 16.1.25 Boolsche Werte für Datentyp CD
- 16.1.26 zeitliche Relation der Therapie zur Operation
- 16.1.27 Gültigkeitsbereich der Patientenzustimmung
- 16.2 Value Sets
- 16.3 OIDs für Templates
- 16.4 ICD-Kodes
- 16.5 OPS-Kodes
- 16.6 Besonderheit ab Version 2.6
- 16.7 Für die Zukunft ...
- 16.1 Codesysteme
- 17 Referenzen
Dokumentenhistorie
| Version | Stand | Bearbeiter | Beschreibung | Dok.-OID |
|---|---|---|---|---|
| 01 | 22.10.10 | FO | Draft | n.a. |
| 02 | 04.11.10 | FO | Erweiterung | n.a. |
| 03 | 05.11.10 | BS | Erweiterung | n.a. |
| 04 | 11.11.10 | FO | Einarbeitung erste Kommentare | n.a. |
| 05 | 12.11.10 | FO | weitere Überarbeitung | n.a. |
| 06 | 20.01.11 | FO | weitere Überarbeitung | n.a. |
| 07 | 27.01.11 | FO | weitere Überarbeitung | n.a. |
| 08 | 21.03.11 | FO | weitere Überarbeitung | n.a. |
| 09 | 09.07.11 | FO | Umstrukturierung, OIDs, Layout | n.a. |
| 10 | 02.05.12 | FO | Wiki | n.a. |
Editor
- Dr. Frank Oemig, Agfa HealthCare GmbH (Bonn)
Autoren
- Dr. Frank Oemig, Agfa HealthCare GmbH, Bonn (FO)
- Stefan Lang, Lang Health IT Consulting (SL)
Mit Beiträgen von
- Dr. Udo Altmann, GTDS und Forum Klinischer Krebsregister, Gießen (UA)
- Esther Amenda, ix.mid, Köln (EA)
- Silke Fontein, megapharm (SF)
- Stefan Lang, Lang Health IT Consulting (SL)
- Matthias Schmitz, Agfa HealthCare, Bonn (MS)
- Dr. Bernd Schütze, Univ.-Kl., Düsseldorf (BS)
- Claas Thiele, Zytoservice Deutschland GmbH, Hennef (CT)
Folgende Organisationen beteiligen sich aktiv an der Diskussion und der Arbeitsgruppe:
- Deutsche Krebsgesellschaft e.V.
- Deutsche Onkologie Centrum Holding GmbH (DOC)
- Gesellschaft der epidemiologischen Krebsregister in Deutschland e.V. (GEKID)
- Gießener Tumordokumentationssystem (GTDS)
- Arbeitsgemeinschaft Deutscher Tumorzentren e.V. (ADT)
- Agfa Healthcare GmbH, Bonn
- GKD Gesellschaft für klinische Dienstleistungen Düsseldorf mbH
- ix.mid, Köln
- Lang Health IT Consulting, Ehringshausen
- megapharm GmbH
- Zytoservice, Hennef
Autoren und Copyright-Hinweis, Nutzungshinweise
Nachnutzungs- bzw. Veröffentlichungsansprüche
Das vorliegende Dokument wurde von der Projektgruppe „onkologische Datenübermittlung" in Kooperation mit der HL7-Benutzergruppe e.V. entwickelt. Die Nachnutzungs- bzw. Veröffentlichungsansprüche sind nicht beschränkt.
Der Inhalt dieser Spezifikation ist öffentlich.
Zu beachten ist, dass Teile dieses Dokuments auf dem HL7-Standard CDA beruhen, für die © Health Level Seven, International gilt. Näheres unter hl7.de und hl7.org.
Die Erweiterung oder Ablehnung der Spezifikation, ganz oder in Teilen, ist dem Vorstand der Benutzergruppe und den Editoren/Autoren schriftlich anzuzeigen.
Alle auf nationale Verhältnisse angepassten und veröffentlichten HL7-Spezifkationen können ohne Lizenz- und Nutzungsgebühren in jeder Art von Anwendungssoftware verwendet werden.
Disclaimer
Obwohl diese Publikation mit größter Sorgfalt erstellt wurde, kann weder die HL7-Benutzergruppe in Deutschland e.V. noch die an der Erstellung beteiligten Firmen keinerlei Haftung für direkten oder indirekten Schaden übernehmen, die durch den Inhalt dieser Spezifikation entstehen könnten.
Des Weiteren werden nach Abschluss des Abstimmungsverfahrens den diversen Tabellen noch ihre endgültige OID zugewiesen.
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Einleitung
Hintergrundinformationen
Im Sommer 2008 traf sich eine kleine Arbeitsgruppe (DOC, ADT, Agfa, Siemens, megapharm, alcedis) bei der DOC Holding in Düsseldorf, um die Optimierung der Datenübertragung in der onkologischen Versorgung zu besprechen. Hintergrund ist die in jedem Bundesland unterschiedliche Spezifikation der epidemiologischen Krebs¬register sowie die verschiedenen Anforderungen der Daten¬übermittlung an klinische Krebsregister und Dienstleister aus der Qualitätssicherung, die zu Problemen und hohen Aufwendungen bei der Implementierung führen. Eine weitere Fragestellung ist die Vereinfachung der Datenübermittlung in der onkologischen Forschung.
Projekthistorie
Die Historie dieses Projektes ist im Anhang aufgeführt.
Scope
Ziel soll es sein, eine modellbasierte Grundlage zu schaffen, um die Daten¬übertragung innerhalb der Onkologie auf einen Standard abzubilden. Daten werden auf verschiedenen Ebenen gesammelt und kommuniziert. Die klinische Betreuung erfolgt noch weitestgehend gestützt auf Papier- oder unstrukturierten elektronischen Dokumenten. In strukturierter Form werden Daten jedoch in der Qualitätssicherung, den klinischen und epidemiologischen Krebsregistern sowie den Organzentren benötigt. Es existiert eine Reihe von Spezifikationen – teilweise bedingt durch gesetzliche Vorgaben auf Landes- und Bundesebene , die einen unterschiedlichen organisatorischen Kontext besitzen und bei ähnlichen Inhalten nicht unbedingt deckungsgleich sind. Praktisch führt dies zu Mehrfachdokumentation und / oder aufwendigen technischen Systemen.
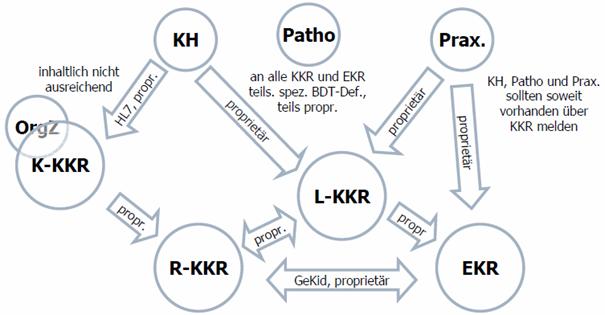
Abbildung 1: gegenwärtige Situation
Die vorhergehende Abbildung verdeutlicht die gegenwärtige Situation. Demnach gilt es folgende Spezifikationen abzubilden/abzulösen:
- Gemeinsamer onkologischer Basisdatensatz (ADT, GeKiD, DKG, KoQK, CCC Forum) auf Basis von XML
- Schnittstellen KKR / EKR (GeKiD)
- ONkeyLINE
- Datensatz Onkologie
- Spezielle Programme: BQS, DMP Mamma, QS / Benchmarking, ..
Als Grundproblem dieser Spezifikationen kann festgehalten werden, dass Prozesse und Rahmenbedingungen nicht ausreichend beachtet bzw. definiert worden sind: Anlass und Frequenz für Informationsfluss (Meldung), sinnvolle Teilmengen/Optionalitäten, lokale Besonderheiten (z.B. Landesgesetze), Verschlüsselung, technischer Transport, Verar¬beitungs¬status, Rückmeldung.
Als Idealfall ist anzustreben, dass jeder an der Behandlung beteiligte seinen inhaltlichen Anteil am Standard einmalig dokumentiert und dabei allenfalls Zusatzinformationen hinterlegt (wie die Zuordnung zu einer bestimmten Erkrankung oder therapeutischen Situation), die es erlauben, diese Informationen in übergeordnetem Kontext (Register, QS, nachbetreuende Behandler) korrekt einzuordnen.
Dieser Leitfaden zielt darauf ab, geeignete Dokumentenstrukturen für die einzelnen Meldungen auf Basis von Komponenten (Content Modules) zu spezifizieren, die die genannten Anforderungen abbilden können.
Basisdokumente
Dieses Dokument versucht die in den nachfolgenden Dokumenten aufgeführten Anforderungen umzusetzen:
- VHitG-Arztbrief (OID ???????)
- Header-Information
- Darstellung von Ärzten, Adressen, Namen, ..
- Grundlagen von Diagnosen und Prozeduren
- Diagnoseleitfaden (OID ??????)
- verfeinerte Diagnose-Information, ICD-O, TNM
- Liste der bisherigen Spezifikationen:
- XML-/CSV-Datensatz des Kooperationsverbundes Qualitätssicherung durch Klinische Krebsregister (KoQK) bzw. der Arbeitsgemeinschaft Deutscher Tumorzentren e.V. (ADT)
- XML-/CSV-Datensatz der Gesellschaft der epidemiologischen Krebsregister in Deutschland e.V. (GEKID)
- CSV-Datensatz des Instituts für angewandte Qualitätsförderung und Forschung im Gesundheitswesen GmbH (AQUA)
- XML-Datensätze der Deutsche Onkologie Centrum Holding GmbH
- Liste der noch nicht berücksichtigten Spezifikationen:
- CSV-Datensatz des GEKID-Datensatzes der Bundesgeschäftsstelle Qualitäts¬sicherung gGmbH (BQS)
- CSV-Datensatz der Gesellschaft für Pädiatrische Onkologie und Häma¬tologie (GPOH)
- BDT-Datensatz des Zentralinstituts für Kassenärztliche Versorgung (ZI)
- XML-Datensatz der Kassenärztlichen Bundesvereinigung (KBV)
- CDA2-Datensatz der Ärztekammer Westfalen-Lippe in Kooperation mit der Bundesgeschäftsstelle Qualitätssicherung gGmbH (BQS)
- XML-Datensatz der Arbeitsgruppe Tumordatenschnittstellen Niedersachsen
Aufbau
Nach dieser Einleitung werden die abzuhandelnden Szenarien beschrieben. Danach erfolgt eine Erläuterung des Analysemodells, das anhand der bisherigen Datensatzbeschreibungen und der dahinter liegenden medizinischen Informationen erarbeitet wurde. Daran schließen sich das dynamische Modell sowie das statische Modell mit der Definition der CDA-Strukturen (Header + Body) an. Im Anhang befinden sich Referenzen, eine offene Punkteliste, Verzeichnisse etc.
HL7 und Referenz-Modelle
Wie später noch weiter ausgeführt hat die Gruppe entschieden, die Umsetzung auf Basis von HL7 CDA zu realisieren. Daher sind die Basisspezifikationen des VHitG-Arztbriefes sowie des Diagnoseleitfadens relevant. Für hilfreiche Erläuterungen sei auf die entsprechenden Dokumente verwiesen.
Konzept und Begründung
Hier folgt das Konzept mit der dazu passenden Begründung des Leitfadens. Inhalt sollten der Zweck, die beteiligten Organisationen, ihre Rollen bei der Erstellung des Leitfadens und der Fokus des Dokuments sein.
Zweck
Dieser Leitfaden soll primär die Erst- und Folgemeldungen an die klin. und epid. Krebs¬register spezifizieren.
Fokus
Dieser Leitfaden konzentriert sich auf die Umsetzung des Analysemodells (s.u.) in HL7 V3 CDA R2.
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Szenarien
Beispielfälle zum Ablauf von Diagnostik und Therapie
Fall 1: Kolorektales Karzinom
Die nachfolgende Tabelle analysiert die Abläufe am Beispiel eines kolorektalen Karzinoms:
| Zeitachse | Aktion / Ergebnis / Ereignis (mögliche Spezialfälle) | Akteur | Entstehende Daten (kursiv Daten außerhalb Basisdaten) | Mögliche Empfänger (weitere siehe auch Anmerkungen am Tabellenende) | Abstraktes Ereignis
Nach dem Muster Grundereignis (– Subspezifikationen) |
|---|---|---|---|---|---|
| 15.01.09 | Koloskopie im Rahmen von Früherkennung Feststellen eines malignitätsverdächtigen Polypen im Colon descendens, Entnahme einer Biopsie |
Niedergelassener Internist | Qualitätsmerkmal vollständige Koloskopie und Polypektomie | Biopsie -> Pathologen | Prätherapeutische Diagnostik – klinische Diagnosesicherung |
| 16.01.09 | Pathobefund: Adenokarzinom | Pathologen | Histologie Lokalisation |
Niedergelassener Internist KFRG (d) |
Prätherapeutische Diagnostik – Pathologie |
| 18.01.09 | Eintreffen des Pathobefundes Adenokarzinom, Infiltration Muscularis propria, Besprechen mit Patienten und Einweisung ins Krankenhaus | Niedergelassener Internist | Diagnosedatum (Def. beachten!) Erfassungsanlaß |
chirurg. Abt. (KH / Darmzentrum) KFRG (a) |
Prätherapeutische Diagnostik |
| 25.01.09 | Aufnahme Röntgenthorax und CT-Abdomen ohne Hinweis auf Metastasen | chirurg. Abt. (KH / Darmzentrum) | Klinischer TNM | - | Prätherapeutische Diagnostik – klinisches Staging |
| 26.01.09 | (prä op Konferenz, z.B. Lebermetastase) | Alle potentiell beteiligten Disziplinen / ambulant oder stationär | Entscheidung im Freitext bzw. Vorgesehene Therapien (Basisdatensatz hat kein Konferenzmodul) |
- | Therapieplanung - prätherapeutisch |
| 27.01.09 | Hemikolektomie | Chirurgische Abteilung | OPS (vor allem 5-... ) OP-Datum (Zusätze wie TME, ...) |
entnommenes Gewebe -> Pathologen KFRG (c) |
Operative Therapie – Tumorresektion - Primärtumor (kann mehrfach vorkommen bei Nachresektionen oder wegen unterschiedlicher OP-Bereiche wie Lymphknoten oder Metastasen) |
| 30.01.09 | Pathobefund: pT2pN1 G2 L0 V0 UICC III, Adenokarzinom, R0 (lokal radikal operiert) |
Pathologe | postoperativer TNM Tumorfreiheit |
chirurg. Abt. (KH / Darmzentrum) KFRG (d) |
Pathologisches Staging |
| 31.01.09 | Tumorkonferenz Empfehlung: Adjuvante Radiochemotherapie |
Interdisziplinär (Chirurg, Onkologe klin. und niedergelassen, Strahlentherapeut, ggf. Gyn, Uro, ...) |
(Vorgesehene Therapien) (ggf. Zuweisung eines Nachsorgeplans) |
Externe Strahlentherapie | Therapieplanung – postoperativ / im weiteren Sinne posttherapeutisch |
| 02.02.09 | Schmerzen, Darmanastomoseninsuffizienz, konservativ behandelt | Chirurgische Abteilung | Komplikation | Unerwünschte Therapiefolge
- Komplikation, kurzfristige oder langfristige Nebenwirkung | |
| (Revisionsop) | Operative Therapie – nicht resezierend | ||||
| 01.03.09 | Beginn der Radiochemotherapie | Externe Strahlentherapie (ambulante Durchführung), zubereitender Apotheker | Beginn Strahlentherapie Art Zielgebiet Intention Beginn Chemo Protokoll |
KH /Darmzentrum KFRG (c) |
Einleitung einer längerdauernden Therapie – Strahlentherapie und Chemotherapie |
| 15.04.09 | Ende der Strahlentherapie | Externe Strahlentherapie (ambulante Durchführung), zubereitender Apotheker | Ende Strahlentherapie Ende Status Dosis Ende Chemo Ende Status Dosierung |
KH / Darmzentrum KFRG (c) |
Beendigung einer längerdauernden Therapie – Strahlentherapie und Chemotherapie |
| 15.05.09 | Strahlentherapienachsorge Leichte Hautrötung |
Externe Strahlentherapie | Nebenwirkung | - | Therapiebezogene Nachsorge – ohne / mit gleichzeitiger Feststellung eines Therapieerfolges |
| 31.07.09 | Tumornachsorge: Beschwerdefreiheit, kein Anhalt für Rezidiv | Niedergelassener Arzt | Verlaufsdaten => Tumorfreiheit | - | Follow-up - Tumornachsorge |
| (Schmerzen, Verwachsungen) | Niedergelassener Arzt | (Verlaufsdaten => Folgeerkrankungen sind nicht mehr in aktuellem Basisdatensatz enthalten) | - | Follow-up - Folgeerkrankung des Tumors oder der Tumortherapie möglicherweise Zielereignis bei bestimmten Fragestellungen | |
| 31.10.09 | Tumornachsorge, metastasenverdächtiger Rundherd in der Leber | Niedergelassener Arzt | Verlaufsdaten => Metastasenlok. | KFRG (b) | Follow-up - Rezidiv / Progress |
| 7.11.09 | Resektion der Lebermetastase | Chirurgische Abteilung | OPS OP-Datum |
KFRG (c) | Operative Therapie – Tumorresektion –Rezidiv |
| 10.11.09 | Pathobefund: Metastase eines Adenoca. Im Gesunden reseziert | Chirurgische Abteilung / Pathologe | Tumorfreiheit | KFRG (d) | Follow-up - Therapieerfolg |
| 12.11.09 | Tumorkonferenz Empfehlung: Adjuvante Chemotherapie |
Interdisziplinär | (Vorgesehene Therapien) | - | Therapieplanung – postoperativ / im weiteren Sinne posttherapeutisch |
| 01.12.09 | Einleitung Chemotherapie | Onkologische Abteilung, zubereitender, Apotheker | Beginn Chemo Intention Protokoll |
KFRG (c) | Einleitung einer längerdauernden Therapie – Strahlentherapie und Chemotherapie |
| 15.01.10 | 2. Zyklus | Niedergelassener Onkologe, zubereitender Apotheker | |||
| ... | ... | ... | ... | ... | ... |
| 15.07.10 | Aufnahme Second-Line-Studie | Niedergelassener Onkologe, zubereitender Apotheker | Aufnahme in Studie Beginn Chemo Intention Protokoll Studienname / Studiennummer |
KFRG (c) | Einleitung einer längerdauernden Therapie – Strahlentherapie und Chemotherapie |
| 15.01.11 | Tod im Krankenhaus an genereller Metastasierung | Medizinische Abteilung | Sterbedatum Krebs/Tod Relation (Leichenschauschein) |
KFRG (b) | Tod |
| 16.01.11 | Autopsie | Pathologie | Krebs/Tod Relation | KFRG (d) | "Follow-up" - Autopsie |
Tabelle 1: Beispielszenario kolorektales Karzinom
- ggf. Organzentrum aus KKR
- ggf. KKR aus EKR
Darmzentrum steht exemplarisch für Organkrebszentrum. Das Organkrebszentrum kann seine Dokumentation unter Umständen auch weitgehend in einem klinischen Register abbilden.
Anmerkungen:
Unklar ist, auf welcher Ebene ein QS Empfänger ist. Zum Teil werden umfassende Daten über den gesamten Behandlungsprozess benötigt. Denkbar wäre eine Ausleitung auf Ebene des jeweiligen Akteurs oder je nachdem auf Ebene der klinischen Register bzw. Organzentren.
Generell sind die genannten Informationen in der Regel auch Bestandteil der ärztlichen Kommunikation, die zur Zeit nicht strukturiert und meist papierbezogen erfolgt. Es stellt sich daher die Frage der Unterstützung des Informationsflusses außerhalb der o.g. Dokumentationssysteme (Organkrebszentrum, klin. und epid. Register). So könnten sie auch als CDA-Dokumente oder in diesen integriert übermittelt werden und Bestandteil der jeweiligen Akte werden, was in diesem Fall auch relevant für Architektur der beteiligten Anwendungssysteme ist.
Follow-up steht für jegliche Art von Verlaufsinformation. Es kann sich um ein geplantes Follow-up im Sinne einer gezielten Nachsorge bei Tumorfreiheit oder Über¬wachungs¬maßnahme bei fortbestehender Erkrankung handeln oder um ein spontanes Ereignis, das den Patienten zum Arzt geführt hat oder das als Nebenbefund bei einer anderen Erkrankung festgestellt wurde (z.B. Lungenmetastase bei Röntgenuntersuchung im Rahmen einer Lungen¬entzündung). Je nach Befund werden ggf. weitere Details erforderlich (z.B. Angabe des Orts der Metastase bei Metastasierung).
Fall 2: Rezidiv eines Mammakarzinoms
Dieser Fall dient exemplarisch der Abbildung des Ablaufs bei Quereinsteigern (Fällen, die nicht komplett seit Diagnosestellung in den beteiligten Systemen geführt wurden). Es werden keine neuen Ereignisse abstrahiert.
| Zeitachse | Aktion / Ergebnis / Ereignis | Akteur | Daten |
|---|---|---|---|
| 15.01.09 | Lokal tastbarer Tumor in Restbrust bei Z.n. Mammaca Januar 2008, behandelt in Brustzentrum A |
Niedergelassener Gynäkologe | |
| 18.01.09 | Ambulante Stanzbiopsie Bestätigung des Verdachts auf Lokalrezidiv durch Pathologen |
Brustzentrum B Pathologe | Anamnestisch Diagnosedatum Histologie |
| 25.01.09 | Mastektomie | Gynäkologische Abteilung | OPS OP-Datum |
| 27.01.09 | Pathobefund rT2N0Mo | Chirurgische Abteilung | postoperativer rTNM Tumorfreiheit |
| (weiter vergleichbar Fall 1) …. |
Tabelle 2: Beispielszenario Rezidiv eines Mammakarzinoms
Wesentlich an diesem Fall ist, dass eine umfangreiche Nacherfassung erfolgen muss.
Steuerung
Hier geht es um Beispiele von Frage – Anforderung / Antwort – Ergebnismitteilung. Diese sind (auf Papier) gängige Praxis in klinischen Krebsregistern.
1.) Abfrage eines bestimmten geplanten Ereignisses (Nachsorge, Durchführung einer Therapie,…) mit Rückantwort
2.) Abfrage des aktuellsten Follow-up Ergebnisses / letzter Information zum Patienten
Abbildung Systeme / Akteure
| System | Akteur |
|---|---|
| PVS | Niedergelassene Haus- und Fachärzte |
| ADT\* / KAS | Fachabteilung: Chirurgie, Onkologie, … |
| OP-System | Chirurg |
| Pathologiesystem | Pathologe |
| Strahlentherapiesystem | Strahlentherapeut |
| Chemotherapieplanungssystem / Apothekensystem | Onkologe |
| Organkrebszentrum / Spezialanwendung Tumorkonferenzsystem | Interdisziplinär / Intersektoral, bei Organkrebszentren oft eine Fach¬abteilung federführend |
| einrichtungsbezogenes klin. Register | klin.Reg. |
| Klin. Register nach KFRG | KFRG |
| Epid. Register | epid.Reg. |
| QS / AQUA / DOC | QS |
| Weitere Systeme mit möglicherweise wenig betroffenen Fällen | |
| Laborsystem (Tumormarker und andere Spezialbefunde) |
(potenzielle fast alle)
Systeme von Studienzentren||Prüfärzte (aus fast allen Fächern) |
\*Admission-Discharge-Transfer
Meldungen für Fall 1
Für den ersten Fall werden folgende Meldungen übermittelt:
| Meldung | KFRG | Datum | Inhalt | Modell |
|---|---|---|---|---|
| Id1 | d | 16.1.09 | Pathobefund | Phänomen |
| Id2 | a | 15.1.09 | Koloskopie | Prozedur |
| ICD10: C18.6 V | Ergebnis | |||
| Polypektomie | Prozedur | |||
| 18.1.09 | ICD10: C18.6 G | Ergebnis | ||
| ICD-O: Datum, Diagnose noch offen | Erkrankung | |||
| Id3 | 25.1.09 | Röntgen Thorax | Prozedur | |
| CT Abdomen | Prozedur | |||
| TNM: cT2 N0 M0 | Phänomen | |||
| Id4 | 27.1.09 | Hemikolektomie offen | Prozedur | |
| OPS: 5-455.61 | Prozedur | |||
| 30.1.09 | TNM: pT2 pN1 G2 L0 V0 R0 (lokal radial operiert) | Phänomen | ||
| Id5 | ICD-O: M8140/3 Adenokarzinom ohne nähere Angaben | Diagnose | ||
| 31.1.09 | TNM: pT2 cT2 pN1 cN0 cM0 G2 C0 V0 R0 (lokal) | Phänomen |
Struktur der Meldung
In den jeweiligen Meldungen sind folgende Inhalte vorgesehen:
| Sektionen | ID | Inhakt | Beispiel |
|---|---|---|---|
| Erkrankung | E1 | ICD | |
| Diagnoseanlass | DA1 | ||
| Diagnose 1 | D1 | C18.6 G | |
| Tumorformel 1 | T1 | cT2 N0 M0 | |
| Maßnahme 1 | M1 | Diagnostik | |
| Maßnahme 2 | M2 | Dto. | |
| Maßnahme 3 | M3 | Dto. | |
| Maßnahme 4 | M4 | Dto. | |
| Maßnahme 5 | M5 | OP | |
| Tumorformel 2 | T2 | pT2 pN1 G2 L0 V0 R0 (lokal radial operiert) | |
| Diagnose 2 | D2 | ICD-O | M8140/3 |
| Tumorformel 3 | T3 | Zusammenfassende Beurteilung T1 + T2 | pT2 cT2 pN1 cN0 cM0 G2 C0 V0 R0 (lokal) |
Alle Sektionen verweisen auf die Erkrankung.
Vergütung
Die Höhe der Vergütung ist abhängig von dem Meldeanlass:
| Meldung | Inhalt | Vergütung |
|---|---|---|
| a | Meldung einer Diagnosestellung eines Tumors nach hinreichender Sicherung (§ 2 I 3 Buchst. a der Krebsregister - Meldevergütung - Vereinbarung vom 15.12.2014) | 18,00€ |
| b | MeldungvonVerlaufsdaten (§ 2 I 3 Buchst. b der Krebsregister-Meldevergütung-Vereinbarung vom 15.12.2014) | 8,00 € |
| c | Meldung von Therapie- und Abschlussdaten (§ 2 I 3 Buchst. c der Krebsregister-Meldevergütung-Vereinbarung vom 15.12.2014) | 5,00 € |
| d | Meldung eines histologischen oder labortechnischen oder zytologischen Befundes (§ 2 I 3 Buchst. d der Krebsregister-Meldevergütung-Vereinbarung vom 15.12.2014) | 4,00 € |
| e | Vergütungsabschlag für zahnärztliche Diagnosemeldung ohne Angabe des ICD-Codes (§ 2 I 4 < Protokollnotiz > der Krebsregister-Meldevergütung Vereinbarung vom 15.12.2014) | 3,00 € |
(Schiedsspruch vom 24.02.2015 gemäß § 65c VI 8 SGB V i.V.m. § 2 II der Krebsregister-Meldevergütung-Vereinbarung vom 15.12.2014.)
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Domänen-Analyse Modell
Vorgehensweise
Von der Vorgehensweise lässt sich das Projekt wie folgt zusammenfassen:
- Analyse der bestehenden Datensatzbeschreibungen
- Zusammenfassung in ein Modell auf Basis von UML: DAM
- Ergänzung des Basis-Modells um entitätsspezifische Details
- Festlegung des dynamischen Verhaltens
- Abbildung der einzelnen Klassen aus dem Modell auf Abschnitte innerhalb mit Angabe von Verknüpfungen
- Festlegung der Vokabularien und Value Sets
Erläuterung
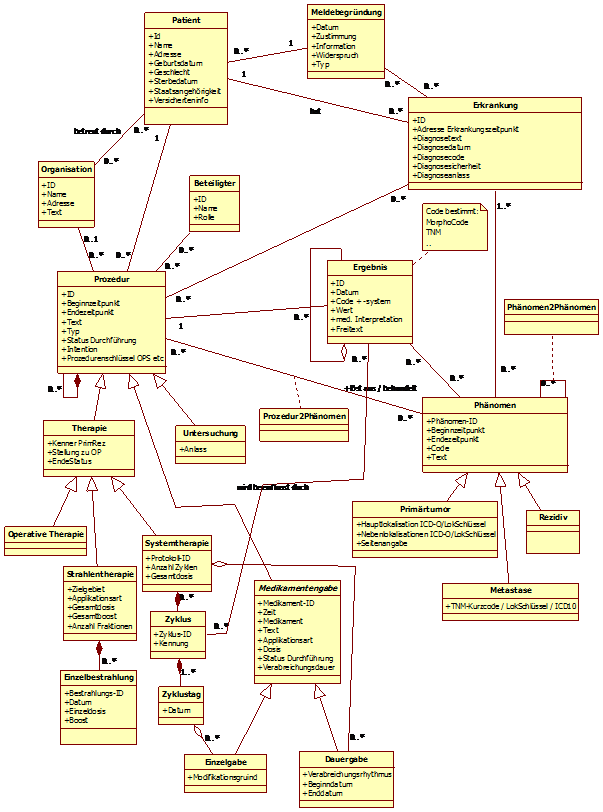
Nachfolgend ist das Domänen-Analyse-Modell (DAM) abgebildet:
Abbildung 2: DAM
Die einzelnen Bereiche daraus werden nachfolgend eingehender erläutert.
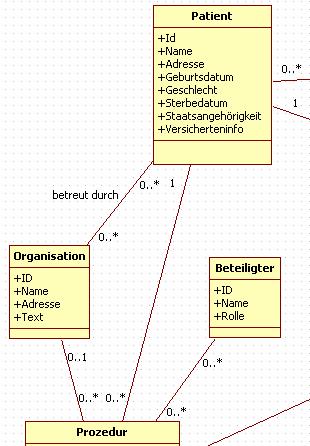
Patient
Die Klasse Patient enthält die aktuellen Angaben zum Patienten. Die Inzidenz¬adresse einer Erkrankung ist in der Klassen Erkrankung zu finden. Die Kostenträgerinformation wird z.B. durch einige klinische Register zur Abrechnung von Fallpauschalen benötigt.
Organisation
Organisation umfasst im weiteren Sinn jegliche Einheit, die in einem direkten Verhältnis zum Patienten steht oder verantwortlich für spezielle Maßnahmen ist. Im Konkreten sind hier beispielsweise Angaben zum Betreuungskontext eines Patienten möglich, also z.B. Krankenhausabteilungen oder Praxen.
Beteiligter
Beteiligte bezieht sich auf direkt an einer Prozedur Beteiligte. Diese müssen von der Organisation getrennt werden (viele Auswertungen verlangen z.B. direkt Operateure wie im Basisdatensatz).
Abbildung 3: DAM: Patient, Organisation, Beteiligte
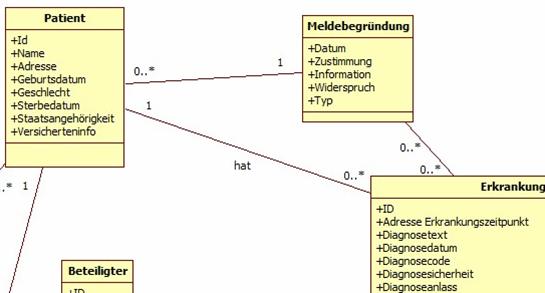
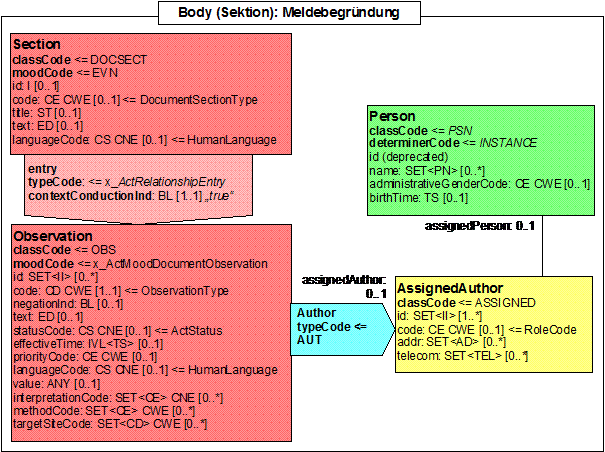
Meldebegründung
Abbildung 4: DAM: Meldebegründung
Dort werden alle Angaben abgelegt, die sich im Kontext von Übermittlung von Daten auf Informations- oder Einwilligungsstatus beziehen. Diese Angaben werden von epidemiologischen Krebsregistern je nach Bundesland vorgegeben. Klinische Krebsregister werden in der Regel eine Einwilligung benötigen. Es ist zu erwarten, dass hierdurch eine Reihe weiterer Zwecke (Forschung, Qualitätssicherung, ...) abzubilden sind, die sich evtl. auch nur auf spezifische Ereignisse oder Bereiche beziehen. Die Meldebegründung hat einen rechtlichen Hintergrund und darf nicht mit dem Anlass einer Meldung verwechselt werden (z.B. Erst-, Folge-, Korrekturmeldung etc.).
Erkrankungen
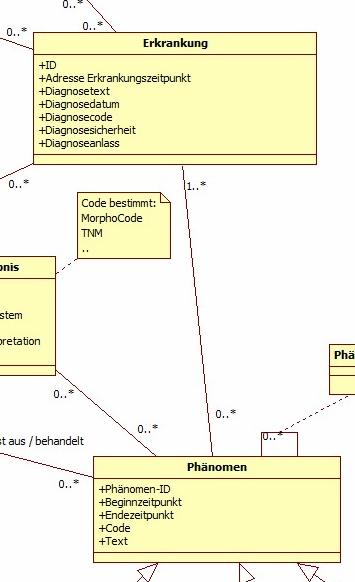
Abbildung 5: DAM: Erkrankung
Dies ist die Klasse für zentrale Daten für beliebige Erkrankungen. Von zentraler Bedeutung sind natürlich Tumorerkrankungen. Durch die Umsetzung des Modells muss sichergestellt werden, dass für jede Tumorerkrankung genau eine Instanz (=ein Eintrag) existiert. Davon unterschieden werden Phänomene, die je nach Art der Erkrankung unterschiedlich sind und die weitere Details enthalten. Für Tumorerkrankungen sind das beispielsweise Primärtumor und Metastasierungen. Nicht-Tumorerkrankungen sind nicht Bestandteil des Basisdatensatzes und in diesem Kontext nicht verpflichtend. Dennoch können sie beispielsweise Therapieentscheidungen beeinflussen und werden in einigen Registern mitgeführt. Bei diesen Erkrankungen sind ggf. einige Attribute nicht verpflichtend.
Phänomen
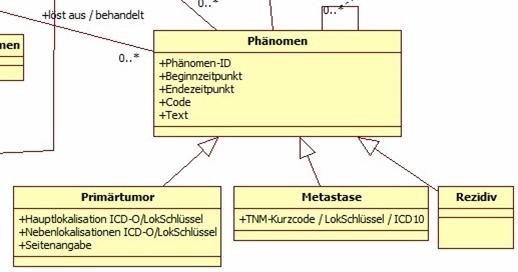
Abbildung 6: DAM: Phänomen
Phänomene sind direkte oder indirekte Erscheinungsformen der Tumorerkrankung. Direkte Phänomene (Primärtumor, auch in Sinne von Systemerkrankung, und Metastasen) sind prinzipiell nachweisbar über Tumorzellen, indirekte sind Erkrankungs- oder Therapiefolgen. Allen Phänomenen ist ein Beginn (Diagnosezeitpunkt) und eine dem Phänomentyp adäquate Codierung gemeinsam. Phänomene können enden und sofern es sich um ein direktes Phänomen handelt, ggf. danach rezidivieren. Darüber hinaus gibt es eine Rezidivierung der Erkrankung an sich, wenn nach kompletter Tumorfreiheit ein direktes Phänomen neu oder wieder auftritt (Primärtumorrezidiv, Rezidiv einer Metastase, Auftreten einer neuen Metastase). Eine Sonderform des Rezidivs ist ein Tumormarker-Rezidiv, das sich zumindest anfänglich jeglichen direkten Nachweises eines Phänomens entzieht. Unter pragmatischen Aspekten der Dokumentation werden unter Umständen prinzipiell auch einzeln nachweisbare Phänomene zusammengefasst:
- Mehrere Primärtumor in einem Organ (z.B. mehrere Basaliome, mehrere Darmtumore, mehrere Mammatumore in einer Brust, ...)
- Generalisierte Metastasierung statt Auflistung aller Metastasenlokalisationen
- Multiple Metastasen in einem Organ werden nicht einzeln notiert
Über direkte Phänomene wird letztendlich die Tumorerkrankung diagnostisch gesichert, weshalb sie über Ergebnisse in Verbindung mit (diagnostischen) Prozeduren steht Das Phänomen Primärtumor mit dem Tumorsitz existiert genau einmal pro Erkrankung. Dies gilt im übertragenen Sinne von Primärerkrankung auch für Systemerkrankungen. Metastasen können bereits bei Diagnose einer Tumorerkrankung vorliegen oder im Verlauf auftreten. In einem bestimmten Prozentsatz gehen sie der Entdeckung eines Primärtumors voraus, oder der Primärtumor wird nie entdeckt, weil er von selbst verschwunden ist oder eine intensive Diagnostik nicht indiziert ist. Außerdem kann es bei Vorliegen mehrerer Tumorerkrankungen unmöglich sein, die Metastase genau einer dieser Erkrankungen zuzuordnen. Daher können Metastasen (und somit Phänomene), mehreren Erkrankungen zugeordnet sein, im Extremfall allen (Tumor-)Erkrankungen. Indirekte Phänomene sind:
- Folgeerkrankungen der Tumorerkrankung an sich (Anämie, Kachexie, Schmerzen, bisher nicht Bestandteil der Dokumentation)
- Folgeerkrankungen und Folgezustände der Therapie, die zumindest teilweise zwangsläufig auftreten (enthalten in der Vorversion des Basisdatensatzes und ggf. noch gängige Praxis in Registern) und die von akuten oder chronischen Nebenwirkungen sowie OP-Komplikationen abzugrenzen sind
- Akute und chronische Nebenwirkungen von Strahlen oder Chemotherapie (internationale Standards wären CTC, RTOG und WHO), in Basisdatensatz enthalten und teilweise für Zertifizierungen wichtig
- OP-Komplikationen, in Basisdatensatz enthalten, teilweise organspezifische Besonderheiten für Zertifizierungen
Über Phanomen2Phänomen können folgende Assoziationen zwischen Phänomenen bestehen.
- ist Rezidiv von:
- Primärtumor: drückt aus, wenn ein Rezidiv an der Stelle des Primärtumors auftritt
- Metastase: wenn eine Metastase an einem Ort wieder auftritt, von dem sie vorher entfernt wurde (oder wurden wenn es sich um mehrere handelt, die nicht einzeln gewertet wurden)
- ist Metastase von:
- Primärtumor: (eigentlich ist das schon durch die Beziehung zur Erkrankung klar, bzw. nicht klar wenn nicht zuordenbar bei mehreren Erkrankungen)
Prozedur (mit Unterklassen Therapie und Untersuchung)
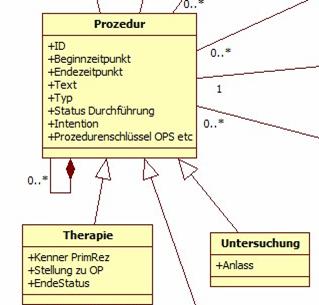
Abbildung 7: DAM: Prozedur
Therapien und/oder Untersuchungen lassen sich in der Realität manchmal nicht trennen. Bezeichnet jegliche Maßnahmen am Patienten. Prozeduren können diagnostisch (im Sinne von Untersuchungen) und / oder therapeutisch sein. Die unterschiedlichen Aspekte drücken sich durch die Unterklassen Therapie und Untersuchung aus. Außerdem können sich Prozeduren aus mehreren Teilen zusammensetzen. Auf sehr hoher Ebene korrelieren sie teilweise mit den Dokumenten des Basisdatensatzes, wobei die eigentlichen Inhalte weitgehend in anderen Klassen wie Ergebnis, Erkrankung und Phänomen stehen.
- Diagnosedaten: Der Prozess der Diagnosestellung an sich
- Verlaufsdaten: Verlaufsdaten sind aus Sicht des Basisdatensatzes Statuserhebungen zu bestimmten Anlässen wie Therapieabschlüssen, Nachsorgen, Untersuchungen wegen Beschwerden etc. Eine entsprechende Differenzierung findet sich in der Unterklasse Untersuchung.
- Operative Therapie, Strahlentherapie und systemische Therapie (oder Kombinationen aus diesen): Diese setzen sich je nach Therapietyp wiederum aus z.B. Einzelprozeduren (z.B. im Sinne von OPS-Ziffern), unterschiedlichen Zielgebieten oder Zyklen etc. zusammen. Die Modellierung erfolgt über entsprechende Unterklassen.
- Life-Status / Abschlussdaten
- Autopsiedaten sind inhaltlich weitgehend mit Verlaufsaussagen deckend.
Aus praktischer Sicht wird häufig eine Fragmentierung dieser Dokumente erfolgen, die sich aus der Zeitachse und ggf. unterschiedliche Akteure ergibt, siehe Präsentation „Dokumentationsfragmente des Basisdatensatzes". Es ist anzustreben, dass jeder Akteur aus Gründen der Datensparsamkeit lediglich seinen Anteil an der Gesamtdokumentation berichtet. Das Konzept „vorgesehene Maßnahme" des Basisdatensatzes wird über den Durch¬führungsstatus umgesetzt. Hier ist eine potentielle Erweiterung der vertieften Dokumentation für Zertifizierungen denkbar, bei der zum Beispiel Prozessqualitäten auch die Planung z.B. im Rahmen von Tumorkonferenzen berücksichtigen, siehe die letzten beiden Seiten „Vorschlag für ein erweitertes Modell der Therapiedokumentation" im Dokument „Basisdokumentation Handbuch V2a.doc" Therapien sind Aussagen zu Intention und gegenseitiger Stellung sowie der planmäßigen Durchführung eigen. Diese können auch durch die explizite Assoziation „Prozedur behandelt Phänomen" ausgedrückt werden. Da Therapien wiederum indirekte Phänomene auslösen können, kann die Assoziation „Prozedur2Phänomen" auch die Qualität löst aus besitzen.
Operative Therapie
Ist bereits weitgehend durch die allgemeinen Felder von Prozedur beschrieben. Eine extra Unterklasse ist dennoch sinnvoll, da z.B. der OP-Schlüssel in einigen Fällen nicht alle Informationsbedürfnisse im Rahmen von Qualitätssicherung abdeckt. Eine Operative Therapie kann sich aus mehreren Operationen (in der Regel OP-Tage) zusammensetzen. Auf der Ebene der Operationen sind die Beziehungen zu den Beteiligten (=Operateure), die OP-Ergebnisse (aus diagnostischer Sicht und aus Erfolgssicht in Klasse Ergebnis), sowie die ggf. ausgelösten OP-Folgen (insbesondere Komplikationen) durch Phänomene ausgedrückt.
Strahlentherapie
Eine Strahlentherapie ist definiert durch ein Zielgebiet, das über eine bestimmte Applikationsart mit einer gewissen Dosis und evtl. einer Boost-Dosis bestrahlt wird (wobei sich ein Boost wohl immer auf eine Teilregion bezieht – müsste die dann nicht explizit als Boost-Zielgebiet gefasst werden?). Eine Strahlenbehandlung kann sich dabei ggf. aus mehreren (auch gleichzeitigen) Therapien (Zielgebieten) zusammensetzen. Die Strahlentherapie kann sich aus mehreren Einzelbestrahlungen zusammensetzen und korreliert hier mit einer einzelnen Dosis. Die Einzelbestrahlung ist nicht Bestandteil des ADT-Datensatzes. Denkbare Anwendung wäre im Rahmen von Studien, in denen Kapazitäten für eine derart detaillierte Erfassung vorhanden sind oder die Annahme von Daten aus einem Bestrahlungssystem. (ist das so? Ich frage mich, ob die Information Boost dann dort drin steckt. Kennt sich da jemand aus? Andernfalls wäre es nicht doch besser, das erst mal wegzulassen?).
Systemtherapie
Ein Zyklus ist ein logischer, ggf. wiederholbarer Teil einer systemischen Behandlung. Die Zyklus-Kennung entspricht in diesem Fall der Zyklusnummer, die aber nicht notwendigerweise numerisch ist, da Therapien eben nicht notwendigerweise wiederholt werden oder ggf. unterzyklisch geplant werden. Letzteres ist insbesondere in einer Kommunikation mit Apothekensystemen zu bedenken und führt insbesondere auch zur Assoziation „wird beeinflusst durch" von Zyklus und Ergebnis (z.B. Körpergröße, Gewicht, Kreatinin, Leukozyten, …).
Die Einzelgabe ist dann die konkrete Klasse für die Medikamentengabe. Unterstellt man, dass hinter einem Protokoll ein Musterverabreichungsplan steht, gehen aus der Gesamtbetrachtung aller Einzelgaben Unterbrechungen und Dosisreduktionen oder –eskalationen hervor. Zusätzlich kann es erforderlich, sein, Modifikationen, d.h. zu begründen, wie es im Basisdatensatz gefordert wird. Die Dauergabe könnte zwar theoretisch ebenfalls über Einzeldosen abgebildet werden, ist aber, da eher durch den Patienten eingenommen, nicht ohne weiteres kontrollierbar, weshalb eine gesonderte Klasse gebildet wurde, die zum Beispiel häufig für Hormontherapien Anwendung finden dürfte. Das gröbere Modell des Basisdatensatzes wird in der auf dem Bogen „Systemische Therapie" dargestellten Form für nicht praktikabel gehalten, da eine Reihe aktueller Protokolle darüber nicht darstellbar ist. Inhaltlich entspricht es diesem jedoch. In der Praxis wir eine derart genaue Dokumentation jedoch nur bei Integration mit Planungssystemen für machbar gehalten.
Wünschenswert wäre ein zentrales „Register" für Definitionsdaten von Therapieprotokollen. Ohne ein solches Register wird es m.E. notwendig sein das Modell um die Definitionsdaten zu erweitern, d.h. unter Umständen die Therapieprotokolldefinition jeweils mit zu übertragen.
Ergebnis
Abbildung 8: DAM: Ergebnis
Die Klasse Ergebnis ist der Container sowohl für direkte Untersuchungsergebnisse als auch deren Zusammenfassung zu einer Beurteilung/Bewertung. Direkte Untersuchungsergebnisse und Beurteilungen/Bewertungen sind sauber auseinanderzuhalten. Während Untersuchungsergebnisse möglicherweise über Schnittstellen aus anderen Systemen über¬tragen werden können und die zwangsläufig begrenzte Sicht eines Einzelbefunders repräsentieren, müssen mehrere Untersuchungsergebnisse häufig zusammenfassend bewertet werden, um daraus beispielsweise eine Therapieindikation abzuleiten. So sieht der Pathologe nur das, was er an Präparat und Begleitinformation bekommt, der behandelnde Arzt kennt aber den ganzen Patienten und muss im Falle von pT und pN den klinisch erhobene M-Status ergänzen. Eine formal vollständige Histologie aus einer Biopsie hat eine andere Aussagekraft als die aus einem Resektionspräparat.
Untersuchungsergebnisse können sich aus Einzelteilen zusammensetzen. So enthält eine TNM-Formel die einzelnen Kategorien zu T, N und M und deren Präfixe etc.. Durch konkrete Instanziierungsregeln ist zu gewährleisten, dass z.B. innerhalb einer TNM-Formel jede Kategorie (T, N, M, ...) bzw. jeder Zusatz (Datum, y-Symbol, Certainty-Faktor, ...) höchstens einmal vorkommt. Vergleichbares gilt für andere strukturierte Angaben wie histologische Befunde oder ein Ann Arbor Klassifikation. Beurteilungen und Bewertungen reflektieren eher das, was Register benötigen, während direkte Untersuchungsergebnisse für weitergehende Analysen im Rahmen von QM oder Studien benötigt werden und möglicherweise näher an Quelldaten in anderen Systemen sind.
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Dynamisches Modell
Grundsatzfrage
Die Übersetzung des DAMs in ein dynamisches Modell bietet grundsätzlich zwei Möglichkeiten:
- Erarbeitung eines Modells zur Nachrichtenübertragung (D-MIM) mit Ableitung von entsprechenden Transaktionen
- Übertragung in ein äquivalentes Dokumentenformat (CDA)
Die bisherigen Meldungen beruhen auf dem Austausch von Dokumenten (=Meldungen). Daher bietet es sich an, CDA (Clinical Document Architcture) als Grundlage zu nehmen und dafür entsprechende Abschnitte (Templates) zu definieren.
Interaktionsdiagramm
Abbildung 9: Interaktionsdiagramm
Die Daten, die an die epidemiologischen Krebsregister geschickt werden, bedürfen weder einer Anonymisierung noch einer Pseudonymisierung. Sollte aber eine Pseudonymisierung erforderlich sein, so wird eine Vertrauensstelle benötigt, die die Daten entsprechend überarbeitet. Im Prinzip spielt diese Vertrauensstelle dann beide Rollen gleichzeitig, wobei aus dem erhaltenen Dokument eine überarbeitete Fassung erstellt wird (TRANSFORM), die dann an den entsprechenden Empfänger weitergereicht wird.
Abbildung 10: Interaktionsdiagramm mit Pseudonymisierung
Vertrauensstelle
Die Gesetze zum Datenschutz in den jeweiligen Bundesländern regeln individuell, wann eine Anonymisierung bzw. Pseudonymisierung der Daten vorzunehmen ist. In einem Bundesland dürfen die Daten nur vom erhebenden Arztbearbeitet werden, in einem anderen nur im Krankenhaus, wieder in einem anderen Bundesland nur bei Einhaltung der ärztlichen Schweigepflicht. usw.
Die Vertrauensstelle hat nun die Aufgabe, die Anonymisierung sowie Pseudonymisierung der Daten/Dokumente sicherzustellen. Alle die Person direkt identifizierenden Daten sind aus dem Dokument zu entfernen und ggf. durch geeignete Platzhalter zu ersetzen.
IHE ITI überlegt derzeit, ein Profil zur Pseudonymisierung zu erstellen.
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Statisches Modell
Einleitung
Dieser Leitfaden setzt auf dem VHitG-Arztbrief auf. Daher werden auch die dortigen Spezifikationen übernommen. Die nachfolgende Tabelle gibt Aufschluss über die in einer Meldung enthaltenen Daten. Die Umsetzung in Form von Abschnitten erfolgt anhand des Analysemodells. Die Spalte „Klasse" referenziert auf die Information in dem Modell. Die letzte Spalte reflektiert in dem Modell dann die Beziehungen der Klassen untereinander.
Dabei kann eine Klasse sowohl aus inhaltlichen als auch nur aus Referenzzwecken übermittelt werden. Wird zum Beispiel eine Erkrankung erstmalig gemeldet, so wird diese Klasse als Inhalt übermittelt. In folgenden Meldungen wird die Erkrankung nur noch als Referenz zur korrekten Herstellung von Bezügen übermittelt. Ein weiterer zu bedenkender Fall wäre eine Korrektur . Über einen noch zu definierenden Mechanismus (Zeitstempel? Extra Attribut?) sollte das empfangende System in der Lage sein, diese Anlässe auseinanderzuhalten.
Abbildung 11: vereinfachte Gesamtübersicht
Grundsätzliche Anforderungen an die Dokumentstruktur
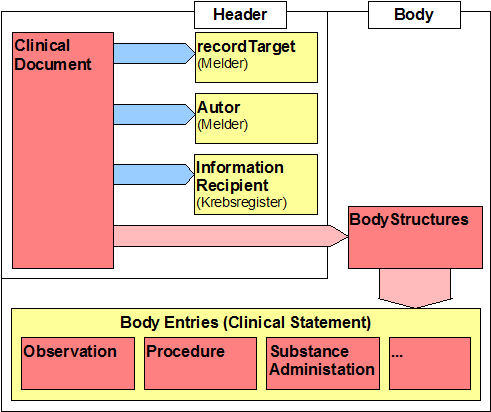
Dokumente setzen sich grundsätzlich aus folgenden Komponenten zusammen:
- dem eigentlichen Inhalt
- der Kontextinformation
Die Kontextinformation soll der korrekten Handhabung des Inhalts dienen. Korrekte Handhabung beinhaltet
- Die Zuordnung zum Patienten, zur Erkrankung und ggf. der gegenwärtigen therapeutischen Situation
- Die Übermittlung von Meldebegründungen, die Aussagen zur weiteren Nutzbarkeit von Daten erlauben
Im Grundsatz ist davon auszugehen, dass zum Patient die Personen identifizierenden Daten übermittelt werden. Dies ist insbesondere anzunehmen, wenn der Datenfluss der Betreuung folgt und natürlich wenn es die Meldebegründungen entsprechend vorsehen. Die Personenidentifikation kann um Zusatzidentifikatoren zum Aufbau und zur Nutzung eines Master-Patient-Index (MPI) erweitert werden, um den Abgleich (Record Linkage) schneller und sicherer zu machen. Bei bestimmten Empfängern ist eine Pseudo¬nymisierung, unter Umständen im Sinne einer Kontrollnummernbildung, erforderlich. Diese kann sowohl bereits bei Absendung der Daten erfolgen als auch erst in einer Vertrauensstelle. Dabei kann es erforderlich werden, dass Teile der Daten kryptographisch geschützt werden (Beispiel Krebsregister Baden-Württemberg).
Beispiel für groben Aufbau
<?xml version="1.0" encoding="UTF-8" ?>
<ClinicalDocument>
<!-- CDA Header -->
... siehe Beschreibung CDA R2 Header
<!-- CDA Body -->
<component>
<structuredBody>
... siehe Beschreibung CDA R2 Body
</structuredBody>
</component>
</ClinicalDocument>
Nachfolgend ein Beispiel, in dem der Header ausführlicher dargestellt ist:
<?xml version="1.0" encoding"UTF-8" ?>
<?xml-stylesheet type="text/xsl" href="?????.xsl"?>
<ClinicalDocument
xmlns:xsi="http://www.w3.org/2001/XMLSchema-instance"
xmlns="urn:hl7-org:v3">
<typeId root="2.16.840.1.113883.1.3" extension="POCD_HD000040"/>
<templateId root="1.2.276.0.76.3.5.7">
<id extension="60467049" root="1.2.276.0.58"/>
<code code="???????" codeSystem="2.16.840.1.113883.6.1"
displayName="???????"/>
<title>Meldung an klinisches Krebsregister</title>
<effectiveTime value="20060924"/>
<confidentialityCode code="N" codeSystem="2.16.840.1.113883.5.25"/>
<languageCode code="de"/>
<setId extension="D1" root="2.16.840.1.113883.3.933"/>
<versionNumber value="1"/>
...
<!-- CDA -->
<component>
<structuredBody>
<!-- Meldebegründung -->
<component>
<section>
<templateId root="1.2.276.0.76.3.1.131.1.10.????"/>
...
<!-- Referenz auf Participant -->
</section>
</component>
...
<!-- Erkrankungsdaten -->
<component>
<section>
<templateId root="1.2.276.0.76.3.1.131.1.10.?????"/>
...
<!-- Referenz auf Phänomendaten -->
</section>
</component>
</structuredBody>
</component>
</ClinicalDocument>
Identifikation von Informationseinheiten
Mechanismen
Ein Informationsobjekt kann grundsätzlich über zwei Mechanismen identifiziert werden
- über ein „neutrale" Identifikationsmerkmal (automatisch vergebene, eindeutige ID)
- über unveränderliche Eigenschaften des Informationsobjektes, die eine eindeutige Identifikation ermöglichen
Während die erste Möglichkeit keine Aussage über das Objekt selbst erlaubt, ist die zweite Möglichkeit unter Umständen nicht gegeben. So können bspw. weder bei Patienten die Namen noch bei Tumoren die Diagnose zur Identifikation herangezogen werden.
Natürlich reicht eine ID alleine nicht aus, um ein Objekt (z.B. eine Erkrankung) zu beschreiben, ermöglicht aber die Übermittlung von Beziehungen und damit die Zuordnung von Korrektur¬informationen.
Zeitstempel der Information
Zeitstempel informieren das Zielsystem über den Zeitpunkt der Entstehung oder Veränderung der Daten. Hierdurch kann das Zielsystem Entscheidungen über notwendige Bearbeitungsschritte treffen (Beispiel siehe unten). Die in den einzelnen Klassen enthaltenen Zeitangaben beziehen sich aber auf die Information selbst und nicht den Zeitpunkt der Veränderung. Eine solche Information müsste im Attribut participant@Time zu der Rolle Author ausgedrückt werden. Diese optionale Information wird nicht immer verfügbar sein. Daher sind grundsätzlich alle Informationen auf Aktualität zu überprüfen und dann ggf. zu korrigieren.
Beispiel Organzentrumssystem (OZ) - Klinisches Krebsregister (KKR)
| Übermittlung OZ => KKR | Aktion im KKR |
|---|---|
| 18.3.2010: (Patient) Erkrankung-ID 3456 / Diagnose-ID 456, Diagnosedatum 1.3.2010 / Diagnosecode C50.9 / Seite rechts | Kennt Erkrankung-ID 3456 noch nicht, legt eigene Erkrankung an kennt Diagnose noch nicht, wird ebenfalls angelegt merkt sich Referenz Erkrankung-ID |
| 25.3.2010: (Patient) Erkrankung-ID 3456 / Diagnose-ID 456, Diagnosedatum 1.3.2010 / Diagnosecode C50.4 / Seite rechts | Kennt Erkrankung-ID 3456 bereits, legt keine neue Erkrankung an, kennt Diagnose bereits, korrigiert Diagnosecode |
| 1.4.2010: (Patient) Erkrankung-ID 3456 / Diagnose-Id 456, Diagnosedatum 1.3.2010 / Diagnosecode C50.4 / Seite rechts Operation brusterhaltend am 27.3.2010, Operation/Prozedur-ID 3478237 | Kennt Erkrankung-ID 3456 bereits, macht keine Korrektur der Erkrankungsdaten verarbeitet Information zu Operation und ordnet sie der korrekten Erkrankung zu |
| 8.4.2010: (Patient) Erkrankung-ID 3456 / Diagnose-ID 456, Diagnosedatum 1.3.2010 / Diagnosecode C50.4 / Seite rechts Operation Brusterhaltend am 27.3.2010, Operation/Prozedur-ID 3478237 Phänomen Wundheilungsstörung 2.4.2010, Phänomen-ID 574547 | Kennt Erkrankung-ID 3456 und Prozedur-ID 3478237 bereits, macht keine Korrektur
verarbeitet Information zur Wund¬heilungs¬störung und ordnet sie der korrekten Operation zu, speichert sich Phänomen-ID |
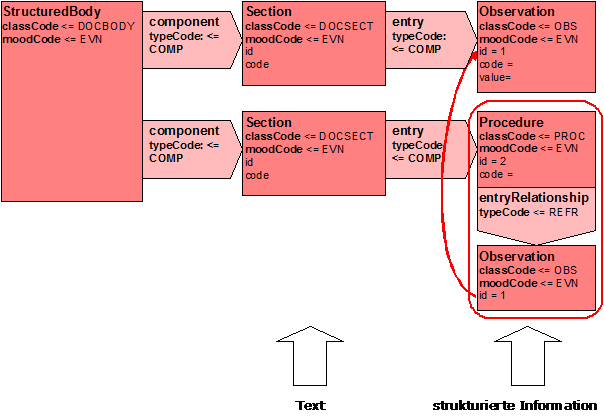
Referenzen auf andere Informationseinheiten
Die Meldung verwendet interne Referenzen gemäß des Analysemodells. Die Funktionsweise soll hier kurz erläutert werden.
Abbildung 12: Handhabung von Referenzen auf Aktivitäten
Über eine Entry-Relationship wird eine Beziehung zu einer anderen Instanz ausgedrückt. Im obigen Beispiel verweist ein Detail einer Maßnahme (Beobachtung) auf eine andere Beobachtung. Welche das konkret ist, kann aus der ID entnommen werden. Im obigen Beispiel verweist die Beobachtung mit der ID=1 (unten) auf die Beobachtung mit den Detailinformationen (oben).
An dieser Stelle sei darauf hingewiesen, dass eine ID sich normalerweise aus zwei Teilen zusammensetzt. Das ist die eigentliche ID, die dann auch alphanumerisch sein kann, sowie eine root-Angabe, die dann auf das ausgebende System sowie die Art der ID verweist. Letzteres wird durch eine OID realisiert. Näheres dazu regelt die FAQ \[DIMDI, FAQ OID\].
Je nach Spezifikation können für Instanzen auch mehrere IDs vergeben werden.
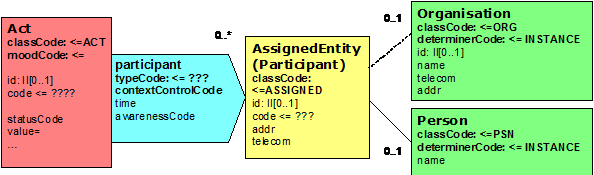
Referenzen auf Beteiligte
Die Handhabung der Referenzen auf die Beteiligten erfolgt gemäß nachfolgendem Schema:
Abbildung 13: Handhabung von Referenzen auf Personen
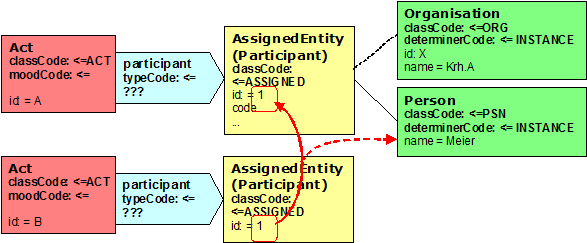
Im Bereich des Clinical Statement Patterns besteht die Möglichkeit, Informationen über Personen einzubinden. Grundsätzlich können hier von einer Aktivität mehrere Verweise (Participations) auf Rollen eingebunden werden. Die Rollen können wiederum weitere Details über Verweise auf Personen bzw. Organisationen realisieren. Hierbei müssen aber keine weiteren Details übermittelt werden. Dieser Mechanismus erlaubt somit, beim ersten Vorkommen alle Details zu den Entities zu übermitteln und bei allen anderen bei den Rollen aufzuhören. Der contextControlCode bestimmt, ob diese Information dem übergeordneten Bereich (Header oder einer anderen hierarchisch übergeordneten Struktur) entspricht. Berichtet z.B. jemand über die Maßnahme eines Dritten, so kann hier dieser Dritte berichtet werden. Grundlage ist hierbei die Nutzung entsprechender Identifikatoren. Nachfolgende Abbildung verdeutlicht dies:
Abbildung 14: Beispiel für die Nutzung von Identifikatoren zwecks Referenzierung
Durch die Wiederholung der ID (hier: „1") wird deutlich, dass bei der zweiten Aktivität (id=B) dieselbe Person („Meier") eingebunden ist wie in der ersten Aktivität (id=A). Hierbei spielt es keine Rolle, welche Beziehung die beiden Aktivitäten zueinander haben. @typeCode Beteiligung CD CWE \[0..1\] Der typeCode der Participation bestimmt hierbei, um welche Art der Beteiligung es sich handelt.
| Lvl | Code | Bedeutung | Erläuterung |
|---|---|---|---|
| 1 | PRF | performer | Ausführender |
| 2 | PPRF | primary performer | 1. Ausführender |
| 2 | SPRF | secondary performer | 2. Ausführender |
| VRF | verifier | Verifizierender | |
| ENT | data entry person | Datentypist | |
| CON | consultant | Berater | |
| DIS | discharger | Entlassender | |
| REF | referrer | Überweiser | |
| ADM | admitter | Aufnehmender | |
| ATT | attender | Beisitzer | |
| AUTHEN | authenticator | ||
| LA | legal authenticator | Unterzeichnender | |
| AUT | author | Autor | |
| RCT | record target | Akte |
Tabelle 3: Vocabulary Domain für die Beteiligung
@contextControlCode Kontext übernehmen BL Dieses Attribut bestimmt, ob der übergeordnete Kontext übernommen wird oder nicht.
Handhabung von Negationen
HL7 V3 stellt hierzu Mechanismen (Negation-Indikator und Null-Flavor) zur Verfügung, die hier jetzt nicht in aller Tiefe erklärt werden können. Deshalb sei hier auf die entsprechenden Materialien verwiesen.
??????????
Handhabung von Korrekturen und Folgemeldungen
Meldeanlässe und Inhalte
Grundsätze:
- Eine Meldung sollte dann ausgelöst werden, wenn eine Betreuungsepisode beendet und alle zu verantwortenden Informationen verfügbar sind.
- Grundsätzlich werden nur die Inhalte übermittelt, die mit eigenen diagnostischen oder therapeutischen Maßnahmen zusammenhängen. Davon sollte nur abgewichen werden, wenn andernfalls Lücken in der Erfassung auftreten würden.
Beispiel: Grundsätzlich ist eine chirurgische Abteilung verantwortlich für die Darstellung der operativen Therapie, des Therapieerfolgs und der Komplikationen. Informationen über eine außerhäusige Diagnosestellung müssten nur insofern übermittelt werden, dass das empfangende System in der Lage ist, die Therapie korrekt einem Tumor zuzuordnen. Wenn bekannt ist, dass dies aus irgendeinem Grund regelhaft nicht funktioniert oder vereinbart wurde, dass diese Informationen im Rahmen der operativen Therapie übermittelt werden sollen, hat die chirurgische Abteilung diese Eingabe zu verantworten.
Nachfolgend eine Liste der Meldeanlässe:
| Lvl | Code | Anlass | Details |
|---|---|---|---|
| 1 | Diagnose |
| |
| 1 | Therapie | ||
| 2 | Operative Therapie |
| |
| 2 | Strahlentherapie |
| |
| 2 | Systemische Therapie |
| |
| 1 | Verlauf |
| |
| 2 | Life-Status (Meldeamt) |
| |
| 1 | Sterbemeldung (Gesundheitsamt, Epid. Register) |
|
Die Meldeanlässe und Inhalte können je nach Umfang und Länge des überschauten Zeitraum und ggf. dem vorgesehenen Empfänger kombiniert werden. Dies betrifft insbesondere auch den Abgleich von Tumordokumentationssystemen unterschiedlicher Stufen (z.B. Spezialsystemen in Organzentren und regionalen Krebsregistern)
graphische Übersicht
Abbildung: Abschnittsübersicht (TODO: Diagramm muss aktualisiert werden!)
<Clinicaldocument>
...
<component>
<structuredBody>
<!-- Erkrankungsdaten -->
<component>
<section>
...
</section>
</component>
<!-- Meldebegründung -->
<component>
<section>
...
</section>
</component>
...
<!-- weitere Abschnitte -->
...
</structuredBody>
</component>
</Clinicaldocument>
offene Punkte
|
|
TODO: Phänomen und Erkrankungsdaten, Spezifikation Diagnoseleitfaden - ist noch nicht ausgearbeitet Dieser Abschnitt muss weiter nach hinten. |
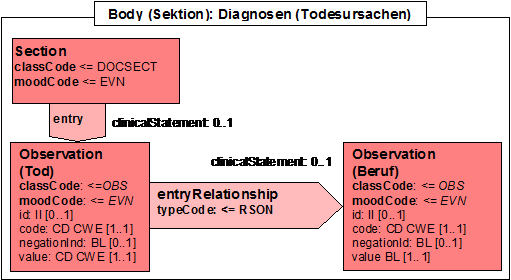
- Phänomen => über entryRelationship/observation
- Erkankungsdaten => über entryRelationship/observation
- Participant => über participation
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
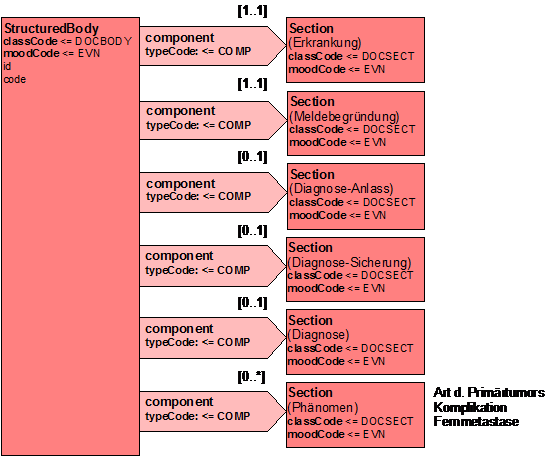
Dokumentenstruktur Krebsregistermeldung
Dieser Abschnitt listet die verschiedenen Meldungen auf, die an ein Krebsregister vorgesehen sind.
Dokumenttypen
Die Template-ID ist ein technischer Identifikator für die Inhalte in diesem Dokument, während der Dokumententyp ein Code aus einem Vokabular darstellt. Zwischen beiden existiert folgende Korrelation:
| Dokumententyp | Beschreibung | Dok.-Template-ID |
|---|---|---|
| Diagnose-Meldung | Meldung bei Feststellung eines Tumors oder bei Abschluss der primären Tumordiagnostik | 1.2.276.0.76.3.1.131.1.10.1.1 |
| Therapie-Meldung | Meldung nach Beginn oder Beendigung der Therapie | 1.2.276.0.76.3.1.131.1.10.1.2 |
| Verlaufs-Meldung | Meldung zum Verlauf/Nachbeobachtung, z.B. Nachsorge | 1.2.276.0.76.3.1.131.1.10.1.3 |
| Abschluss-Meldung | Meldung zum Abschluss der Erkrankung (z.B. Tod des Patienten oder Lost to Follow Up) | 1.2.276.0.76.3.1.131.1.10.1.4 |
Tabelle: Dokumententyp
CDA-Header
Im CDA-Header wird auf diverse Details verwiesen, deren Verwendung hier kurz aufgeführt wird:
| Element (Sequenz) | Datentyp | Bedeutung | Kard. |
|---|---|---|---|
| ClinicalDocument Klasse | |||
| realmCode | CS | eingesetzter Bereich | 1..1 |
| typeID | II | - konstant - | 1..1 |
| templateID | II | Template ID für das ganze Dokument | 1..1 |
| id | II | Dokumenten-ID | 1..1 |
| code | CE | Dokumententyp | 1..1 |
| title | ST | Titel des Dokuments | 0..1 |
| effectiveTime | TS | Erstellungsdatum des Dokuments | 1..1 |
| confidentialityCode | CE | Vertraulichkeitsgrad | 1..1 |
| languageCode | CS | Sprache des Dokuments | 1..1 |
| setID | II | Set-Kennung | 0..1 |
| versionNumber | INT | Versionsnummer | 0..1 |
| copyTime | TS | - nicht verwendet - | 0..0 |
| Participations | |||
| recordTarget | Patient | 1..1 | |
| author | Autor (hier: nur das meldende Softwaresystem) | 1..1 | |
| dataEnterer | Datentypist: med. Dokumentar(in) | 0..1 | |
| informant | Informationsquelle, Melder im Sinne der Krebsregister-Terminologie (Arzt und/oder Organisation) | 1..* | |
| custodian | Absender (Organisation). Kann identisch mit dem Melder sein oder z.B. ein regionales Tumorregister, das Daten an das Landeskrebsregister weiterleitet. | 1..1 | |
| informationRecipient | Empfänger: klinisches oder epidemiologisches Krebsregister | 1..1 | |
| legalAuthenticator | - nicht verwendet - | 0..0 | |
| authenticator | - nicht verwendet - | 0..0 | |
| participant | diverse Teilnehmer, d.h. Person bzw. Organisation, z.B. Krankenversicherung |
0..* | |
| Act Relationship | |||
| inFullfillmentOf | 0..0 | ||
| documentationOf | 0..0 | ||
| relatedDocuments | Verweis auf frühere Versionen | 0..1 | |
| authorization | 0..0 | ||
| componentOf | 0..0 | ||
| component | 0..0 |
Tabelle: Dokumentenstruktur (-bestandteile) für den Header
CDA-Body
An dieser Stelle sei auf den VHitG-Arztbrief verwiesen.
Die nachfolgende Tabelle regelt, welche Abschnitte in welchen Dokumenttypen enthalten sind, jeweils mit Optionalität und Kardinalität:
| Dokumenttyp: | Diagnose | Therapie | Verlauf | Abschluss | |||||
|---|---|---|---|---|---|---|---|---|---|
| Sektion | Opt. | Kard. | Opt. | Kard. | Opt. | Kard. | Opt. | Kard. | Template ID |
| ergänzende Patientendaten | O | 0..1 | O | 0..1 | O | 0..1 | O | 0..1 | 1.2.276.0.76.3.1.131.1.10.2.11 |
| Meldebegründungsdaten | R | 1..1 | R | 1..1 | R | 1..1 | R | 1..1 | 1.2.276.0.76.3.1.131.1.10.2.1 |
| Erkrankungsdaten | R | 1..1 | R | 1..1 | R | 1..1 | R | 1..1 | 1.2.276.0.76.3.1.131.1.10.2.2 |
| Diagnostik | R | 1..1 | 1.2.276.0.76.3.1.131.1.10.2.3 | ||||||
| Phänomendaten: Primär | R | 1..1 | 1.2.276.0.76.3.1.131.1.10.2.6 | ||||||
| Phänomendaten: Metastasen | O | 0..* | O | 0..* | 1.2.276.0.76.3.1.131.1.10.2.6 | ||||
| Operation | R | 0..* | 1.2.276.0.76.3.1.131.1.10.2.7 | ||||||
| Strahlentherapie | R | 0..* | 1.2.276.0.76.3.1.131.1.10.2.7 | ||||||
| systemische Therapie | R | 0..* | 1.2.276.0.76.3.1.131.1.10.2.7 | ||||||
| Medikation | O | 0..* | vgl. Arztbrief 2012 TODO | ||||||
| Status (Nachsorge und andere Follow-Up) | R | 1..1 | R | 1..1 | 1.2.276.0.76.3.1.131.1.10.2.8 | ||||
| Studiendaten | O | 0..* | O | 0..* | O | 0..* | 1.2.276.0.76.3.1.131.1.10.2.10 | ||
| Abschlussdaten | R | 1..1 | 1.2.276.0.76.3.1.131.1.10.2.9 | ||||||
| Therapieplanung | O | 0..1 | O | 0..1 | TODO | ||||
Tabelle 4: Dokumenttypen mit Zuordnung der Sektionen
|
|
Bei der Therapie muss einer der folgende Abschnitte vorhanden sein:
|
Abschnitt entspricht Component,
Klasse entspricht Section.
TODO: Dieser Abschnitt stellt noch nicht den aktuellen Stand dar!
| Abschnitt (Section) | Kard. | Klasse | Referenz auf Abschnitt (Header/Entry) |
|---|---|---|---|
| Meldebegründungdaten | 0..1 | ||
| Meldebegründung | Participant | ||
| Diagnostik | 0..* | ||
| Untersuchung | |||
| Ergebnis | |||
| Phänomen | |||
| Erkrankungsdaten | |||
| Beteiligter | Participant | ||
| Erkrankungsdaten | 0..* | ||
| Meldebegründungdaten | |||
| Erkrankung | |||
| Phänomendaten | |||
| Phänomendaten | 0..* | ||
| Phänomen | |||
| Operation | 0..* | ||
| Beteiligter | Participant | ||
| Operative Therapie | |||
| Erkrankungsdaten | |||
| Phänomendaten | |||
| Bestrahlung | 0..* | ||
| Beteiligter | Participant | ||
| Strahlentherapie | |||
| Erkrankungsdaten | |||
| Phänomendaten | |||
| Einzelbestrahlung | |||
| Medikamentendaten | 0..* | ||
| Einzelgabe | |||
| Dauergabe | |||
| Systemisch | 0..* | ||
| Beteiligter | Participant | ||
| Systemtherapie | |||
| Erkrankungsdaten | |||
| Phänomendaten | |||
| Zyklus | |||
| Zyklustag | |||
| Medikamentendaten | |||
| Status (Nachsorge und anderes Follow-up) | 0..* | ||
| Prozedur | |||
| Ergebnis | |||
| Erkrankungsdaten | |||
| Phänomendaten | |||
| Studiendaten | 0..* | ||
| Studie | |||
| Erkrankungsdaten | |||
| Abschlussdaten | 0..* | ||
| Prozedur | |||
| Ergebnis | |||
| Erkrankungsdaten | |||
| Planung | 0..* | ||
| Prozedur | |||
| Ergebnis | |||
| Erkrankungsdaten | |||
| Phänomendaten | |||
| Operation | |||
| Bestrahlung | |||
| Systemisch | |||
| Status (Nachsorge und anderes Follow-up) | |||
| Studiendaten | |||
| Diagnostik |
Tabelle: Dokument-Inhalte
Beteiligte (Header)
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
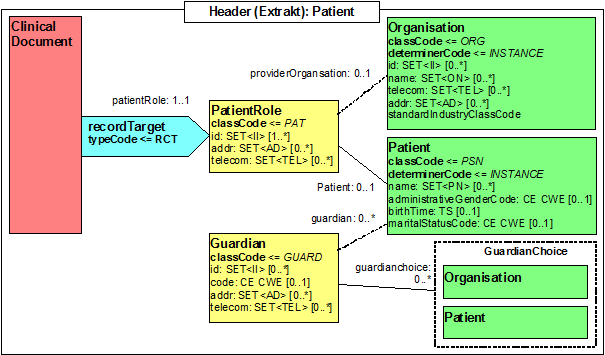
Participant: Patient (recordTarget)
| Template ID | <OID für das Template> | |
| General Description | Dieses Template spezifiziert, wie die Patientendaten in einer Krebsregistermeldung angegeben werden. | |
| Opt. | Description | |
| required | ||
|
|
Abgleich mit [Patient aus dem Arztbrief]. |
Abbildung: Identifikation des Patienten
| Lvl | RIM | Name | Desc | DT | Kard | Conf | Beschreibung
|
|---|---|---|---|---|---|---|---|
| 0 | act | ClinicalDocument | Dokument | 1..1 | M | ||
| 1 | part | recordTarget | Patient | 1..1 | M | ||
| 2 | part | @typeCode | "RCT" | CS CNE | 1..1 | M | fix |
| 2 | role | patientRole | Patient | 1..1 | M | ||
| 3 | role | @classCode | "PAT" | CS CNE | 1..1 | M | fix |
| 3 | role | id | Patient-ID | II.DE | 1..* | M | Identifiziert eindeutig den Patienten (OID des sendenden System, um Patienten eindeutig zu identifizieren) |
| 3 | role | addr | Adresse | AD.DE | 0..* | Es soll immer eine Adresse übertragen werden. Bei anonymisierten bzw. pseudonymisierten Patienten kann dies auf eine Postleitzahl oder Bundesland reduziert werden. | |
| 3 | role | telecom | Kontaktinformationen | TEL.DE | 0..0 | nicht verwendet | |
| 4 | ent | patient | Patient (Personendaten) | 0..1 | optional | ||
| 5 | ent | @classCode | "PSN" | CS CNE | 1..1 | M | fix |
| 5 | ent | @determinerCode | "INSTANCE" | CS CNE | 1..1 | M | fix |
| 5 | ent | name | Name des Patienten | PN.DE.PSEUDO | 0..* | required | Folgende Pseudonyme werden vorgesehen:
Bestimmte Attribute wie Namen oder Geburtsdatum sind dann optional, die dann in ganz definierten Kommunikationskontexten durch Kontrollnummern ersetzt werden. Die Identifikatoren unter 1. wären in jedem Fall sinnvoll für das automatisierte Record Linkage im Zielsystem, wenn es hier nicht geht, dann woanders |
| 5 | ent | administrativeGenderCode | Geschlecht | CE CWE | 0..1 | optional | Codesystem 2.16.840.1.113883.5.1 |
| 6 | ent | @code | Code für das Geschlecht | 1..1 | optional | ||
| 6 | ent | @codeSystem | "2.16.840.1.113883.5.1" | 1..1 | optional | fix | |
| 5 | ent | birthTime | Geburtsdatum | TS | 0..1 | optional | |
| 6 | ent | @value | Zeitpunkt der Geburt | 1..1 | optional | tagesgenau | |
| 4 | ent | providerOrganisation | Krankenhaus | 0..1 | nicht verwendet |
| Code | Bedeutung |
|---|---|
| F | weiblich |
| M | männlich |
| UN | undifferenziert, z.B. Hermaphrodit |
Tabelle: Geschlecht (Codesystem OID: 2.16.840.1.113883.5.1)
<recordTarget typeCode="RCT">
<patientRole classCode="PAT">
<id extension="12345678" root="1.2.276.0.76.3.1.131.1.4.3.9999.9999.999902"/>
<addr>
<streetName>Musterweg</streetName>
<houseNumber>10a</houseNumber>
<country>DE</country>
<postalCode>12345</postalCode>
<city>Musterstadt</city>
</addr>
<patient classCode="PSN" determinerCode="INSTANCE">
<name>
<prefix>Frau</prefix>
<prefix qualifier="AC">Dr. h.c.</prefix>
<given>Maria</given>
<family>Mustermann</family>
<family qualifier="BR">Musterfrau</family>
<family qualifier="AD">Musterfamilie</family>
</name>
<administrativeGenderCode code="F" codeSystem="2.16.840.1.113883.5.1"/>
<birthTime value="19500619"/>
</patient>
</patientRole>
</recordTarget>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
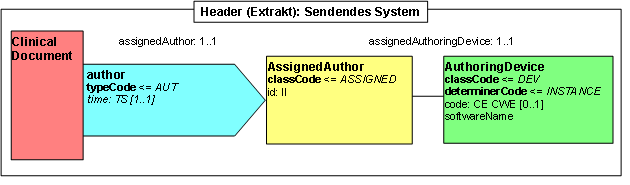
Participant: Sendendes System (author)
Abbildung: Sendendes System
| Lvl | RIM | Name | Desc | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|---|
| 0 | act | ClinicalDocument | |||||
| 1 | part | author | Autor (hier: nur das sendende System) | 1..1 | M | ||
| 2 | part | @typeCode | "AUT" | CS CNE | 1..1 | M | fix |
| 2 | part | time | Zeitpunkt der Erstellung des Dokuments | TS | 1..1 | M | |
| 3 | part | @value | der eigentliche Zeitpunkt | 1..1 | M | ||
| 2 | role | assignedAuthor | Sendendes System | 1..1 | M | Informationen über das sendende System | |
| 3 | role | @classCode | "ASSIGNED" | 1..1 | M | fix | |
| 3 | role | id | ID des sendenden Systems | II | 1..1 | M | |
| 4 | role | @extension | eigentliche Identifikationsnummer des sendenden Systems | 1..1 | M | ||
| 4 | role | @root | OID für Software-Instanzen | 1..1 | M | OID, um Instanzen von Softwaresystemen eindeutig zu identifizieren. Vergeben beispielsweise durch den Softwarehersteller. | |
| 3 | ent | assignedAuthoringDevice | 1..1 | M | |||
| 4 | ent | @classCode | "DEV" | 1..1 | M | fix | |
| 4 | ent | @determinerCode | "INSTANCE" | 1..1 | M | fix | |
| 4 | ent | softwareName | Name der Software(-Instanz) | ST | 1..1 | M |
Beispiel
<author typeCode="AUT">
<time value="20120825"/>
<assignedAuthor classCode="ASSIGNED">
<id extension="eine_id_des_sendenden_systems"
root="1.2.276.0.76.3.1.131.1.4.4.9999.4.999941"/>
<assignedAuthoringDevice classCode="DEV" determinerCode="INSTANCE">
<softwareName>DerHersteller DieSoftware V. 3.2.1 im KKH Musterstadt</softwareName>
</assignedAuthoringDevice>
</assignedAuthor>
</author>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
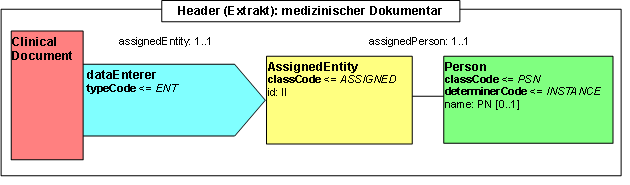
Participant: medizinischer Dokumentar (dataEnterer)
Diese Information gibt an, wer die Information in das System eingegeben hat. Üblicherweise ist dies der medizinische Dokumentar. Dieser Abschnitt ist optional.
|
|
Vom cdaab2-Template ableiten! |
Abbildung: medizinischer Dokumentar
| Lvl | RIM | Name | Desc | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|---|
| 0 | act | ClinicalDocument | |||||
| 1 | part | dataEnterer | Medizinischer Dokumentar | 0..1 | optional | ||
| 2 | part | @typeCode | "ENT" | 1..1 | optional | fix | |
| 2 | role | assignedEntity | 1..1 | M | |||
| 3 | role | @classCode | "ASSIGNED" | 1..1 | M | fix | |
| 3 | role | id | Identifikator des Dokumentars | II | 1..1 | M | |
| 4 | role | @extension | Identifikation des Dokumentars im sendenden System | 1..1 | M | ||
| 4 | role | @root | OID für Dokumentare | 1..1 | M | OID des sendenden Systems, um das Dokumentare eindeutig zu identifizieren | |
| 4 | ent | assignedPerson | Dokumentar | 1..1 | M | ||
| 5 | ent | name | Name des Dokumentars | PN.DE | 0..1 | optional | |
| 6 | ent | prefix | Anrede | ST | 0..1 | optional | z.B. Herr, Frau, ... |
| 6 | ent | prefix | akademischer Titel | ST | 0..1 | optional | |
| 7 | ent | @qualifier | "AC" | 1..1 | optional | fix (Qualifier für akademischen Titel) | |
| 6 | ent | given | Vorname | ST | 0..1 | optional | |
| 6 | ent | family | Nachname | ST | 0..1 | optional |
Beispiel
<dataEnterer typeCode="ENT">
<assignedEntity classCode="ASSIGNED">
<id extension="eine_id_des_dokumentars"
root="1.2.276.0.76.3.1.131.1.4.3.9999.9999.999942"/>
<assignedPerson>
<name>
<prefix>Frau</prefix>
<prefix qualifier="AC">Dr.</prefix>
<given>Martha</given>
<family>Meier</family>
</name>
</assignedPerson>
</assignedEntity>
</dataEnterer>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
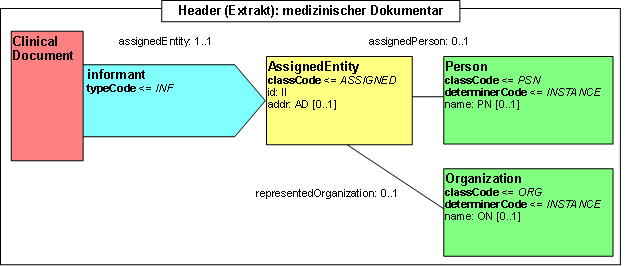
Participant: Arzt, Melder (informant)
Diese Participation gibt an, von wem die gemeldete Information stammt. Normalerweise wird die Information aus einem oder mehreren Arztbriefen entnommen. Hierüber kann angegeben werden, wer diese Arztbriefe verfasst hat. Daher kann dieser Abschnitt mehrfach vorkommen. In der Terminologie der Krebsregister entspricht dies dem Melder.
Abbildung: Arzt, Melder
| Lvl | RIM | AE | Name | Desc | DT | Kard | Conf | Beschreibung
|
|---|---|---|---|---|---|---|---|---|
| 0 | act | elm | ClinicalDocument | |||||
| 1 | part | elm | informant | Arzt, Melder | 1..* | M | ||
| 2 | part | att | @typeCode | "INF" | 1..1 | M | fix | |
| 2 | role | elm | assignedEntity | 1..1 | M | |||
| 3 | role | att | @classCode | "ASSIGNED" | 1..1 | M | fix | |
| 3 | role | elm | id | Identifikator des Melders | II | 1..1 | M | |
| 4 | role | att | @extension | Identifikation des Melders | 1..1 | M | z.B. vom empfangenden Krebsregister vergeben | |
| 4 | role | att | @root | OID für Melder | 1..1 | M | z.B. vom empfangenden Krebsregister vergeben | |
| 3 | role | elm | addr | Adresse | AD | 0..1 | ||
| 4 | role | elm | streetname | Strasse | ST | 0..1 | M | |
| 4 | role | elm | houseNumber | Hausnummer | ST | 0..1 | ||
| 4 | role | elm | postalCode | Postleitzahl | ST | 0..1 | M | PLZ ohne Länderkennzeichen |
| 4 | role | elm | city | Wohnort | ST | 0..1 | M | |
| 4 | role | elm | country | Land | ST | 0..1 | M | |
| 4 | ent | elm | assignedPerson | Melder (Person) | 0..1 | optional | Wird verwendet, wenn der Melder eine Person ist. Es soll immer eine Person oder/und eine Organisation enthalten sein. | |
| 5 | ent | att | @classCode | "PSN" | CS CNE | 1..1 | optional | fix |
| 5 | ent | att | @determinerCode | "INSTANCE" | CS CNE | 1..1 | optional | fix |
| 5 | ent | elm | name | Name des Melders | PN | 0..1 | optional | |
| 6 | ent | elm | prefix | Anrede | ST | 0..1 | optional | z.B. Herr, Frau, ... |
| 6 | ent | elm | prefix | akademischer Titel | ST | 0..1 | optional | |
| 7 | ent | att | @qualifier | "AC" | 1..1 | optional | fix (Qualifier für akademischen Titel) | |
| 6 | ent | elm | given | Vorname | SET<ST> | 0..* | optional | |
| 6 | ent | elm | family | Familienname | ST | 0..1 | optional | |
| 4 | ent | elm | representedOrganization | Melder (Organisation) | 0..1 | optional | Wird verwendet, wenn der Melder eine Organisation ist. | |
| 5 | ent | att | @classCode | "ORG" | CS CNE | 1..1 | optional | fix |
| 5 | ent | att | @determinerCode | "INSTANCE" | CS CNE | 1..1 | optional | fix |
| 5 | ent | elm | name | Name der Organisation | ON | 0..1 | optional |
<informant typeCode="INF">
<assignedEntity classCode="ASSIGNED">
<id extension="eine_id_des_melders" root="1.2.276.0.76.3.1.131.1.4.4.9999.4.999901"/>
<addr>
<streetName>Kliniksweg</streetName>
<houseNumber>125-131</houseNumber>
<country>DE</country>
<postalCode>54321</postalCode>
<city>Musterhausen</city>
</addr>
<assignedPerson classCode="PSN" determinerCode="INSTANCE">
<name>
<prefix qualifier="AC">Dr. med.</prefix>
<given>Thilo</given>
<family>Testmann</family>
</name>
</assignedPerson>
<representedOrganization classCode="ORG" determinerCode="INSTANCE">
<name>Städtisches Klinikum Musterhausen</name>
</representedOrganization>
</assignedEntity>
</informant>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Participant: verwaltende Organisation (custodian)
Wer hat verwaltet das Dokument? Das ist üblicherweise entweder das Krankenhaus oder das Tumorzentrum.
Normalerweise wird das über die Transportinformation gelöst. Wenn das aber persistent mit Bestandteil des Dokumentes sein soll, dann ist der Custodian der beste Platz, weil der "Verwalter" des Dokumentes dann auch am ehesten der "Absender" ist.
Abbildung: Identifikation der verwaltenden Organisation
| Lvl | RIM | Name | Desc | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|---|
| 0 | act | ClinicalDocument | |||||
| 1 | part | custodian | verwaltende Organisation | 1..1 | M | Wer hat verwaltet das Dokument. | |
| 2 | part | @typeCode | "CST" | CS CNE | 1..1 | M | |
| 2 | role | assignedCustodian | 1..1 | M | |||
| 3 | role | @classCode | "ASSIGNED" | CS CNE | 1..1 | M |
|
| 4 | ent | representedCustodianOrganisation | verwaltende/absendede Organisation | 1..1 | |||
| 5 | ent | id | Identifikation der Organisation | SET<II> | 1..* | M | |
| 6 | ent | @extension | eigentliche ID der Organisation | 1..1 | M | In der Regel vom Datenempfänger vergeben | |
| 6 | ent | @root | OID der Organisation | 1..1 | M | In der Regel vom Datenempfänger vergeben | |
| 5 | ent | name | Name der Organisation | ON | 0..1 | ||
| 5 | ent | telecom | Kontaktinformation der Organisation | TEL | 0..0 | nicht verwendet | |
| 5 | ent | addr | Adresse | AD | 0..1 | ||
| 6 | ent | streetname | Strasse | ST | 0..1 | ||
| 6 | ent | houseNumber | Hausnummer | ST | 0..1 | ||
| 6 | ent | postalCode | Postleitzahl | ST | 0..1 | PLZ ohne Länderkennzeichen | |
| 6 | ent | city | Wohnort | ST | 0..1 | ||
| 6 | ent | country | Land | ST | 0..1 |
<custodian typeCode="CST">
<assignedCustodian classCode="ASSIGNED">
<representedCustodianOrganization>
<id extension="eine_id_des_datenlieferanten" root="1.2.276.0.76.3.1.131.1.4.4.9999.4.999961"/>
<name>Regionales KKR für Süd-Ostwestfalen Nord</name>
<addr>
<streetName>Register-Allee</streetName>
<houseNumber>228</houseNumber>
<country>DE</country>
<postalCode>55555</postalCode>
<city>Krebsstadt</city>
</addr>
</representedCustodianOrganization>
</assignedCustodian>
</custodian>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Participant: Empfänger (informationRecipient)
Als Empfänger kommen hier sowohl die Krebsregister als auch Praxen und Krankenhäuser in Frage.
In dem Attribut „informationRecipient" wird angegeben, an welches Krebsregister oder Praxis/Krankenhaus die Daten übermittelt werden. Hier wird die „scoping" Entität "Organisation" (gestrichelte Linie) genutzt.
Abbildung: Identifikation des Empfängers
| Lvl | RIM | Name | Desc | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|---|
| 0 | act | ClinicalDocument | |||||
| 1 | part | informationRecipient | Empfänger | 0..1 | optional | ||
| 2 | part | @typeCode | "PRCP" | CS CNE | 1..1 | M | fix |
| 2 | role | intendedRecipient | 1..1 | M | |||
| 3 | role | @classCode | "ASSIGNED" | 1..1 | M | fix | |
| 3 | role | id | ID des Datenempfängers (z.B. Krebsregister) | II | 0..0 | nicht verwendet | |
| 4 | ent | informationRecipient | 0..0 | nicht verwendet | |||
| 4 | ent | receivedOrganisation | 0..1 | optional | |||
| 5 | ent | name | Name der Organisation | ON.DE | 0..1 | optional | |
| 5 | ent | addr | Adresse | AD.DE | 0..1 | optional |
<informationRecipient typeCode="PRCP">
<intendedRecipient classCode="ASSIGNED">
<receivedOrganization>
<name>Landeskrebsregister NRW</name>
<addr>
<streetName>Robert-Koch-Strasse</streetName>
<houseNumber>40</houseNumber>
<country>DE</country>
<postalCode>48149</postalCode>
<city>Münster</city>
</addr>
</receivedOrganization>
</intendedRecipient>
</informationRecipient>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Versicherungsnehmer und Kostenträger
Abschnitte
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Section: Ergänzende Patientendaten
|
|
TODO: Evtl. wird dieser Abschnitt in der Endfassung als "Nationalität" bezeichnet (mit entsprechend anderem LOINC Code). |
| Template-Metadaten | |
| Template-Typ | Section |
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.11 |
| generischeres Template | |
| genutztes Templates | |
| nutzende Templates | |
| abgeleitete Templates | |
| Schwester-Templates | |
| generelle Beschreibung | In diesem Abschnitt werden ergänzende Daten des Patienten übermittelt (derzeit nur die Nationalität). |
| allg. Erläuterung | |
| Verhältnis zu IHE | dt.Übersetzung oder Ergänzung oder neu |
| Ballotierungsstatus | |
| Erweiterbarkeit | offen oder geschlossen |
Modell
|
|
TODO: hier fehlt das Diagramm |
Attribute
| Lvl | RIM | Name | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|
| 1 | act | section | 0..1 | F | classCode="DOCSECT", moodCode="EVN" | |
| 2 | act | templateId | II | 1..1 | M | @root="1.2.276.0.76.3.1.131.1.10.2.11" |
| 2 | act | code | CE CWE | 0..1 | O | "Patient Information" code="52460-3", codeSystem="2.16.840.1.113883.6.1" |
| 2 | act | title | ST | 1..1 | F | "Ergänzende Patientendaten" |
| 2 | act | text | ED | 1..1 | R | textliche Beschreibung des codierten Inhalts der Section, i.d.R. vom sendenden System automatisch generiert |
| 2 | rel | entry | 1..1 | R | ||
| 3 | act | observation | 1..1 | R | classCode="OBS", moodCode="EVN" | |
| 4 | act | code | CD CWE | 1..1 | R | Country of citizenship code="66476-3", codeSystem="2.16.840.1.113883.6.1" (LOINC) |
| 4 | act | text | ED | 1..1 | R | textliche Beschreibung des codierten Inhalts der Observation, i.d.R. vom sendenden System automatisch generiert |
| 4 | act | value | CD | 1..1 | R | Nationalität code=Landescode (ISO-3166-1 ALPHA-2), Value Set: 1.3.6.1.4.1.12559.11.10.1.3.1.42.4 (VS11_epSOSCountry) |
Beispiel
<section>
<templateId root="1.2.276.0.76.3.1.131.1.10.2.11"/>
<code code="52460-3" codeSystem="2.16.840.1.113883.6.1"
displayName="Patient Information" />
<title>Ergänzende Patientendaten</title>
<text>
<paragraph>
Nationalität: deutsch
</paragraph>
</text>
<entry>
<observation classCode="OBS" moodCode="EVN">
<code code="66476-3" codeSystem="2.16.840.1.113883.6.1"/>
<text>
Nationalität: deutsch
</text>
<value xsi:type="CD" code="DE" codeSystem="1.0.3166.1.2.2"/>
</observation>
</entry>
</section>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Section: Meldebegründung
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.1 | |
| General Description | In diesem Abschnitt wird die Begründung für die Meldung übermittelt. | |
| LOINC Code | Opt. | Description |
| 64292-6 | O | Release of information consent |
Eine Meldebegründung gibt den rechtlichen Kontext der Meldung wieder. Dies unterscheidet sich von Bundesland zu Bundesland. In Ländern mit Meldepflicht muss der Patient in der Regel informiert werden. Ausnahmen gibt es ggf. wenn der Patient verstorben ist oder ihm eine Mitteilung nicht zugemutet werden kann (oder bei Meldungen von Pathologen). Bei Meldepflicht gibt es grundsätzlich ein Widerspruchsrecht. Bei Melderecht muss der Patient zustimmen.
Weitere Variationen bestehen im Einverständnis zur Nutzung der Daten für weitergehende Forschung. An dieser Stelle sollte auch die Zustimmung zur Meldung ans klinische Krebsregister berücksichtigt werden. Diese kann ggf. zukünftig auch nach unterschiedlichen Nutzungszwecken unterschieden werden und wird zu einem späteren Zeitpunkt berücksichtigt.
Abbildung: Observation für die Meldebegründung
| Lvl | RIM | Name | Desc | DT | Kard | Conf | Beschreibung | ||
|---|---|---|---|---|---|---|---|---|---|
| 1 | act | section | 1..1 | required | |||||
| 2 | act | @classCode | "DOCSECT" | CS CNE | 1..1 | required | |||
| 2 | act | @moodCode | "EVN" | CS CNE | 1..1 | required | |||
| 2 | act | templateID | Meldebegründungsabschnitt | II | 1..1 | required | |||
| 3 | act | @root | "1.2.276.0.76.3.1.131.1.10.2.1" | 1..1 | required | fix | |||
| 2 | act | code | Code für Meldebegründungsabschnitt | CE CWE | 0..1 | optional | |||
| 3 | act | @code | "64292-6" | 1..1 | optional | fix | |||
| 3 | act | @codeSystem | "2.16.840.1.113883.6.1" | 1..1 | optional | fix | |||
| 2 | act | title | "Meldebegründung" | ST | 1..1 | required | fix | ||
| 2 | act | text | textliche Beschreibung des codierten Inhalts der Section | ED | 1..1 | required | i.d.R. vom sendenden System automatisch generiert | ||
| 2 | rel | entry | 1..1 | required | |||||
| 3 | act | observation | 1..1 | required | |||||
| 4 | act | @classCode | "OBS" | CS CNE | 1..1 | required | fix | ||
| 4 | act | @moodCode | "EVN" | CS CNE | 1..1 | required | fix | ||
| 4 | act | id | ID der Meldebegründung | II | 0..1 | optional | |||
| 5 | act | root | OID für Meldebegründungen | 1..1 | optional | OID des sendenden System, um Meldebegründungen eindeutig zu identifizieren | |||
| 5 | act | @extension | ID der Meldebegründung im sendenden System | 1..1 | optional | ||||
| 4 | act | code | CD CWE | 0..1 | required | Markierung dieser Observation als Meldebegründung | |||
| 5 | act | @code | "gekidMeldebegruendung" | 1..1 | required | fix: GEKID Meldebegründung | |||
| 5 | act | @codeSystem | "1.2.276.0.76.3.1.131.1.5.1" | 1..1 | optional | fix: DKG-Labels
| |||
| 4 | act | text | textliche Beschreibung des codierten Inhalts der Observation | ED | 1..1 | required | i.d.R. vom sendenden System automatisch generiert | ||
| 4 | act | effectiveTime | Zeitpunkt der Meldebegründung | TS | 0..1 | optional | Das Datum, an dem der Patient unterrichtet wurde oder an dem er zugestimmt hat. | ||
| 4 | act | value | Meldebegrüdung | CD | 1..1 | required | |||
| 5 | act | @code | eigentliche Meldebegründung | 1..1 | required | Value Set: abhängig vom Bundesland | |||
| 5 | act | @codeSystem | "1.2.276.0.76.3.1.131.1.5.2" | 1..1 | required | fix: GEKID Meldebegründung
| |||
| 5 | act | qualifier | CR | 0..* | optional | für jeden Gültigkeitsbereich darf maximal 1 Qualifier übermittelt werden | |||
| 6 | act | name | Gültigkeitsbereich der Zustimmung | CV | 1..1 | required | |||
| 7 | act | @code | Code für den Gültigkeitsbereich | 1..1 | required | Codesystem: 1.2.276.0.76.3.1.131.1.5.63 | |||
| 7 | act | @codeSystem | "1.2.276.0.76.3.1.131.1.5.63" | 1..1 | required | fix | |||
| 6 | act | value | ja/nein-Angabe | CV | 1..1 | required | |||
| 7 | act | @code | "true" oder "false" | 1..1 | required | Codesystem: 1.2.276.0.76.3.1.131.1.5.61 | |||
| 7 | act | @codeSystem | "1.2.276.0.76.3.1.131.1.5.61" | 1..1 | required | fix | |||
| 4 | part | author | 0..0 | nicht verwendet |
|
|
Bei nachträglichem Widerspruch zur Übermittlung sind die vorher übermittelten Daten wieder zu löschen. TODO: Hierfür sollte eine separate Dokument-Art spezifiziert werden. Da jedoch der Widerspruch zur Übermittlung untergeordnete Bedeutung besitzt und auch die organisatorischen und gesetzlichen Rahmenbedingungen nicht geklärt sind, wird dies in einer späteren Version dieses Leitfadens geschehen. |
Qualifier: Gültigkeitsbereich der Zustimmung
In einem bzw. mehreren Qualifiern kann angegeben werden, an welche Empfänger die im Dokument enthaltenen Daten übermittelt werden dürfen oder nicht.
Wird kein solcher Qualifier übermittelt, so wird von einer Zustimmung für die Übermittlung an das epidemiologische Krebsregister bzw. das Landeskrebsregister ausgegangen.
|
|
TODO: Diese Aussage muss noch geprüft werden! |
Beispiele
Beispiel 1
Das 1. Beispiel entspricht der obigen Tabelle. Es wird zukünftig in dem 2. Beispiel aufgehen. Da das 2. Beispiel noch nicht ausmodelliert ist, ist bis auf weiteres das 1. Beispiel relevant.
<section classCode="DOCSECT" moodCode="EVN">
<templateId root="1.2.276.0.76.3.1.131.1.10.2.1"/>
<code code="64292-6" codeSystem="2.16.840.1.113883.6.1"/>
<title>Meldebegründungsdaten</title>
<text>
<paragraph>
Der Patient hat in die Meldung an das epidemiologische Krebsregister eingewilligt.
Der Patient hat der Meldung an das klinische Krebsregister widersprochen.
</paragraph>
</text>
<entry>
<observation classCode="OBS" moodCode="EVN">
<id extension="eine_meldebegruendungs_id"
root="1.2.276.0.76.3.1.131.1.4.3.9999.9999.999916"/>
<code code="gekidMeldebegruendung"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
Patient hat eingewilligt
Gilt für epidemiologisches Krebsregister
Gilt nicht für epidemiologisches Krebsregister
</text>
<effectiveTime value="20120724"/>
<value xsi:type="CD" code="E"
codeSystem="1.2.276.0.76.3.1.131.1.5.2">
<qualifier>
<name code="EKR"
codeSystem="1.2.276.0.76.3.1.131.1.5.63"/>
<value xsi:type="CD" code="true"
codeSystem="1.2.276.0.76.3.1.131.1.5.61"/>
</qualifier>
<qualifier>
<name code="KKR"
codeSystem="1.2.276.0.76.3.1.131.1.5.63"/>
<value xsi:type="CD" code="false"
codeSystem="1.2.276.0.76.3.1.131.1.5.61"/>
</qualifier>
</value>
</observation>
</entry>
</section>
Beispiel 2
<section>
<templateId root="1.2.276.0.76.3.1.131.1.10.2.1"/>
<code code="?????" codeSystem="2.16.840.1.113883.6.1"
codeSystemName="LOINC"/>
<title>Meldebegründung</title>
<text>Der Patient hat in die Meldung eingewilligt.</text>
<entry>
<!-- Meldebegründung mit Datum -->
<observation classCode="OBS" moodCode="EVN">
<id>3473847/01</id> <!-- optionale ID für Referenzzwecke -->
<!-- Wo würde dargestellt, wo die Meldebegründung erstellt wurde,
Referenz auf Participant -->
<code code="gekidMeldebegruendung" codeSystem="1.2.276.0.76.3.1.131.1.5.1"
codeSystemName="DKG-Labels" displayName="Meldebegründung"/>
<statusCode code="completed"/>
<effectiveTime>
<low value="20101022"/>
</effectiveTime>
<value xsi:type="CD"
code="E" displayName="Meldung an EKR-BW "
codeSystem="1.2.276.0.76.3.1.131.1.5.2"
codeSystemName="Meldebegruendung"/>
</value>
</observation>
</entry>
<entry>
<!-- Kodierung der Zustimmung -->
<observation classCode="OBS" moodCode="EVN">
<code code="?????" codeSystem="??????"
codeSystemName="LOINC" displayName="Zustimmung"/>
<statusCode code="completed"/>
<value xsi:type="CD"
code="????" displayName="?????"
codeSystem="?????"
codeSystemName="?????"/>
</value>
</observation>
</entry>
<entry>
<!-- Kodierung der Information -->
<observation classCode="OBS" moodCode="EVN">
<code code="?????" codeSystem="??????"
codeSystemName="LOINC" displayName="Information über Meldung"/>
<statusCode code="completed"/>
<value xsi:type="CD"
code="????" displayName="?????"
codeSystem="?????"
codeSystemName="?????"/>
</value>
</observation>
</entry>
<entry>
<!-- Kodierung des Widerspruchs -->
<observation classCode="OBS" moodCode="EVN">
<code code="?????" codeSystem="??????"
codeSystemName="LOINC" displayName="Widerspruch gegen Meldung"/>
<statusCode code="completed"/>
<value xsi:type="CD"
code="????" displayName="?????"
codeSystem="?????"
codeSystemName="?????"/>
</value>
</observation>
</entry>
</section>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Section: Diagnostik
| Template-Metadaten | |
| Template-Typ | Section |
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.3 |
| generischeres Template | |
| genutzte Templates | TNM-Klassifikation R-Klassifikation |
| nutzende Templates | |
| abgeleitete Templates | |
| Schwester-Templates | |
| Generelle Beschreibung | In diesem Abschnitt werden die Diagnostikdaten zum Patienten übermittelt. |
| allg. Erläuterung | |
| Verhältnis zu IHE | dt.Übersetzung oder Ergänzung oder neu |
| Ballotierungsstatus | in Arbeit |
| Erweiterbarkeit | offen |
Beschreibung
Die Diagnostik umfasst die in den Unterabschnitten aufgeführten Punkte, die jeweils über separate Observations ausgedrückt werden.
Modell
Abbildung: Diagnostik
genutzte Entries
Folgende Entries werden für die Diagnostik benötigt:
| Entry | Conf. | Kard. | Bemerkung |
|---|---|---|---|
| TNM-Klassifikation | O | [0..*] | es können wir patholog. als auch klinische TNM-Bewertungen übermittelt werden. |
| R-Klassifikation | O | [0..1] | |
| Morphologie | O | [0..1] | |
| Frühere Tumorerkrankung | O | [0..1] | |
| Generische Observation mit Messwert | O | [0..1] | |
| Generische Observation mit boolescher Angabe | O | [0..1] | |
| Generische Observation mit codierter Angabe | O | [0..1] | |
| Ann-Arbor-Stadium | O | [0..1] | |
| Gleason-Score | O | [0..1] |
Attribute
Die nachfolgende Tabelle stellt das Grundgerüst für den Diagnostik-Abschnitt dar, das in den jeweiligen Unterabschnitten als Entry Template detailliert ist.
| Lvl | RIM | Name | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|
| 1 | act | section | 1..1 | F | classCode="DOCSECT", moodCode="EVN" | |
| 2 | act | templateId | II | 1..1 | R | Diagnostik-Abschnitt root="1.2.276.0.76.3.1.131.1.10.2.3" |
| 2 | act | code | CE CWE | 0..1 | O | Code für Diagnostik-Abschnitt code="29308-4" (Diagnosis), codeSystem="2.16.840.1.113883.6.1" (LOINC) |
| 2 | act | title | ST | 1..1 | F | "Diagnostik" |
| 2 | act | text | ED | 1..1 | R | textliche Beschreibung des codierten Inhalts der Section, i.d.R. vom sendenden System automatisch generiert |
| 2 | rel | entry | 1..* | R | ||
| 3 | rel | @typeCode | 1..1 | R | ||
| 3 | act | observation | 1..1 | R | Observation: Unterelemente lt. Entry Template classCode="OBS", moodCode="EVN" | |
| 4 | act | ... | zur Detaillierung der möglichen Observations vgl. die Unterabschnitte (Entry Templates) |
Beispiel
<section classCode="DOCSECT" moodCode="EVN">
<templateId root="1.2.276.0.76.3.1.131.1.10.2.3"/>
<code code="29308-4" codeSystem="2.16.840.1.113883.6.1"/>
<title>Diagnostik</title>
<text>
<paragraph>
Histologiebefund H4711/2012 der Pathologie Universitätsklinikum Musterstadt vom 24.03.2011:<br/>
Intraduktales papilläres Adenokarzinom mit Invasion, G2, T-Zell.<br/>
Primärtumor ist gesichert, Grobstadium: lokal begrenzt<br/>
Anlass der Diagnose: Screening
</paragraph>
<paragraph>
Früherer Tumor ist bekannt:<br/>
05/2009: Bösartige Neubildung Colon ascendens<br/>
Frühere Chemotherapie: ja<br/>
Frühere Strahlentherapie: unbekannt
</paragraph>
<list>
<caption>Begleiterkrankungen:</caption>
<item>Diabetes mellitus mit Ketoazidose seit 11/2003</item>
</list>
<paragraph>
Tumorklassifikation vom 26.03.2011: UICC IV<br/>
yr pT3C4 pN3(SN)C5(4/5) cM1C2 S2 L1 V1 PnX<br/>
Lymphknoten befallen/untersucht: 13/18
</paragraph>
<paragraph>
...
</paragraph>
...
</text>
<entry>
<observation classCode="OBS" moodCode="EVN">
...
</observation>
</entry>
<entry>
<observation classCode="OBS" moodCode="EVN">
...
</observation>
</entry>
...
</section>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Entry: Morphologie
| Template-Metadaten | |
| Template-Typ | Entry |
| Template ID | 1.2.276.0.76.3.1.131.1.10.3.3 |
| generischeres Template | |
| genutztes Templates | |
| nutzende Templates | |
| abgeleitete Templates | |
| Generelle Beschreibung | Angaben zur Morphologie des Tumors |
| allg. Erläuterung | |
| Verhältnis zu IHE | dt.Übersetzung oder Ergänzung oder neu |
| Ballotierungsstatus | |
| Erweiterbarkeit | offen oder geschlossen |
Modell
|
|
TODO: Abgleich mit dem Diagnoseleitfaden ist erforderlich! |
|
|
TODO: Abgleich mit ICD-O Template aus APSR 2.0 erforderlich! |
Abbildung: Morphologie
Attribute
| Lvl | RIM | Name | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|
| 1 | act | observation | 1..1 | required | ||
| 4 | act | @classCode | CS CNE | 1..1 | F | "OBS" |
| 4 | act | @moodCode | CS CNE | 1..1 | F | "EVN" |
| 4 | act | templateId | II | 1..1 | required | Morphologie |
| 5 | act | @root | 1..1 | F | "1.2.276.0.76.3.1.131.1.10.3.3" | |
| 4 | act | id | II | 0..1 | optional | ID der diagnostischen Angabe |
| 5 | act | @root | 1..1 | optional | OID für diagnostische Angaben: OID des sendenden System, um diagnostische Angaben eindeutig zu identifizieren | |
| 5 | act | @extension | 1..1 | optional | ID der diagnostischen Angabe im sendenden System | |
| 4 | act | code | CD CWE | 1..1 | required | |
| 5 | act | @code | 1..1 | F | "31205-8" | |
| 5 | act | @codeSystem | 1..1 | fix: LOINC | "2.16.840.1.113883.6.1" | |
| 4 | act | text | ED | 1..1 | R | textliche Beschreibung des codierten Inhalts der Observation, i.d.R. vom sendenden System automatisch generiert |
| 4 | act | effectiveTime | TS | 0..1 | optional | Das Datum, an dem die Diagnose gestellt wurde |
| 4 | act | value | CD | 1..1 | required | Code für die Morphologie |
| 5 | act | @code | 1..1 | R | eigentlicher Code: Codesystem 2.16.840.1.113883.6.43.1 | |
| 5 | act | @codeSystem | 1..1 | F | "2.16.840.1.113883.6.43.1" | |
| 5 | act | originalText | ED | 0..1 | optional | Freitext des Originalbefundes |
| 5 | act | CR | 0..1 | optional | qualifier | |
| 6 | act | name | CV | 1..1 | required | Dignität |
| 7 | act | @code | 1..1 | fix | "335" | |
| 7 | act | @codeSystem | 1..1 | fix: SCIPHOX | "2.16.840.1.113883.3.7.1.0" | |
| 6 | act | value | CV | 1..1 | required | Code für die Dignität |
| 7 | act | @code | 1..1 | required | eigentlicher Code, Codesystem 1.2.276.0.76.5.335 | |
| 7 | act | @codeSystem | 1..1 | fix | "1.2.276.0.76.5.335" | |
| 5 | act | qualifier | CR | 0..1 | optional | |
| 6 | act | name | CV | 1..1 | required | histopathologisches Grading |
| 7 | act | @code | 1..1 | fix | "336" | |
| 7 | act | @codeSystem | 1..1 | fix: SCIPHOX | "2.16.840.1.113883.3.7.1.0" | |
| 6 | act | value | CV | 1..1 | required | Code für das histopathologische Grading |
| 7 | act | @code | 1..1 | required | eigentlicher Code, Codesystem 1.2.276.0.76.5.336 | |
| 7 | act | @codeSystem | 1..1 | fix | "1.2.276.0.76.5.336" | |
| 5 | act | qualifier | CR | 0..1 | optional | |
| 6 | act | name | CV | 1..1 | required | Immunophänotyp |
| 7 | act | @code | 1..1 | fix | "413" | |
| 7 | act | @codeSystem | 1..1 | fix: SCIPHOX | "2.16.840.1.113883.3.7.1.0" | |
| 6 | act | value | CV | 1..1 | required | Code für den Immunophänotyp |
| 7 | act | @code | 1..1 | required | eigentlicher Code, Codesystem 1.2.276.0.76.5.??? TODO: diese OID .413 wurde doppelt vergeben (auch für ICD-10-GM 2013!) | |
| 7 | act | @codeSystem | 1..1 | required | fix TODO: diese OID .413 wurde doppelt vergeben (auch für ICD-10-GM 2013!) "1.2.276.0.76.5.???" | |
| 4 | part | author | 0..1 | optional | Autor des Pathologie-Befundes, wird nur übermittelt, wenn das pathologische Institut bekannt ist | |
| 5 | part | @typeCode | CS CNE | 1..1 | fix | "AUT" |
| 5 | part | time | TS | 1..1 | required | Zeitpunkt, nur aufgrund von CDA-Konformität |
| 6 | part | @nullFlavor | 1..1 | fix | "UNK" | |
| 5 | role | assignedAuthor | 1..1 | required | Pathologisches Institut, Informationen über das pathologische Institut | |
| 6 | role | @classCode | 1..1 | fix | "ASSIGNED" | |
| 6 | role | id | II | 1..1 | M | ID des sendenden Systems für Pathologische Institute; Es muss entweder die Kombination der Attribute root und extension oder das Attribut nullFlavor übermittelt werden |
| 7 | role | @root | 0..1 | M | OID des sendenden Systems für Pathologische Institute | |
| 7 | role | @extension | 0..1 | M | eigentliche Identifikationsnummer des Pathologischen Instituts | |
| 7 | role | @nullFlavor | 0..1 | R | verwenden, wenn keine ID des pathologischen Instituts bekannt ist; "UNK" | |
| 6 | ent | assignedPerson | 1..1 | required | nur aufgrund von CDA-Konformität | |
| 7 | ent | @nullFlavor | 1..1 | fix | "UNK" | |
| 6 | ent | representedOrganization | 1..1 | required | Pathologisches Institut | |
| 7 | ent | @classCode | 1..1 | required | "ORG" | |
| 7 | ent | @determinerCode | 1..1 | required | "INSTANCE" | |
| 7 | ent | name | ON.DE | 1..1 | required | Name des pathologischen Instituts |
| 4 | rel | reference | 0..1 | optional | ||
| 5 | rel | @typeCode | 1..1 | fix | "REFR" | |
| 5 | act | externalDocument | 1..1 | required | ||
| 6 | act | @classCode | 1..1 | fix | "DOC" | |
| 6 | act | @moodCode | 1..1 | fix | "EVN" | |
| 6 | act | id | II | 0..1 | M | ID (Befundnummer) des Pathologie-Befundes; Wenn keine OID verfügbar ist, kann die Befundnummer auch ausschließlich im text-Element übermittelt werden |
| 7 | act | @root | 1..1 | required | OID für Befunde eines bestimmten pathologischen Instituts; OID des sendenden System, um Befunde eines bestimmten pathologischen Instituts eindeutig zu identifizieren | |
| 7 | act | @extension | 1..1 | required | Befundnummer des Pathologie-Befundes | |
| 6 | act | text | ED | 1..1 | required | Befundnummer des Pathologie-Befundes |
Beispiel
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.276.0.76.3.1.131.1.10.3.3"/>
<id extension="diag12345"
root="1.2.276.0.76.3.1.131.1.4.3.9999.9999.999907"/>
<code code="31205-8"
codeSystem="2.16.840.1.113883.6.1"/>
<text>
Histologiebefund der Pathologie Universitätsklinikum Musterstadt vom 24.03.2011:
Intraduktales papilläres Adenokarzinom mit Invasion, G2, T-Zell
</text>
<effectiveTime value="20110324"/>
<value xsi:type="CD" code="8503"
codeSystem="2.16.840.1.113883.6.43.1">
<originalText>
Intraduktales papilläres Adenokarzinom mit Invasion,
G2, T-Zell
</originalText>
<qualifier>
<name code="335"
codeSystem="2.16.840.1.113883.3.7.1.0"/>
<value xsi:type="CD" code="3"
codeSystem="1.2.276.0.76.5.335"/>
</qualifier>
<qualifier>
<name code="336"
codeSystem="2.16.840.1.113883.3.7.1.0"/>
<value xsi:type="CD" code="2"
codeSystem="1.2.276.0.76.5.336"/>
</qualifier>
<qualifier>
<name code="413"
codeSystem="2.16.840.1.113883.3.7.1.0"/>
<value xsi:type="CD" code="5"
codeSystem="1.2.276.0.76.5.413"/>
</qualifier>
</value>
<author typeCode="AUT">
<time nullFlavor="UNK"/>
<assignedAuthor classCode="ASSIGNED">
<id nullFlavor="UNK"/>
<assignedPerson nullFlavor="UNK"/>
<representedOrganization classCode="ORG" determinerCode="INSTANCE">
<name>Pathologie Universitätsklinikum Musterstadt</name>
</representedOrganization>
</assignedAuthor>
</author>
<reference typeCode="REFR">
<externalDocument classCode="DOC" moodCode="EVN">
<id extension="H4711/2012"
root="1.2.276.0.76.3.1.131.1.4.3.9999.9999.999955"/>
<text>H4711/2012</text>
</externalDocument>
</reference>
</observation>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Entry: frühere Tumorerkrankungen
| Template-Metadaten | |
| Template-Typ | Entry |
| Template ID | 1.2.276.0.76.3.1.131.1.10.3.4 |
| generischeres Template | |
| genutztes Templates | |
| nutzende Templates | |
| abgeleitete Templates | |
| Generelle Beschreibung | Angaben zu früheren Tumorerkrankungen des Patienten |
| allg. Erläuterung | |
| Verhältnis zu IHE | dt.Übersetzung oder Ergänzung oder neu |
| Ballotierungsstatus | |
| Erweiterbarkeit | offen oder geschlossen |
Modell
Abbildung: frühere Tumorerkrankungen
Attribute
| Lvl | RIM | Name | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|
| 3 | act | observation | 1..1 | required | ||
| 4 | act | @classCode | CS CNE | 1..1 | fix | "OBS" |
| 4 | act | @moodCode | CS CNE | 1..1 | fix | "EVN" |
| 4 | act | templateId | II | 1..1 | required | frühere Tumorerkrankungen |
| 5 | act | @root | 1..1 | fix | "1.2.276.0.76.3.1.131.1.10.3.4" | |
| 4 | act | id | II | 0..1 | optional | ID der diagnostischen Angabe |
| 5 | act | @root | 1..1 | optional | OID für diagnostische Angaben: OID des sendenden System, um diagnostische Angaben eindeutig zu identifizieren | |
| 5 | act | @extension | 1..1 | optional | ID der diagnostischen Angabe im sendenden System | |
| 4 | act | code | CD CWE | 1..1 | required | |
| 5 | act | @code | 1..1 | fix | "56837-8" | |
| 5 | act | @codeSystem | 1..1 | F | LOINC: "2.16.840.1.113883.6.1" | |
| 4 | act | text | ED | 1..1 | required | textliche Beschreibung des codierten Inhalts der Observation, i.d.R. vom sendenden System automatisch generiert |
| 4 | act | effectiveTime | TS | 0..1 | optional | Zeitpunkt der Feststellung: Das Datum, an dem die Information festgestellt wurde |
| 4 | act | value | BL | 1..1 | required | Es wird entweder das value-Attribut oder das nullFlavor-Attribut übermittelt, Ja/Nein-Angabe, ob frühere Tumoren bekannt sind |
| 5 | act | @value | 0..1 | M | "true" oder "false" | |
| 5 | act | @nullFlavor | 0..1 | fix | "UNK" | |
| 4 | rel | entryRelationship | 0..* | optional | ||
| 5 | rel | @typeCode | 1..1 | fix | "REFR" | |
| 5 | act | observation | 1..1 | required | Angaben zu einem früheren Tumor | |
| 6 | act | @classCode | CS CNE | 1..1 | fix | "OBS" |
| 6 | act | @moodCode | CS CNE | 1..1 | fix | "EVN" |
| 6 | act | id | II | 0..1 | optional | ID der früheren Tumorerkrankung |
| 7 | act | @root | 1..1 | required | OID für früheren Tumorerkrankungen, OID des sendenden System, um frühere Tumorerkrankungen eindeutig zu identifizieren | |
| 7 | act | @extension | 1..1 | required | ID der früheren Tumorerkrankung im sendenden System | |
| 6 | act | code | CD CWE | 1..1 | required | |
| 7 | act | @code | 1..1 | fix | "NEO" | |
| 7 | act | @codeSystem | 1..1 | F | HL7-D Typisierung Diagnose, "1.2.276.0.76.5.342" | |
| 6 | act | text | ED | 1..1 | required | textliche Beschreibung des codierten Inhalts der Observation, i.d.R. vom sendenden System automatisch generiert |
| 6 | act | effectiveTime | TS | 0..1 | optional | Zeitpunkt der Diagnose der früheren Tumorerkrankung: Das Datum, an dem die frühere Tumorerkrankung diagnostiziert wurde |
| 6 | act | value | CD | 1..1 | required | Diagnose der früheren Tumorerkrankung |
| 7 | act | @code | 1..1 | required | Code für die frühere Tumorerkrankung: Codesystem ICD-10 (OID: jahresabhängig) | |
| 7 | act | @codeSystem | 1..1 | required | OID des Codesystems: ICD-10 (jahresabhängig) |
Beispiel
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.276.0.76.3.1.131.1.10.3.4"/>
<id extension="1234004"
root="1.2.276.0.76.3.1.131.1.4.3.9999.9999.999919"/>
<code code="56837-8"
codeSystem="2.16.840.1.113883.6.1"/>
<text>
Früherer Tumor: ja
</text>
<effectiveTime value="20110326"/>
<value xsi:type="BL" value="true"/>
<entryRelationship typeCode="REFR">
<observation classCode="OBS" moodCode="EVN">
<id extension="frtumor12345"
root="1.2.276.0.76.3.1.131.1.4.3.9999.9999.999908"/>
<code code="NEO"
codeSystem="1.2.276.0.76.5.342"/>
<text>
05/2009: Bösartige Neubildung Colon ascendens
</text>
<effectiveTime value="200905"/>
<value xsi:type="CD" code="C18.2"
codeSystem="1.2.276.0.76.5.356"/>
</observation>
</entryRelationship>
<entryRelationship typeCode="REFR">
<observation classCode="OBS" moodCode="EVN">
...
</observation>
</entryRelationship>
...
</observation>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Entry: Ann Arbor Stadium
| Template-Metadaten | |
| Template-Typ | Entry |
| Template ID | 1.2.276.0.76.3.1.131.1.10.3.6 |
| generischeres Template | cdaab2:Assessment_Scales_Entry_(Template) |
| genutztes Template | |
| nutzende Templates | |
| abgeleitete Templates | |
| Schwester-Templates | cdaab2:Gleason-Score-Entry_(Template) |
| Generelle Beschreibung | In diesem Entry wird das Ann Arbor Stadium übermittelt. |
| allg. Erläuterung | Scores_und_Assessments |
| Verhältnis zu IHE | |
| Ballotierungsstatus | in Arbeit |
| Erweiterbarkeit | offen oder geschlossen |
|
|
TODO: Modell verifizieren, Abgleich mit Diagnoseleitfaden |
Bei Lymphomen ist die TNM Klassifikation nicht sinnvoll, hier wird die Ann-Arbor Klassifikation angewendet. Bei der Ann-Arbor Klassifikation werden die Stadien I-IV unterschieden, denen ohne Allgemeinsymptome jeweils A, mit Allgemeinsymptomen B, bei Befall eines extralymphatischen Organs E und bei Befall der Milz S zugeordnet werden z.B. IIIB E+S.
Auch bei der Ann-Arbor Klassifikation wird heute zwischen einer klinischen (cS) und einer pathologischen (pS) Stadieneinteilung unterschieden.
Weitere Stadieneinteilung sind die nach Durie & Salmon mit den Stadien I-III und die SWOG Stadieneinteilung mit den Stadien 1-4 beim Multiplen Myelom.
Modell
Abbildung: Ann Arbor Stadium
|
|
Der extranodale Befall, die klinische Symptomatik und eine Konglomerattumorbildung werden als Qualifier zum Ann-Arbor-Stadium ausgedrückt, die Benennung eines konkreten extranodalen und extrasplenischen Organs ist nicht Bestandteil des originären Ann-Arbor Stadiums. Das Schema muss modifiziert werden |
Attribute
| Lvl | RIM | Name | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|
| 3 | act | observation | 1..1 | R | Ann-Arbor | |
| 4 | act | @classCode | CS CNE | 1..1 | F | "OBS" |
| 4 | act | @moodCode | CS CNE | 1..1 | F | "EVN" |
| 4 | act | templateId | II | 1..1 | R | Ann Arbor Stadium |
| 5 | act | @root | 1..1 | F | "1.2.276.0.76.3.1.131.1.10.3.6" | |
| 4 | act | id | II | 0..1 | O | ID der diagnostischen Angabe |
| 5 | act | @root | 1..1 | O | OID für diagnostische Angaben: OID des sendenden System, um diagnostische Angaben eindeutig zu identifizieren | |
| 5 | act | @extension | 1..1 | O | ID der diagnostischen Angabe im sendenden System | |
| 4 | act | code | CD CWE | 1..1 | R | Ann Arbor Stadium |
| 5 | act | @code | 1..1 | R | "AnnArborStage" | |
| 5 | act | @codeSystem | 1..1 | F | DKG-Labels: "1.2.276.0.76.3.1.131.1.5.1" | |
| 4 | act | text | ED | 1..1 | R | textliche Beschreibung des codierten Inhalts der Observation, i.d.R. vom sendenden System automatisch generiert |
| 4 | act | effectiveTime | TS | 0..1 | O | Zeitpunkt der Feststellung: Das Datum, an dem die Diagnose gestellt wurde |
| 4 | act | value | CD | 1..1 | R | Code für das Ann Arbor Stadium |
| 5 | act | @code | 1..1 | R | eigentlicher Code: Codesystem 1.2.276.0.76.5.405 | |
| 5 | act | @codeSystem | 1..1 | F | "1.2.276.0.76.5.405" | |
| 4 | rel | entryRelationship | 0..1 | O | ||
| 5 | rel | @typeCode | 1..1 | R | "COMP" | |
| 5 | act | observation | 1..1 | R | ||
| 6 | act | @classCode | CS CNE | 1..1 | F | "OBS" |
| 6 | act | @moodCode | CS CNE | 1..1 | F | "EVN"
|
| 6 | act | code | CD CWE | 1..1 | R | Ann Arbor B-Symptome |
| 7 | act | @code | 1..1 | F | "AnnArborBSymptoms" | |
| 7 | act | @codeSystem | 1..1 | F | DKG-Labels "1.2.276.0.76.3.1.131.1.5.1" | |
| 6 | act | value | CD | 1..1 | R | |
| 7 | act | @code | 1..1 | R | Code für Ann Arbor B-Symptome: Codesystem 1.2.276.0.76.5.xxx | |
| 7 | act | @codeSystem | 1..1 | F | "1.2.276.0.76.5.xxx" | |
| 4 | rel | entryRelationship | 0..* | O | ||
| 5 | rel | @typeCode | 1..1 | F | "COMP" | |
| 5 | act | observation | 1..1 | R | ||
| 6 | act | @classCode | CS CNE | 1..1 | F | "OBS" |
| 6 | act | @moodCode | CS CNE | 1..1 | F | "EVN" |
| 6 | act | code | CD CWE | 1..1 | R | Ann Arbor extranodaler Befall |
| 7 | act | @code | 1..1 | F | "AnnArborExtranodal" | |
| 7 | act | @codeSystem | 1..1 | F | DKG-Labels: "1.2.276.0.76.3.1.131.1.5.1" | |
| 6 | act | value | CD | 1..1 | R | |
| 7 | act | @code | 1..1 | R | Code für Ann Arbor extranodalen Befall: Codesystem 1.2.276.0.76.5.yyy | |
| 7 | act | @codeSystem | 1..1 | F | "1.2.276.0.76.5.yyy" | |
| 7 | act | qualifier | CR | 0..* | O | |
| 8 | act | name | CV | 1..1 | R | Angabe zum Organbefall |
| 9 | act | @code | 1..1 | R | Organ: Value Set 1.2.276.0.76.3.1.131.1.11.5 | |
| 9 | act | @codeSystem | 1..1 | F | SNOMED CT: "2.16.840.1.113883.6.96" | |
| 8 | act | value | CV | 1..1 | R | Es wird entweder die Kombination der Attribute code und codeSystem oder das Attribut nullFlavor übermittelt; Ja/Nein-Angabe bzw. unbekannt |
| 9 | act | @code | 0..1 | M | Codesystem 1.2.276.0.76.3.1.131.1.5.61: "true" oder "false" | |
| 9 | act | @codeSystem | 0..1 | F | "1.2.276.0.76.3.1.131.1.5.61" | |
| 9 | act | @nullFlavor | 0..1 | F | "UNK" |
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.276.0.76.3.1.131.1.10.3.6"/>
<id extension="1234017"
root="1.2.276.0.76.3.1.131.1.4.3.9999.9999.999919"/>
<code code="AnnArborStage"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
Ann Arbor III/BS
befallene Organe: Milz, Lunge, Leber, Pleura, Nebenniere, Haut
</text>
<effectiveTime value="20110326"/>
<value xsi:type="CD" code="III"
codeSystem="1.2.276.0.76.5.405">
<qualifier>
<!-- Allgmeinsymptome -->
<name code="xxx"
codeSystem="2.16.840.1.113883.6.96"/>
<value xsi:type="CD" code="A"
codeSystem="1.2.276.0.76.5.xxx"/>
</qualifier>
<qualifier>
<!-- Organbefall -->
<name code="yyy"
codeSystem="2.16.840.1.113883.6.96"/>
<value xsi:type="CD" code="E"
codeSystem="1.2.276.0.76.5.yyy"/>
</qualifier>
</value>
<!-- B-Symptome -->
<entryRelationship typeCode="COMP">
<observation classCode="OBS" moodCode="EVN">
<code code="AnnArborBSymptoms"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="B"
codeSystem="1.2.276.0.76.5.xxx"/>
</observation>
</entryRelationship>
<!-- Organbefall -->
<entryRelationship typeCode="COMP">
<observation classCode="OBS" moodCode="EVN">
<code code="AnnArborExtranodal"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="S" codeSystem="1.2.276.0.76.5.yyy">
<qualifier>
<name code="127230005"
codeSystem="2.16.840.1.113883.6.96"/>
<value xsi:type="CD" code="true"
codeSystem="1.2.276.0.76.3.1.131.1.5.61"/>
</qualifier>
...
</value>
</observation>
</entryRelationship>
</observation>
Vokabular
Snomed CT Codes:
- Ann Arbor lymphoma staging system: 254372002
- Ann Arbor Hodgkin's disease tumour staging system: 254373007
- Ann Arbor non-Hodgkin lymphoma tumor staging system: 254374001
Ann-Arbor-Stadium
| Code | Description |
|---|---|
| I | cancer is located in a single region, usually one lymph node and the surrounding area |
| II | cancer is located in two separate regions, an affected lymph node or organ and a second affected area, and that both affected areas are confined to one side of the diaphragm |
| II2 | |
| II1 | |
| III | cancer has spread to both sides of the diaphragm, including one organ or area near the lymph nodes or the spleen |
| IV | diffuse or disseminated involve¬ment of one or more extra¬lymphatic organs |
Tabelle 33: Ann-Arbor-Stadium (OID 1.2.276.0.76.5.405)
Ann-Arbor Stadium qualifier
| Code | Beschreibung |
|---|---|
| A | Ohne Allgemeinsymptome |
| B | Mit Allgemeinsymptome |
| E | extranodaler Befall |
| X | bulky disease (>10 cm durchmessende Konglomerate) |
| S | Milzbeteiligung |
Tabelle: Ann-Arbor Stadium Qualifier
Ann-Arbor Organbefall
| Code | Beschreibung |
|---|---|
| E | Extralymphatisches Organ befallen |
| S | Milz befallen |
| X |
Tabelle: Ann-Arbor Organbefall (als qualifier, OID 1.2.276.0.76.5.yyy)
Ann-Arbor B.Symptome
|
|
tbd: hier fehlt die Liste.... |
Organbefall
Hier werden noch weitere Organe aufgelistet. Damit haben wir einen Value Set mit zwei unterschiedlichen Codelisten:
| Code | Beschreibung |
|---|---|
| Blut | |
| Milz | |
| Knochen | |
| Knochenmark | |
| Haut |
Tabelle: Organbefall (als dedizierte Organangabe, OID 1.2.276.0.76.5.yyy)
|
|
Diese Information sollte als Code im value enthalten sein. Damit sagt der Code aus, dass es sich hier um ein befallenes Organ handelt. Besser wäre hierfür die Nutzung des targetSiteCode in der primären Observation! |
Hier sollte eine Kodierung in Snomed CT erfolgen.
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Entry: R-Klassifikation
| Template-Metadaten | |
| Template-Typ | Entry |
| Template ID | 1.2.276.0.76.3.1.131.1.10.3.7 |
| generischeres Template | |
| genutztes Template | |
| nutzende Templates | |
| abgeleitete Templates | |
| Schwester-Templates | |
| generelle Beschreibung | R-Klassifikation (Residualtumor) |
| allg. Erläuterung | |
| Verhältnis zu IHE | neu |
| Ballotierungsstatus | in Arbeit |
| Erweiterbarkeit | geschlossen |
Beschreibung
Die R-Klassifikation beschreibt den nach therapeutischen Maßnahmen (insbesondere Operationen) im Körper verbliebenen Residualtumor. Sie bezieht sich aber nicht nur auf Operationen, in deren Kontext sie allerdings die praktisch größte Bedeutung hat, sondern berücksichtigt teilweise auch klinische Angaben.
Die sogenannte globale R-Klassifikation betrachtet die gesamte Erkrankung, also sowohl das lokale Geschehen als auch etwaige Fernmetastasen. Diese Klassifikation führt der betreuende Kliniker unter Berücksichtigung etwaiger Befunde der Pathologen durch.
Die lokale Radikalität einer Operation wird gleichartig codiert, betrachtet aber nur das Resektat und wird durch den Pathologen bestimmt.
Hinweis: Es existieren zwei semantisch widersprüchliche Definitionen für die Codierung R2a/R2b bzw. R2a/R2b/R2c. Hier findet die erstgenannte Verwendung (vgl. Value Set). Die letztgenannte Definition, entstammend dem TNM-Supplement (4. Auflage), lässt sich wie folgt übersetzen:
- R2a: R2 mit Qualifier 'Vorhandensein Residualtumor' = "L"
- R2b: R2 mit Qualifier 'Vorhandensein Residualtumor' = "M"
- R2c: R2 mit Qualifier 'Vorhandensein Residualtumor' = "B"
|
|
Die vorgenannte Darstellungsweise zur Auflösung der widersprüchlichen Definition befindet sich noch in Diskussion und ist daher als vorläufig zu betrachten. |
Modell
|
|
TODO: R-Status ist Teil von TNM. Abgleich mit TNM-Template aus APSR 2.0 erforderlich! |
Abbildung: R-Klassifikation
Attribute
| Lvl | RIM | Name | DT | Kard | Conf | Beschreibung | ||
|---|---|---|---|---|---|---|---|---|
| 3 | act | observation | 1..1 | F | @classCode="OBS", @moodCode="EVN" | |||
| 4 | act | templateId | II | 1..1 | F | @root="1.2.276.0.76.3.1.131.1.10.3.7" | ||
| 4 | act | id | II | 0..1 | O | ID der diagnostischen Angabe | ||
| 5 | act | @root | 1..1 | O | OID des sendenden System, um diagnostische Angaben eindeutig zu identifizieren | |||
| 5 | act | @extension | 1..1 | O | ID der diagnostischen Angabe im sendenden System | |||
| 4 | act | code | CD CWE | 1..1 | F | @code="tnmR", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" | ||
| 4 | act | text | ED | 1..1 | R | textliche Beschreibung des codierten Inhalts der Observation; i.d.R. vom sendenden System automatisch generiert | ||
| 4 | act | effectiveTime | TS | 0..1 | O | Das Datum, an dem die Diagnose (fest)gestellt wurde | ||
| 4 | act | value | CD | 1..1 | R | Value Set 1.2.276.0.76.11.4 | ||
| 5 | act | qualifier | CR | 0..1 | O | Gültigkeitsbereich.
Wird dieser Qualifier nicht übermittelt, so gilt implizit der Code "G" (gültig für die Gesamtsituation der Erkrankung). | ||
| 6 | act | name | CV | 1..1 | F | @code="tnmRDomain", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" | ||
| 6 | act | value | CV | 1..1 | R | TODO: Value Set OID definieren: fix: @codeSystem="1.2.276.0.76.5.???"
| ||
| 5 | act | qualifier | CR | 0..1 | O | Existenz eines Residualtumors.
Dieser Qualifier darf nur bei globaler Gültigkeit der R-Klassifikation übermittelt werden | ||
| 6 | act | name | CV | 1..1 | F | @code="tnmRLocation", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" | ||
| 6 | act | value | CV | 1..1 | R | TODO: Value Set OID vergeben. fix: @codeSystem="1.2.276.0.76.5.???"
Die explizite Angabe für unbekannt wird durch @nullFlavor="UNK" ausgedrückt.
| ||
| 4 | rel | entryRelationship | 0..n | F | @typeCode="SPRT" | |||
| 5 | act | observation | 1..1 | R | Zusatzinformationen zur R-Klassifikation Jeder Zusatzinformation (Code) darf maximal ein Mal übermittelt werden. Wird die entsprechende Angabe nicht übermittelt, gilt implizit der Wert "false" | |||
| 6 | act | code | CD CWE | 1..1 | R | vgl. Tabelle "Zusatzinformationen zur R-Klassifikation". | ||
| 6 | act | value | BL | 1..1 | R |
Verwendete Terminologie
Zusatzinformationen
| Code | Suffix-Symbol | Bedeutung |
|---|---|---|
| residualIs | (is) | invasives Karzinom komplett entfernt, aber in-situ Komponente am Schnittrand |
| residualCy+ | (cy+) | bei Spültechniken zytologisch entdeckte Tumorzellen |
| residualUn | (un) | unsichere R0 Resektion (TNM-Supplement, 4. Auflage, Seite 25) |
| residualGt1mm | >1mm | R0, Abstand Resektionsrand > 1mm, entspricht CRM negative (TNM-Supplement, 4. Auflage, Seite 25(??) |
| residualLt1mm | <1mm | R0, Abstand Resektionsrand < 1mm, entspricht CRM positive (TNM-Supplement, 4. Auflage, Seite 25(??) |
| residualDir | -dir | Tumor direkt am Resektionsrand durchschnitten |
Tabelle: Zusatzinformationen zur R-Klassifikation
Value Set (OID TODO)
Codesystem: 1.2.276.0.76.3.1.131.1.5.1
Residualtumor
| Code | Description | Bedeutung |
|---|---|---|
| R0 | No residual tumour | kein Residualtumor |
| R0a | kein Residualtumor, Tumormarker normal | |
| R0b | kein Residualtumor, Tumormarker erhöht | |
| R1 | Microscopic residual tumour | nur mikroskopisch nachweisbarer Residualtumor |
| R2 | Macroscopic residual tumour | makroskopischer Residualtumor |
| R2a | makroskopischer Residualtumor, mikroskopisch nicht bestätigt | |
| R2b | makroskopischer Residualtumor, mikroskopisch bestätigt | |
| RX | Presence of residual tumour cannot be assessed | Unbekannt |
Tabelle: Residualtumor-Codes
Value Set (OID 1.2.276.0.76.11.4)
UICC 5. Auflage (OID 2.16.840.1.113883.15.8)
UICC 6. Auflage (OID 2.16.840.1.113883.15.7)
UICC 7. Auflage (OID 2.16.840.1.113883.15.6)
Gültigkeitsbereich der Beurteilung des Residualtumors
Auf welchen Bereich bezieht sich die Beurteilung?
| Code | Bedeutung |
|---|---|
| L | Lokoregionärer Tumor |
| M | Fernmetastasen |
| G | Gesamtsituation der Erkrankung |
Tabelle: Gültigkeitsbereich-Codes
Value Set (OID TODO)
Codesystem: "1.2.276.0.76.5.???" (TODO: vorläufig, die OID .414 wurde doppelt vergeben!)
Vorhandensein Residualtumor
Wo ist der Residualtumor lokalisiert?
| Code | Bedeutung |
|---|---|
| L | Lokoregionärer Tumor |
| M | Fernmetastasen |
| B | beides |
Tabelle: Gültigkeitsbereich-Codes
Value Set (OID TODO)
Codesystem: "1.2.276.0.76.5.???" (TODO: vorläufig, die OID .415 wurde doppelt vergeben!)
Beispiel
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.276.0.76.3.1.131.1.10.3.7"/>
<id extension="1234041"
root="1.2.276.0.76.3.1.131.1.4.3.9999.9999.999919"/>
<code code="tnmR" codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
gesamt R1, Fernmetastasen vorhanden
</text>
<effectiveTime value="20110326"/>
<value xsi:type="CD" code="R1"
codeSystem="2.16.840.1.113883.15.6">
<qualifier>
<name code="tnmRDomain"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="G" codeSystem="1.2.276.0.76.5.414"/>
</qualifier>
<qualifier>
<name code="tnmRLocation"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="L"
codeSystem="1.2.276.0.76.5.415"/>
</qualifier>
</value>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="residualIs"
codeSystem="1.2.276.0.76.3.1.131.1.5.1 "/>
<value xsi:type="BL" value="true"/>
</observation>
</entryRelationship>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="residualGt1mm"
codeSystem="1.2.276.0.76.3.1.131.1.5.1 "/>
<value xsi:type="BL" value="true"/>
</observation>
</entryRelationship>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="residualCy+"
codeSystem="1.2.276.0.76.3.1.131.1.5.1 "/>
<value xsi:type="BL" value="false"/>
</observation>
</entryRelationship>
</observation>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Entry: TNM-Klassifikation
| Template-Metadaten | |
| Template-Typ | Entry |
| Template ID | 1.2.276.0.76.3.1.131.1.10.3.5 ????? |
| generischeres Template | <Verweis auf das Template> |
| genutztes Templates | R-Klassifikation |
| abgeleitete Templates | cdapath:TNM-Klassifikation-Entry (Template) |
| Generelle Beschreibung | Hiermit wird auf Entry-Level die Übermittlung von Tumordiagnosedaten (UICC TNM-Klassifikation) definiert. |
| allg. Erläuterung | |
| Verhältnis zu IHE | neu: Abstimmung mit QRPH und AP |
| Ballotierungsstatus | in Arbeit |
| Erweiterbarkeit | geschlossen |
Darstellung der TNM-Klassifikation in HL7
Die Darstellung der TNM-Klassifikation in HL7 V3 erfolgt nach folgendem Schema. Es gibt eine zentrale Observation-Klasse, über welche das UICC-Stadium (als value) und/oder die Tumorformel (als Unterlement originalText dieses values) angegeben werden. Ist das UICC-Stadium nicht bekannt (z.B. weil bei einer pathologischen Befundung keine Angaben zu Metastasen existieren), wird im value der Haupt-Observation ein nullFlavor="NA" übermittelt. Die Tumorformel kann dennoch im originalText übermittelt werden.
Einzelangaben des werden ebenfalls in Observation-Klassen eingebunden, die über eine ActRelationShip-Klasse mit dem code SPRT mit der zentralen Observation-Klasse verbunden werden.
Diese Angaben sind (in Fachterminologie):
| TNM-Symbol | Bedeutung |
|---|---|
| y | nach neoadjuvanter Therapie |
| r | Beurteilung eines Rezidivs |
| a | Beurteilung erstmals aus Anlass einer Autopsie |
| T | (Primär-)Tumor |
| N | Nodus = Lymphknoten |
| M | Metastasen |
| S | Serumtumormarker (für bestimmte Tumorentitäten) |
| L | Lymphgefäßinvasion |
| V | Veneninvasion |
| Pn | Perineuralinvasion |
| G | Histopathologisches Grading |
| R | Residualtumor |
Weitere Angaben werden als qualifier für die jeweiligen values angegeben:
| TNM-Symbol | Bedeutung | gültig für |
|---|---|---|
| c/u/ct/mr/p | klinisch (Ultraschall, CT, MRT)/pathologisch festgestellt | T, N, M |
| C | Certainty Factor | T, N, M |
| m | multipler Tumor (unquantifiziert) bzw. Angabe der Anzahl der Tumore |
T |
| sn | Sentinel Nodes | N |
| m1, m2, m3 | auf Mukosa beschränkt, 1., 2. 3. Drittel | T, gilt nur für gastrointestinale Karzinome |
| sm1, sm2, sm3 | auf Submukosa beschränkt, 1., 2. 3. Drittel | T, gilt nur für gastrointestinale Karzinome |
| (Lokalisationscode) | Lokalisation der Metastasen | M |
Weiterhin existieren Zusatzangaben zu bestimmten Observations, die wiederum über eine ActRelationship als Observation angegeben werden:
| TNM-Symbol | Bedeutung | gültig für |
|---|---|---|
| (kein) | Multiplizität (Anzahl der Tumorherde, numerisch) | T |
| i- | immunhistochemisch nachgewiesene isolierte Tumorzellen (ITC) | N, M |
| i+ | immunhistochemisch ausgeschlossene isolierte Tumorzellen (ITC) | N, M |
| mol+ | molekularpathologisch nachgewiesene isolierte Tumorzellen (ITC) | N, M |
| mol- | molekularpathologisch ausgeschlossene isolierte Tumorzellen (ITC) | N, M |
| (kein) | Anzahl entfernter und befallener Lymphknoten wird angegeben als RTO aus befallenen zu entfernten Lymphknoten |
N |
Isolierte Tumorzellen sind einzeln oder in Clustern von bis zu 200 Zellen oder bis zu 200 Mikrometern Clusterdurchmesser vorkommende Tumorzellen, die bis auf das Melanom mit N0 kodiert werden. Mikrometastasen in Lymphknoten sind größer als 200 Mikrometer und kleiner als 2 mm, in aller Regel nur durch mikroskopische Untersuchung sichtbar.
Abbildung: Darstellung der TNM-Klassifikation
|
|
TODO: Abgleich mit TNM-Template aus APSR 2.0 erforderlich! |
Anmerkung: Die Begriffe "davor" bzw. "dahinter" in obiger Grafik geben an, ob dieser Qualifier in der Darstellung als Tumorformel vor oder hinter der entsprechenden Kategorie (T, N oder M) aufgeführt wird. In der XML-Darstellung spielt dies jedoch keine Rolle.
Zum einfacheren Verständnis kann dies auch in einer hierarchischen Form dargestellt werden. Die jeweils anwendbaren Qualifier sind dabei in Klammern angegeben:
+- UICC-Stadium, Tumorformel +- y +- r +- a +- T-Kategorie (c/u/ct/mr/p, C, m, m1/m2/m3, sm1/sm2/sm3) +- N-Kategorie (c/u/ct/mr/p, C, sn) | +- (i+/i-: ITC durch Immunhistochemie nur für p) | +- (mol+/mol-: ITC durch Molekularpathologie nur für p) | +- Anzahl der untersuchten und positiv bewerteten Knoten +- M-Kategorie (c/u/ct/mr/p, C, Lokalisationscodes) | +- (i+/i-: ITC durch Immunhistochemie) | +- (mol+/mol-: ITC durch Molekularpathologie) +- S (Serumtumormarker) +- L (Lymphgefäßinvasion) +- V (Veneninvasion) +- Pn (Perineuralscheideninvasion) +- G (histopathologisches Grading) +- R (Residualtumor)
Abbildung: Baumdarstellung der TNM-Klassifikation
Erläuterung
| Lvl | RIM | Name | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|
TNM-Klassifikation UICC-Stadien) | ||||||
| 3 | act | observation | 1..1 | F | @classCode="OBS", @moodCode="EVN" | |
| 4 | act | templateId | II | 1..1 | F | @root="1.2.276.0.76.3.1.131.1.10.3.5" |
| 4 | act | id | II | 0..1 | optional | ID der diagnostischen Angabe |
| 5 | act | @root | 1..1 | optional | OID des sendenden Systems, um diagnostische Angaben eindeutig zu identifizieren | |
| 5 | act | @extension | 1..1 | optional | ID der diagnostischen Angabe im sendenden System | |
| 4 | act | code | CD CWE | 1..1 | F | @code="tnmStage", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 4 | act | text | ED | 1..1 | required | textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert |
| 4 | act | effectiveTime | TS | 0..1 | optional | Das Datum, an dem die Diagnose gestellt wurde |
| 4 | act | value | CD | 1..1 | R | Code für das UICC-Stadium Value Set: 1.2.276.0.76.11.5 |
| 4 | rel | entryRelationship | 0..1 | optional | fix: @typeCode="SPRT" | |
TNM y-Symbol (Präfix) | ||||||
| 5 | act | observation | 1..1 | F | Stadium nach neoadjuvanter Therapie @classCode="OBS", @moodCode="EVN" | |
| 6 | act | code | CD CWE | 1..1 | F | @code="tnmYSymbol", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 6 | act | text | ED | 1..1 | required | textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert |
| 6 | act | value | BL | 1..1 | required | |
| 4 | rel | entryRelationship | 0..1 | optional | fix: @typeCode="SPRT" | |
TNM r-Symbol (Präfix) | ||||||
| 5 | act | observation | 1..1 | F | Stadium eines Rezidive nach tumorfreiem Intervall classCode="OBS", @moodCode="EVN" | |
| 6 | act | code | CD CWE | 1..1 | required | fix: @code="tnmRSymbol", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 6 | act | text | ED | 1..1 | R | textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert |
| 6 | act | value | BL | 1..1 | R | |
| 4 | rel | entryRelationship | 0..1 | F | @typeCode="SPRT" | |
| 5 | TNM a-Symbol (Präfix) | |||||
| 5 | act | observation | 1..1 | F | Beurteilung erstmals aus Anlass einer Autopsie classCode="OBS", @moodCode="EVN" | |
| 6 | act | code | CD CWE | 1..1 | required | fix: @code="tnmASymbol", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 6 | act | text | ED | 1..1 | R | textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert |
| 6 | act | value | BL | 1..1 | R | |
| 4 | rel | entryRelationship | 0..1 | F | @typeCode="SPRT" | |
| 5 | T: Tumor | |||||
| 5 | act | observation | 1..1 | F | @classCode="OBS", @moodCode="EVN" | |
| 6 | act | code | CD CWE | 1..1 | F | @code="tnmT", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 6 | act | text | ED | 1..1 | required | textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert |
| 6 | act | value | CD | 1..1 | required | Value Set: 1.2.276.0.76.11.1 |
| 7 | act | qualifier | CR | 0..1 | optional | Art der Sicherung (qualifier)(klinisch (Ultraschall, CT, MRT), pathologisch) |
| 8 | act | name | CV | 1..1 | F | @code="tnmCp", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | CV | 1..1 | R | Value Set: 1.2.276.0.76.11.9 |
| 7 | act | qualifier | CR | 0..1 | O | C-Faktor (Certainty) (qualifier) |
| 8 | act | name | CV | 1..1 | F | @code="tnmCFactor", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | CV | 1..1 | required | TODO: Value Set OID; fix: @codeSystem="1.2.276.0.76.5.341" |
| 7 | act | qualifier | CR | 0..1 | O | m-Symbol: Multiplizität (multiple tumor) (qualifier) |
| 8 | act | name | CV | 1..1 | F | @code="tnmMSymbol", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | BL | 1..1 | R | |
| 7 | act | qualifier | CR | 0..1 | required | Multiplizität (numerisch): Anzahl der Tumorherde (qualifier) |
| 8 | act | code | CV | 1..1 | required | fix: @code="tnmMultiplicity", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | INT.NONNEG | 1..1 | required | |
| 7 | act | qualifier | CR | 0..1 | O | Mucosainfiltration (qualifier) |
| 8 | act | name | CV | 1..1 | F | @code="tnmMucosaSymbol", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | CV | 1..1 | required | TODO: Value Set OID (Code "m1", "m2" oder "m3"); fix: @codeSystem="2.16.840.1.113883.15.6" |
| 7 | act | qualifier | CR | 0..1 | O | Submucosainfiltration (qualifier) |
| 8 | act | name | CV | 1..1 | F | @code="tnmSubmucosaSymbol", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | CV | 1..1 | required | TODO: Value Set OID (Code "sm1", "sm2" oder "sm3"); fix: @codeSystem="2.16.840.1.113883.15.6" |
| 4 | rel | entryRelationship | 0..1 | optional | fix: @typeCode="SPRT" | |
N: Nodalstatus | ||||||
| 5 | act | observation | 1..1 | F | @classCode="OBS", @moodCode="EVN" | |
| 6 | act | code | CD CWE | 1..1 | required | fix: @code="tnmN", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 6 | act | text | ED | 1..1 | required | textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert |
| 6 | act | value | CD | 1..1 | required | Value Set: 1.2.276.0.76.11.2 |
| 7 | act | qualifier | CR | 0..1 | optional | Art der Sicherung (qualifier)Art der Sicherung (klinisch (Ultraschall, CT, MRT), pathologisch) |
| 8 | act | name | CV | 1..1 | required | fix: @code="tnmCp", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | CV | 1..1 | required | Value Set: 1.2.276.0.76.11.9 |
| 7 | act | qualifier | CR | 0..1 | optional | C-Faktor (Certainty) |
| 8 | act | name | CV | 1..1 | required | fix: @code="tnmCFactor", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | CV | 1..1 | required | TODO: Value Set OID; fix: @codeSystem="1.2.276.0.76.5.341" |
| 7 | act | qualifier | CR | 0..1 | optional | sn-Symbol (Sentinel Nodes) |
| 8 | act | name | CV | 1..1 | required | fix: @code="tnmSn", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | CV | 1..1 | required | TODO: Value Set OID; fix: @codeSystem="2.16.840.1.113883.15.6" |
| 7 | act | qualifier | 1..1 | required | i (Suffix): ITC durch Immunhistochemie (qualifier) | |
| 8 | act | name | CD CWE | 1..1 | F | @code="tnmI", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | CD | 1..1 | required | TODO: Value Set OID (Code "i+" oder "i-"); fix: @codeSystem="2.16.840.1.113883.15.6" |
| 7 | act | qualifier | 1..1 | required | mol (Suffix): ITC durch Molekularpathologiefix: @classCode="OBS", @moodCode="EVN" | |
| 8 | act | name | CD CWE | 1..1 | required | fix: @code="tnmMol", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | CD | 1..1 | required | TODO: Value Set OID (Code "mol+" oder "mol-"); fix: @codeSystem="2.16.840.1.113883.15.6" |
| 6 | rel | entryRelationship | 0..1 | optional | fix: @typeCode="SPRT" | |
| 7 | act | observation | 1..1 | required | Anzahl LymphknotenDiese Observation kann auch standalone bzw. in anderem Kontext verwendet werden. | |
| 8 | act | code | CD CWE | 1..1 | required | für die Angabe von Lymphknoten allgemein fix: @code="21894-1", @codeSystem="2.16.840.1.113883.6.1" für die Angabe von Sentinel Nodes fix: @code="SentinelNodes", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | RTO_PQ_PQ | 1..1 | required | |
| 9 | act | numerator | PQ | 1..1 | required | @value: Anzahl der befallenen Lymphknoten
fix: @unit="1" |
| 9 | act | denominator | PQ | 1..1 | required | @value: Anzahl der entfernten (untersuchten) Lymphknoten
fix: @unit="1" |
| 4 | rel | entryRelationship | 0..1 | optional | fix: @typeCode="SPRT" | |
M: Metastasen | ||||||
| 5 | act | observation | 1..1 | F | @classCode="OBS", @moodCode="EVN" | |
| 6 | act | code | CD CWE | 1..1 | required | fix: @code="tnmM", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 6 | act | text | ED | 1..1 | required | textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert |
| 6 | act | value | CD | 1..1 | required | Value Set: 1.2.276.0.76.11.3 |
| 7 | act | qualifier | CR | 0..1 | optional | Art der Sicherung (qualifier)Art der Sicherung (klinisch (Ultraschall, CT, MRT), pathologisch) |
| 8 | act | name | CV | 1..1 | required | fix: @code="tnmCp", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | CV | 1..1 | required | Value Set: 1.2.276.0.76.11.9 |
| 7 | act | qualifier | CR | 0..1 | optional | C-Faktor (Certainty) |
| 8 | act | name | CV | 1..1 | required | fix: @code="tnmCFactor", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | CV | 1..1 | required | TODO: Value Set OID; fix: @codeSystem="1.2.276.0.76.5.341" |
| 7 | act | qualifier | CR | 0..* | optional | Lokalisationscodes |
| 8 | act | name | CV | 1..1 | required | fix: @code="21920-4", @codeSystem="2.16.840.1.113883.6.1" |
| 8 | act | value | CV | 1..1 | required | TODO: Value Set OID; fix: @codeSystem="1.2.276.0.76.5.401" |
| 7 | act | qualifier | CR | 1..1 | required | i (Suffix): ITC durch Immunhistochemie (qualifier)fix: @classCode="OBS", @moodCode="EVN" |
| 8 | act | name | CD CWE | 1..1 | required | fix: @code="tnmI", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | CD | 1..1 | required | TODO: Value Set OID (Code "i+" oder "i-"); fix: @codeSystem="2.16.840.1.113883.15.6" |
| 7 | act | qualifier | CR | 1..1 | required | mol (Suffix): RT-PCR (ITC durch Molekularpathologie) (qualifier)fix: @classCode="OBS", @moodCode="EVN" |
| 8 | act | name | CD CWE | 1..1 | required | fix: @code="tnmMol", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | CD | 1..1 | required | TODO: Value Set OID (Code "mol+" oder "mol-"); fix: @codeSystem="2.16.840.1.113883.15.6" |
| 4 | rel | entryRelationship | 0..1 | optional | fix: @typeCode="SPRT" | |
S: Serumtumormarker | ||||||
| 5 | act | observation | 1..1 | F | @classCode="OBS", @moodCode="EVN" | |
| 6 | act | code | CD CWE | 1..1 | required | fix: @code="tnmS", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 6 | act | text | ED | 1..1 | required | textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert |
| 6 | act | value | CD | 1..1 | required | TODO Value Set OID |
| 4 | rel | entryRelationship | 0..1 | optional | fix: @typeCode="SPRT" | |
L: Lymphgefäßinvasion | ||||||
| 5 | act | observation | 1..1 | F | @classCode="OBS", @moodCode="EVN" | |
| 6 | act | code | CD CWE | 1..1 | required | fix: @code="tnmL", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 6 | act | text | ED | 1..1 | required | textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert |
| 6 | act | value | CD | 1..1 | required | Value Set: 1.2.276.0.76.11.7 |
| 4 | rel | entryRelationship | 0..1 | optional | fix: @typeCode="SPRT" | |
V: Veneninvasion | ||||||
| 5 | act | observation | 1..1 | F | @classCode="OBS", @moodCode="EVN" | |
| 6 | act | code | CD CWE | 1..1 | required | fix: @code="tnmV", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 6 | act | text | ED | 1..1 | required | textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert |
| 6 | act | value | CD | 1..1 | required | Value Set: 1.2.276.0.76.11.6 |
| 4 | rel | entryRelationship | 0..1 | optional | fix: @typeCode="SPRT" | |
Pn: Perineuralinvasion | ||||||
| 5 | act | observation | 1..1 | F | @classCode="OBS", @moodCode="EVN" | |
| 6 | act | code | CD CWE | 1..1 | required | fix: @code="tnmPn", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 6 | act | text | ED | 1..1 | required | textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert |
| 6 | act | value | CD | 1..1 | required | Value Set: 1.2.276.0.76.11.8 |
| 4 | rel | entryRelationship | 0..1 | optional | fix: @typeCode="SPRT" | |
G: histopathologisches Grading | ||||||
| 5 | act | observation | 1..1 | required | siehe Template Grading | |
| 4 | rel | entryRelationship | 0..1 | optional | fix: @typeCode="SPRT" | |
R: Residualtumor | ||||||
| 5 | act | observation | 1..1 | required | siehe Template |
Beispiel
Das folgende Beispiel stellt das Maximum der möglichen Angaben dar, wohl wissend, dass dies aus medizinisch-fachlicher Sicht nicht korrekt ist.
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.276.0.76.3.1.131.1.10.3.5"/>
<id extension="tnm12345"
root="1.2.276.0.76.3.1.131.1.4.3.9999.9999.999909"/>
<code code="tnmStage"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
Tumorklassifikation vom 26.03.2011: ayr UICC IV
</text>
<effectiveTime value="20110326"/>
<value xsi:type="CD" code="IV"
codeSystem="2.16.840.1.113883.15.6" />
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="tnmYSymbol"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
y
</text>
<value xsi:type="BL" value="true" />
</observation>
</entryRelationship>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="tnmRSymbol"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
r
</text>
<value xsi:type="BL" value="true" />
</observation>
</entryRelationship>
<!-- T value -->
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="tnmT"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
pT3am(4)(m1)(sm2)C4
</text>
<value xsi:type="CD" code="T3a"
codeSystem="2.16.840.1.113883.15.6">
<qualifier>
<name code="tnmCp"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="p"
codeSystem="2.16.840.1.113883.15.6"/>
</qualifier>
<qualifier>
<name code="tnmCFactor"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="C4"
codeSystem="1.2.276.0.76.5.341"/>
</qualifier>
<qualifier>
<name code="tnmMSymbol"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="m"
codeSystem="2.16.840.1.113883.15.6"/>
</qualifier>
<qualifier>
<code code="tnmMultiplicity"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="INT.NONNEG" value="4" />
</qualifier>
<qualifier>
<name code="tnmMucosaSymbol"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="m1"
codeSystem="2.16.840.1.113883.15.6"/>
</qualifier>
<qualifier>
<name code="tnmSubmucosaSymbol"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="sm2"
codeSystem="2.16.840.1.113883.15.6"/>
</qualifier>
</value>
</observation>
</entryRelationship>
<!-- N value -->
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="tnmN"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
pN3(sn)(i+)(mol+)C5
</text>
<value xsi:type="CD" code="N3"
codeSystem="2.16.840.1.113883.15.6">
<qualifier>
<name code="tnmCp"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="p"
codeSystem="1.2.276.0.76.5.341"/>
</qualifier>
<qualifier>
<name code="tnmCFactor"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="C5"
codeSystem="1.2.276.0.76.5.341"/>
</qualifier>
<qualifier>
<name code="tnmSn"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="sn"
codeSystem="2.16.840.1.113883.15.6"/>
</qualifier>
<qualifier>
<code code="tnmI"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="BL" code="true"/>
</qualifier>
<qualifier>
<code code="tnmMol"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="BL" value="true"/>
</qualifier>
</value>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="21894-1" codeSystem="2.16.840.1.113883.6.1"/>
<value xsi:type="RTO_PQ_PQ">
<numerator value="2" unit="1"/>
<denominator value="12" unit="1"/>
</value>
</observation>
</entryRelationship>
</observation>
</entryRelationship>
<!-- M value -->
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="tnmM"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
cM1(sn)(i+)(mol+)C2
</text>
<value xsi:type="CD" code="M1"
codeSystem="2.16.840.1.113883.15.6">
<qualifier>
<name code="tnmCp"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="c"
codeSystem="1.2.276.0.76.5.341"/>
</qualifier>
<qualifier>
<name code="tnmCFactor"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="C2"
codeSystem="1.2.276.0.76.5.341"/>
</qualifier>
<qualifier>
<name code="tnmI"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="BL" value="true" />
</qualifier>
<qualifier>
<code code="tnmMol"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="BL" value="true" />
</qualifier>
</value>
</observation>
</entryRelationship>
<!-- further attributes -->
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="tnmS"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
S2
</text>
<value xsi:type="CD" code="S2"
codeSystem="2.16.840.1.113883.15.6"/>
</observation>
</entryRelationship>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="tnmL"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
L1
</text>
<value xsi:type="CD" code="L1"
codeSystem="2.16.840.1.113883.15.6"/>
</observation>
</entryRelationship>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="tnmV"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
V1
</text>
<value xsi:type="CD" code="V1"
codeSystem="2.16.840.1.113883.15.6"/>
</observation>
</entryRelationship>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="tnmPn"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
PnX
</text>
<value xsi:type="CD" code="PnX"
codeSystem="2.16.840.1.113883.15.6"/>
</observation>
</entryRelationship>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="336"
codeSystem="2.16.840.1.113883.3.7.1.0"/>
<value xsi:type="CD" code="2"
codeSystem="1.2.276.0.76.5.336"/>
</observation>
</entryRelationship>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.276.0.76.3.1.131.1.10.3.7"/>
<id extension="1234041"
root="1.2.276.0.76.3.1.131.1.4.3.9999.9999.999919"/>
<code code="tnmR" codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
gesamt R1, Fernmetastasen vorhanden
</text>
<effectiveTime value="20110326"/>
<value xsi:type="CD" code="R1"
codeSystem="2.16.840.1.113883.15.6">
<qualifier>
<name code="tnmRDomain"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="G" codeSystem="1.2.276.0.76.5.414"/>
</qualifier>
<qualifier>
<name code="tnmRLocation"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="L"
codeSystem="1.2.276.0.76.5.415"/>
</qualifier>
</value>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="residualIs"
codeSystem="1.2.276.0.76.3.1.131.1.5.1 "/>
<value xsi:type="BL" value="true"/>
</observation>
</entryRelationship>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="residualGt1mm"
codeSystem="1.2.276.0.76.3.1.131.1.5.1 "/>
<value xsi:type="BL" value="true"/>
</observation>
</entryRelationship>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="residualCy+"
codeSystem="1.2.276.0.76.3.1.131.1.5.1 "/>
<value xsi:type="BL" value="false"/>
</observation>
</entryRelationship>
</observation>
verwendete Terminologie
|
|
Die TNM-Klassifikation gemäß UICC wird insgesamt als Codeystem aufgefasst. Somit haben die verschiedenen Tabellen die gleiche OID, die dann in der Übermittlung anzugeben ist. Die einfachste Möglichkeit, damit umzugehen, ist die Definition eines Value Sets mit Festlegung der jeweils möglichen Werte. Darüber hinaus werden für diese Tabellen dann angegeben, welche Werte daraus aus den entsprechenden UICC Ausgaben (Auflagen) entstammen und somit über das jeweilige konkrete Kodiersystem angegeben werden können. |
Stadiengruppierung nach UICC
| UICC | |||||
|---|---|---|---|---|---|
| Code | Beschreibung | Bedeutung | 5. | 6. | 7. |
| okk | TX, N0, M0 | X | X | X | |
| 0 | Carcinoma in situ | Tis, N0, M0 | X | X | X |
| 0a | X | X | X | ||
| 0is | X | X | X | ||
| I | X | X | |||
| IA | T1, N0, M0 | X | X | X | |
| IA1 | X | X | X | ||
| IA2 | X | X | X | ||
| IB | T2, N0, M0 | X | X | X | |
| IB1 | X | X | X | ||
| IB2 | X | X | X | ||
| IC | X | X | X | ||
| II | X | X | X | ||
| IIA | T1, N1, M0 | X | X | X | |
| IIA1 | X | ||||
| IIA2 | X | ||||
| IIB | T2, N1, M0 | X | X | X | |
| IIC | T3, N0, M0 | X | X | X | |
| III | X | X | X | ||
| IIIA | T1, N2, M0 | X | X | X | |
| T2, N2, M0 | |||||
| T3, N1,2, M0 | |||||
| IIIB | T4, jedes N, M0 | X | X | X | |
| jedes T, N3, M0 | |||||
| IIIC | X | X | X | ||
| IS | X | X | X | ||
| IV | jedes T, jedes N, M1 | X | X | X | |
| IVA | X | X | X | ||
| IVB | X | X | X | ||
| IVC | X | X | X |
Tabelle: Codes für Stadiengruppierung nach UICC16
Value Set (OID 1.2.276.0.76.11.5)
UICC 5. Auflage (OID 2.16.840.1.113883.15.8)
UICC 6. Auflage (OID 2.16.840.1.113883.15.7)
UICC 7. Auflage (OID 2.16.840.1.113883.15.6)
Ausdehnung des Primärtumors (T)
Bekannte T-Kategorien (ohne Darstellung möglicher Zusätze). Die Beschreibung variiert je nach Entität. Der Code soll nur in Zusammenhang mit der Art der Tumorerkrankung übermittelt werden, da die Kodes mit unterschiedlichen Bedeutungen belegt sind (z.B. T1 entspricht 2-3 cm oder 2-4 cm).
| UICC | ||||||
|---|---|---|---|---|---|---|
| Code | Description | Bedeutung | 5. | 6. | 7. | SCT |
| Ta | Noninvasive papillary urothelial carcinoma | Nicht invasiv | X | X | X | X |
| Tis | Carcinoma in situ | Nicht invasiv | X | X | X | X |
| Tis(pu) | in situ urothelial carcinoma of prostatic urethra | Nicht invasiv | X | X | ||
| Tis(pd) | in situ urothelial carcinoma of prostatic gland ducts | Nicht invasiv | X | X | ||
| T0 | No evidence of primary tumour | Kein Primärtumor nach¬weisbar | X | X | X | |
| T1 | X | X | X | X | ||
| T1mic | Microinvasion | X | X | X | ||
| T1mi | Microinvasion | X | ||||
| T1a | X | X | X | X | ||
| T1a1 | X | X | X | X | ||
| T1a2 | X | X | X | X | ||
| T1b | X | X | X | X | ||
| T1b1 | X | X | X | X | ||
| T1b2 | X | X | X | X | ||
| T1c | X | X | X | X | ||
| T1d | X | X | ||||
| T2 | X | X | X | X | ||
| T2a | X | X | X | X | ||
| T2a1 | X | X | ||||
| T2a2 | X | X | ||||
| T2b | X | X | X | X | ||
| T2c | X | X | X | X | ||
| T2d | X | X | ||||
| T3 | X | X | X | X | ||
| T3a | X | X | X | X | ||
| T3b | X | X | X | X | ||
| T3c | X | X | X | X | ||
| T3d | X | X | X | |||
| T4 | X | X | X | X | ||
| T4a | X | X | X | X | ||
| T4b | X | X | X | X | ||
| T4c | X | X | X | X | ||
| T4d | X | X | X | X | ||
| T4e | X | X | ||||
| TX | Primary tumour cannot be assessed | Stadium des Primärtumors kann nicht nachgewiesen werden. |
Tabelle: Codes für Ausdehnung des Primärtumors (T)
Value Set (OID 1.2.276.0.76.11.1)
UICC 5. Auflage (OID 2.16.840.1.113883.15.8)
UICC 6. Auflage (OID 2.16.840.1.113883.15.7)
UICC 7. Auflage (OID 2.16.840.1.113883.15.6)
Nodalstatus (regionäre Lymphknoten) (N)
| UICC | |||||||
|---|---|---|---|---|---|---|---|
| Code | Description | Bedeutung | Entität | 5. | 6. | 7. | SCT |
| N0 | No regional lymph node metastasis | Kein Lymphknotenbefall | alle | X | X | X | X |
| N1 | X | X | X | X | |||
| N1mi | lymph node micrometastasis | alle | X | X?? | |||
| N1mi | Bilateral regional lymph node metastasis | Vulva | X | X ???? | |||
| N1a | alle | X | X | X | X | ||
| N1b | X | X | X | X | |||
| N1b1 | X | ??? | X | ||||
| N1b2 | X | ??? | X | ||||
| N1b3 | X | ??? | X | ||||
| N1b4 | X | ??? | X | ||||
| N1c | X | X ??? | |||||
| N2 | X | X | X | X | |||
| N2a | X | X | X | X | |||
| N2b | X | X | X | X | |||
| N2c | X | X | X | X | |||
| N3 | X | X | X | X | |||
| N3a | X | X | X | ||||
| N3b | X | X | X | ||||
| N3c | X | X | |||||
| NX | Regional lymph nodes cannot be assessed | Lymphknotenbefall unbekannt | X | X | X |
Tabelle: Codes für Nodalstatus (N)
Value Set (OID 1.2.276.0.76.11.2)
UICC 5. Auflage (OID 2.16.840.1.113883.15.8)
UICC 6. Auflage OID 2.16.840.1.113883.15.7)
UICC 7. Auflage (OID 2.16.840.1.113883.15.6)
Fernmetastasen (M)
Der Code zur Beurteilung von Fernmetastasen soll nur in Zusammenhang mit der Art der Tumorerkrankung übermittelt werden
| UICC | ||||||
|---|---|---|---|---|---|---|
| Code | Description | Bedeutung | Entität | 5. | 6. | 7. |
| M0 | No distant metastasis | Fernmetastasen nicht vorhanden (für p nicht anwendbar) | alle | X | X | X |
| M1 | Distant metastasis | Fernmetastasen vorhanden | alle | X | X | X |
| M1a | nur Ösophagus und Prostata | X | X | X | ||
| M1b | nur Ösophagus und Prostata | X | X | X | ||
| M1c | X | X | X | |||
| M1d | X | |||||
| M1e | X | |||||
| MX | Distant metastasis cannot be assessed (für p nicht anwendbar) | alle | X | X | X |
Tabelle 22: Codes für Fernmetastasen (M)
Value Set (OID 1.2.276.0.76.11.3)
UICC 5. Auflage (OID 2.16.840.1.113883.15.8)
UICC 6. Auflage (OID 2.16.840.1.113883.15.7)
UICC 7. Auflage (OID 2.16.840.1.113883.15.6)
Veneninvasion
| Code | Description | Bedeutung |
|---|---|---|
| V0 | no venous invasion | keine Veneninvasion |
| V1 | microscopic venous invasion | mikroskopische Veneninvasion |
| V2 | macroscopic venous invasion | makroskopische Veneninvasion |
| VX | venous invasion cannot be assessed | Veneninvasion nicht feststellbar |
Tabelle: Codes für Veneninvasion
Value Set (OID 1.2.276.0.76.11.6)
UICC 5. Auflage (OID 2.16.840.1.113883.15.8)
UICC 6. Auflage (OID 2.16.840.1.113883.15.7)
UICC 7. Auflage (OID 2.16.840.1.113883.15.6)
Lymphgefäßinvasion
| Code | Description | Bedeutung |
|---|---|---|
| L0 | no lymphatic invasion | keine Lymphsysteminvasion |
| L1 | lymphatic invasion | Lymphsysteminvasion |
| LX | lmphatic invasion cannot be assessed | Lymphsysteminvasion nicht fest¬stellbar |
Tabelle: Codes für Lymphgefäßinvasion
Value Set (OID 1.2.276.0.76.11.7)
UICC 5. Auflage (OID 2.16.840.1.113883.15.8)
UICC 6. Auflage (OID 2.16.840.1.113883.15.7)
UICC 7. Auflage (OID 2.16.840.1.113883.15.6)
Perineuralscheideninvasion
| Code | Description | Bedeutung |
|---|---|---|
| Pn0 | no perineural invasion | keine perineurale Invasion |
| Pn1 | Perineural invasion | perineurale Invasion |
| PnX | unknown | Unbekannt |
Tabelle: Codes für Perineuralinvasion
Value Set (OID 1.2.276.0.76.11.8)
UICC 5. Auflage (OID 2.16.840.1.113883.15.8)
UICC 6. Auflage (OID 2.16.840.1.113883.15.7)
UICC 7. Auflage (OID 2.16.840.1.113883.15.6)
Unterscheidung autoptisch/klinisch/pathologisch
| Code | Description | Bedeutung |
|---|---|---|
| a | autoptisch | |
| c | Assessment according to clinical criteria | Beurteilung nach klinischen Kriterien |
| u | Assessment according to clinical criteria based on ultraound evidence | Beurteilung nach klinischen Kriterien / Ultraschall |
| ct | Assessment according to clinical criteria based on CT evidence | Beurteilung nach klinischen Kriterien /CT |
| mr | Assessment according to clinical criteria based on magnetic resonance evidence | Beurteilung nach klinischen Kriterien / MRT |
| p | Assessment according to the pathological results | nach dem pathologischen Befund |
Tabelle: Codes für Unterscheidung klinisch/pathologisch
Value Set (OID 1.2.276.0.76.11.9)
UICC 5. Auflage (OID 2.16.840.1.113883.15.8)
UICC 6. Auflage (OID 2.16.840.1.113883.15.7)
UICC 7. Auflage (OID 2.16.840.1.113883.15.6)
Beschränkung auf Mukosa
Gilt nur für gastrointestinale Karzinome.
| Code | Description | Bedeutung |
|---|---|---|
| m1 | restricted to mucosa | auf Mukosa beschränkt, 1. Drittel |
| m2 | restricted to mucosa | auf Mukosa beschränkt, 2. Drittel |
| m3 | restricted to mucosa | auf Mukosa beschränkt, 3. Drittel |
Tabelle: Codes für Beschränkung auf Mukosa
Value Set (OID 1.2.276.0.76.11.TODO)
UICC 5. Auflage (OID 2.16.840.1.113883.15.8)
UICC 6. Auflage (OID 2.16.840.1.113883.15.7)
UICC 7. Auflage (OID 2.16.840.1.113883.15.6)
Beschränkung auf Submukosa
Gilt nur für gastrointestinale Karzinome.
| Code | Description | Bedeutung |
|---|---|---|
| sm1 | restricted to submucosa | auf Submukosa beschränkt, 1. Drittel |
| sm2 | restricted to submucosa | auf Submukosa beschränkt, 2. Drittel |
| sm3 | restricted to submucosa | auf Submukosa beschränkt, 3. Drittel |
Tabelle: Codes für Beschränkung auf Submukosa
Value Set (OID 1.2.276.0.76.11.TODO)
UICC 5. Auflage (OID 2.16.840.1.113883.15.8)
UICC 6. Auflage (OID 2.16.840.1.113883.15.7)
UICC 7. Auflage (OID 2.16.840.1.113883.15.6)
C-Faktor (Certainty, Diagnosesicherheit)
| Code | Description | Nachweis |
|---|---|---|
| C1 | Evidence from standard diagnostic means (e.g., inspection, palpation, and standard radiography, intra¬luminal endoscopy for tumours of certain organs) | Nachweis durch diagnostische Standardmethoden (Inspektion, Palpation, einfache Röntgenaufnahmen) |
| C2 | Evidence obtained by special diag¬nostic means (e.g., radiographic imaging in special projections, tomography, computerized tomo¬graphy \[CT\], ultrasonography, lympho¬graphy, angiography; scinti¬graphy; magnetic resonance imaging \[MRI\]; endoscopy, biopsy, and cytology) | Nachweis durch spezielle klinische diagnostische Methoden einschließlich Computertomogramm, Magnet-Resonanz-Tomographie |
| C3 | Evidence from surgical exploration, including biopsy and cytology | Nachweis durch Operation, einschließlich Biopsie und Zytologie |
| C4 | Evidence of the extent of disease following definitive surgery and pathological examination of the resected specimen | Nachweis durch operative Behandlung mit pathologischer Untersuchung der entnommenen Gewebeteile |
| C5 | Evidence from autopsy | Nachweis durch Autopsie |
Tabelle: Codes für C-Faktor (OID 1.2.276.0.76.5.341)
Lokalisation von Fernmetastasen
| Code | Description | Bedeutung | |
|---|---|---|---|
| PUL | Pulmonary | Pulmonal | Lungenmetastase |
| OSS | Osseous | Ossär | Knochenmetastase |
| HEP | Hepatic | Hepatisch | Lebermetastase |
| BRA | Brain | Cerebral | Hirnmetastase |
| LYM | Lymph Nodes | Lymphonodulär | Lymphknotenmetastase |
| OTH | Others | Andere | Andere Metastase |
| MAR | Bone Marrow | Medullär | Knochenmarkmetastase |
| PLE | Pleura | Pleural | RippenfellMetastase |
| ADR | Adrenals | Adrenal | Nebennierenmetastase |
| SKI | Skin | dermal | Hautmetastase |
Tabelle: Codes für Lokalisation von Fernmetastasen (OID 1.2.276.0.76.5.401)
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Entry: TNM-Klassifikation (zur Übermittlung onkolog. Daten)
| Template-Metadaten | |
| Template-Typ | Entry |
| Template ID | 1.2.276.0.76.3.1.131.1.10.3.5 |
| generischeres Template | cdaab2:TNM-Klassifikation-Entry_(Template) |
| genutztes Templates | |
| nutzende Templates | cdaonk:Diagnostik-Section_(Template) |
| abgeleitete Templates | |
| Schwester-Templates | |
| Generelle Beschreibung | TNM-Klassifikation für die onkolog.Übermittlung |
| allg. Erläuterung | |
| Verhältnis zu IHE | neu |
| Ballotierungsstatus | in Arbeit |
| Erweiterbarkeit | geschlossen |
Beschreibung
In einer Diagnostik-Sektion sind mehrere Entries des Typs TNM-Klassifikation zulässig, beispielsweise um sowohl die klinische als auch die pathologische Beurteilung zu übermitteln.
Attribute
|
|
Hier müssen noch die Einschränkungen in Bezug auf das generischere Entry-Template hin! |
|
|
TODO: Abgleich mit TNM-Template aus APSR 2.0 erforderlich! |
| Lvl | RIM | Name | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|
| 1 | act | observation |
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Entry: Generische Observation mit Messwert
| Template-Metadaten | |
| Template-Typ | Entry |
| Template ID | 1.2.276.0.76.3.1.131.1.10.3.8 |
| generischeres Template | |
| genutztes Templates | |
| nutzende Templates | |
| abgeleitete Templates | |
| Generelle Beschreibung | Generische Observation für diagnostische Angaben als Messwert |
| allg. Erläuterung | |
| Verhältnis zu IHE | neu |
| Ballotierungsstatus | in Arbeit |
| Erweiterbarkeit | offen |
Beschreibung
Diagnostische Angaben können als Messwerte übermittelt werden. Für Angaben mit anderen Datentypen vgl. die entsprechenden Abschnitte.
Derzeit wird in diesem Unterabschnitt nur eine Angabe abgebildet. Im Hinblick auf zukünftige Ergänzungen gilt dieses Template jedoch generisch.
Modell
Folgende Struktur wird verwendet:
Abbildung: Generische Observation mit Messwert
Attribute
| Lvl | RIM | Name | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|
| 1 | act | observation | 1..1 | required | ||
| 2 | act | @classCode | CS CNE | 1..1 | F | "OBS" |
| 2 | act | @moodCode | CS CNE | 1..1 | F | "EVN" |
| 2 | act | templateId | II | 1..1 | R | generische Observation mit Messwert |
| 3 | act | @root | 1..1 | F | "1.2.276.0.76.3.1.131.1.10.3.8" | |
| 2 | act | id | II | 0..1 | O | ID der diagnostischen Angabe |
| 3 | act | @root | 1..1 | O | OID des sendenden System, um diagnostische Angaben eindeutig zu identifizieren | |
| 3 | act | @extension | 1..1 | O | ID der diagnostischen Angabe im sendenden System | |
| 2 | act | code | CD CWE | 1..1 | R | |
| 3 | act | @code | 1..1 | R | Code für die Art der Angabe: vgl. nachfolgende Tabelle | |
| 3 | act | @codeSystem | 1..1 | R | Codesystem für die Art der Angabe: vgl. nachfolgende Tabelle | |
| 2 | act | text | ED | 1..1 | R | textliche Beschreibung des codierten Inhalts der Observation, i.d.R. vom sendenden System automatisch generiert |
| 2 | act | effectiveTime | TS | 0..1 | O | Das Datum, an dem die Diagnose gestellt wurde |
| 2 | act | value | PQ | 1..1 | R | Zahlenwert |
| 3 | act | @value | 1..1 | R | numerischer Messwert | |
| 3 | act | @unit | 1..1 | R | Maßeinheit |
Vokabular
Die folgenden diagnostischen Angaben sind zulässig:
| code@code | code@codeSystem | Bedeutung | Kard | zul. Maßeinheiten (value@unit) |
|---|---|---|---|---|
| BreslowMM | 1.2.276.0.76.3.1.131.1.5.1 | Tumordicke in mm nach Breslow (Hauttumoren) | 0..1 | mm |
Tabelle: Diagnostische Angaben, als Messwert übermittelt
Beispiel
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.276.0.76.3.1.131.1.10.3.8"/>
<id extension="774411"
root="1.2.276.0.76.3.1.131.1.4.3.9999.9999.999919"/>
<code code="BreslowMM"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
Tumordicke nach Breslow: 1,95 mm
</text>
<effectiveTime value="20120825"/>
<value xsi:type="PQ" value="1.95" unit="mm"/>
</observation>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Entry: Generische Observation mit boolscher Angabe
| Template-Metadaten | |
| Template-Typ | Entry |
| Template ID | 1.2.276.0.76.3.1.131.1.10.3.2 |
| generischeres Template | |
| genutztes Templates | |
| nutzende Templates | |
| abgeleitete Templates | |
| Generelle Beschreibung | Generische Observation für boolsche diagnostische Angaben |
| allg. Erläuterung | |
| Verhältnis zu IHE | neu |
| Ballotierungsstatus | |
| Erweiterbarkeit | offen |
Beschreibung
Einige diagnostische Angaben werden als boolsche Werte übermittelt. Für Angaben mit anderen Datentypen vgl. die entsprechenden Abschnitte.
Modell
Folgende Struktur wird verwendet:
Abbildung: Generische Observation mit boolscher Angabe
Attribute
| Lvl | RIM | Name | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|
| 1 | act | observation | 1..1 | R | ||
| 2 | act | @classCode | CS CNE | 1..1 | F | "OBS" |
| 4 | act | @moodCode | CS CNE | 1..1 | F | "EVN" |
| 4 | act | templateId | II | 1..1 | R | generische Observation mit boolscher Angabe
@root="1.2.276.0.76.3.1.131.1.10.3.2" |
| 4 | act | id | II | 0..1 | O | ID der diagnostischen Angabe |
| 4 | act | code | CD CWE | 1..1 | R | Code für die Art der Angabe: vgl. nachfolgende Tabelle |
| 4 | act | text | ED | 1..1 | R | textliche Beschreibung des codierten Inhalts der Observation, i.d.R. vom sendenden System automatisch generiert |
| 4 | act | effectiveTime | TS | 0..1 | O | Das Datum, an dem die Diagnose gestellt wurde |
| 4 | act | value | BL | 1..1 | R | boolscher Wert |
| 5 | act | @value | 0..1 | R | "true" oder "false" | |
| 5 | act | @nullFlavor | 0..1 | F | "UNK" |
Constraint
- es muss entweder das value-Attribut oder das nullFlavor-Attribut existieren
Vokabular
Die folgenden diagnostischen Angaben sind zulässig:
| code@code | code@codeSystem | Bedeutung | Kard |
|---|---|---|---|
| fruehereChemotherapie | 1.2.276.0.76.3.1.131.1.5.1 | frühere Chemotherapie | 0..1 |
| fruehereStrahlentherapie | 1.2.276.0.76.3.1.131.1.5.1 | frühere Strahlentherapie | 0..1 |
Tabelle: Diagnostische Angaben, als boolsche Werte übermittelt
Beispiel
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.276.0.76.3.1.131.1.10.3.2"/>
<id extension="885522"
root="1.2.276.0.76.3.1.131.1.4.3.9999.9999.999918"/>
<code code="fruehereChemotherapie"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
Frühere Chemotherapie: ja
</text>
<effectiveTime value="2003"/>
<value xsi:type="BL" value="true"/>
</observation>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Entry: Generische Observation mit codierter Angabe
| Template-Metadaten | |
| Template-Typ | Entry |
| Template ID | 1.2.276.0.76.3.1.131.1.10.3.1 |
| generischeres Template | |
| genutztes Templates | |
| abgeleitete Templates | |
| Generelle Beschreibung | Generische Observation für codierte diagnostische Angaben |
| allg. Erläuterung | |
| Verhältnis zu IHE | |
| Ballotierungsstatus | in Arbeit |
| Erweiterbarkeit | offen |
Beschreibung
Zahlreiche diagnostische Angaben werden als codierte Werte übermittelt. Für Angaben mit anderen Datentypen vgl. die entsprechenden Abschnitte.
Modell
Folgende Struktur wird verwendet:
Abbildung: Generische Observation mit codierter Angabe
Attribute
| Lvl | RIM | Name | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|
| 1 | act | observation | 1..1 | M | generische Beobachtung zur Übermittlung kodierter Informationen | |
| 2 | act | @classCode | CS CNE | 1..1 | F | "OBS" |
| 2 | act | @moodCode | CS CNE | 1..1 | F | "EVN" |
| 2 | act | templateId | II | 1..1 | R | generische Observation mit codierter Angabe |
| 3 | act | @root | 1..1 | F | "1.2.276.0.76.3.1.131.1.10.3.1" | |
| 2 | act | id | II | 0..1 | O | ID der diagnostischen Angabe |
| 3 | act | @root | 1..1 | R | OID des sendenden System, um diagnostische Angaben eindeutig zu identifizieren | |
| 3 | act | @extension | 1..1 | R | ID der diagnostischen Angabe im sendenden System | |
| 2 | act | code | CD CWE | 1..1 | R | welche Information wird hier kodiert übermittelt (vgl. nachfolgende Tabelle) |
| 2 | act | text | ED | 0..1 | O | textliche Beschreibung des codierten Inhalts der Observation: i.d.R. vom sendenden System automatisch generiert |
| 2 | act | effectiveTime | TS | 0..1 | O | Das Datum, an dem die Beobachtung gestellt wurde |
| 2 | act | value | CD | 1..1 | R | Code für die jeweilige Beobachtung; die Werte werden aus dem entsprehcendne Value Set entnommen (vgl. nachfolgende Tabelle) |
Value Sets
Die folgenden diagnostischen Angaben sind zulässig. Die Kardinalität gibt hierbei an, wie oft die gesamte Beobachtung innerhalb einer Sektion auftaruchen kann:
| Code | Codesystem | Bedeutung | Valueset | Kard. |
|---|---|---|---|---|
| gekidDiagnosesicherung | 1.2.276.0.76.3.1.131.1.5.1 | Art der Diagnosesicherung | 1.2.276.0.76.3.1.131.1.5.3 | 0..1 |
| gekidDiagnoseanlass | 1.2.276.0.76.3.1.131.1.5.1 | Diagnoseanlass | 1.2.276.0.76.3.1.131.1.5.4 | 0..1 |
| gekidGrobstadium | 1.2.276.0.76.3.1.131.1.5.1 | Grobstadium | 1.2.276.0.76.3.1.131.1.5.5 | 0..1 |
| CCCOND | 1.2.276.0.76.5.342 | Begleiterkrankung | ICD-10 (jahresabhängig) | 0..* |
Tabelle: Diagnostische Angaben, codiert übermittelt
|
|
Der Großteil der zu übermittelnden Werte lassen sich als Scores und Assessments ausdrücken! |
Beispiel
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.276.0.76.3.1.131.1.10.3.1"/>
<id extension="beglerk12345"
root="1.2.276.0.76.3.1.131.1.4.3.9999.9999.999908"/>
<code code="CCCOND"
codeSystem="1.2.276.0.76.5.342"/>
<text>
Begleiterkrankung: Diabetes mellitus mit Ketoazidose seit 11/2003
</text>
<effectiveTime value="200311"/>
<value xsi:type="CD" code="E10.1"
codeSystem="1.2.276.0.76.5.409"/>
</observation>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Entry: Gleason-Score
| Template-Metadaten | |
| Template-Typ | Entry |
| Template ID | 1.2.276.0.76.3.1.131.1.10.3.9 |
| generischeres Template | Assessment Scales |
| genutztes Template | |
| nutzende Templates | |
| abgeleitete Templates | |
| Schwester-Templates | |
| Generelle Beschreibung | Gleason-Score (Prostatakarzinom) |
| allg. Erläuterung | |
| Ballotierungsstatus | in Arbeit |
| Erweiterbarkeit | geschlossen |
Der Gleason Score setzt sich aus der Summe des häufigsten und des zweithäufigsten Gleason-Musters im OP-Präparat bzw. des häufigsten und des höchsten Gleason-Musters in der Stanze zusammen. Ist nur ein Muster erkennbar (häufig in Stanzbiopsien), so wird der Score aus dem verdoppelten Musterwert gebildet. An der Stanze können Gleason-Muster <3 nicht sicher erkannt werden, weshalb die kleinsten Werte in der Regel mindestens 3 sind.
Für die Erkennung des sekundären Gleason Musters werden mindestens 5% Anteil am
Tumorgewebe gefordert. Ist das nicht erreicht, wird ebenfalls ein verdoppelter Musterwert
des primären Musters gebildet.
Damit ergeben sich Gesamtwerte von 2 bis 10. Da die Reihenfolge primär/sekundär in
einigen Fällen prognostisch relevant sind, tauchen bei reiner Summenangabe auch Werte
wie 7a (=3+4) und 7b (=4+3) auf. Die Summenwerte korrelieren wiederum mit der reinen
Gradingangabe
In Resektionspräparaten kann ein drittes und höheres Muster gefunden werden, das auch weniger 5% Anteil des Tumors beteiffen kann. Der Gleason Score wird in seiner Summe (Addition zweier Werte) davon jedoch nicht beinflusst. Die Gesamtdarstellung in klinischen Befundtexten könnte lauten: "Gleason-Score 3+4=7, tertiäres Muster 5".
Modell
|
|
TODO: Ergänzung durch neues Gleason-Grading von 2015 (zusätzlich zum Score) erforderlich! |
Attribute
| Lvl | RIM | Name | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|
| 3 | act | observation | 1..1 | M | Gleason-Score fix: @classCode="OBS" | |
| 4 | act | templateId | II | 1..1 | F | @root="1.2.276.0.76.3.1.131.1.10.3.9" |
| 4 | act | id | II | 0..1 | optional | ID der diagnostischen Angabe @root=OID des sendenden System, um diagnostische Angaben eindeutig zu identifizieren |
| 4 | act | code | CD CWE | 1..1 | F | @code="35266-6" @codeSystem=LOINC: "2.16.840.1.113883.6.1" |
| 4 | act | text | ED | 1..1 | required | textliche Beschreibung des codierten Inhalts der Observation; wird i.d.R. vom sendenden System automatisch generiert |
| 4 | act | effectiveTime | TS | 0..1 | O | Das Datum, an dem die Diagnose gestellt wurde |
| 4 | act | value | CD | 1..1 | required | Gleason-Score Value Set: todo.valueset.gleason001 |
| 5 | act | entryRelationship | 0..1 | O | @typeCode="COMP" | |
| 6 | act | observation | 1..1 | M | Gleason 1 (primäres Gleason-Muster) | |
| 7 | act | code | 1..1 | F | @code=LOINC: "44641-9"
@codeSystem=LOINC: "2.16.840.1.113883.6.1" | |
| 6 | act | value | CV | 1..1 | required |
Value Set: todo.valueset.gleason002 |
| 5 | act | entryRelationship | 0..1 | optional | @typeCode="COMP" | |
| 6 | act | observation | 1..1 | M | Gleason 2 (sekundäres Gleason-Muster) | |
| 6 | act | code | CV | 1..1 | F | @code=LOINC: "44642-7" @codeSystem=LOINC: "2.16.840.1.113883.6.1" |
| 6 | act | value | CV | 1..1 | required |
Value Set: todo.valueset.gleason002 |
| 5 | act | entryRelationship | 0..1 | optional | @typeCode="COMP" | |
| 6 | act | observation | 1..1 | M | Gleason 3 (tertiäres Gleason-Muster) | |
| 6 | act | code | CV | 1..1 | required | @code= LOINC: "44643-5" @codeSystem=LOINC: "2.16.840.1.113883.6.1" |
| 6 | act | value | CV | 1..1 | required |
Value Set: todo.valueset.gleason002 |
Beispiel
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.276.0.76.3.1.131.1.10.3.9"/>
<id extension="1234009"
root="1.2.276.0.76.3.1.131.1.4.3.9999.9999.999919"/>
<code code="35266-6"
codeSystem="2.16.840.1.113883.6.1"/>
<text>
Gleason-Score: 4+5=9
</text>
<effectiveTime value="20110326"/>
<value xsi:type="CD" code="9" codeSystem="1.2.276.0.76.5.404"/>
<!-- Einzelwerte zum Score: 1. Wert -->
<entryRelationship typeCode='COMP'>
<observation moodCode='EVN' classCode='OBS'>
<code code='44641-9' displayName=''
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC' />
<value xsi:type='CD' code='4' codeSystem='1.2.276.0.76.5.402'/>
</observation>
</entryRelationship>
<!-- Einzelwerte zum Score: 2. Wert -->
<entryRelationship typeCode='COMP'>
<observation moodCode='EVN' classCode='OBS'>
<code code='44642-7' displayName=''
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC' />
<value xsi:type='CD' code='5' codeSystem='1.2.276.0.76.5.402'/>
</observation>
</entryRelationship>
</observation>
Vokabular
Gleason Score
Value Set OID todo.valueset.gleason001
Code=code="35266-6"
codeSystem=LOINC: "2.16.840.1.113883.6.1"
Code=code="372278000"
codeSystem=Snomed CT: "2.16.840.1.113883.6.96"
Code System OID 1.2.276.0.76.5.404
Ergänzend zur Codierung ist auch die Zuordnung zum Grading (Malignitätsgrad) hier aufgeführt.
| Snomed CT | Code | Bedeutung | Malignitätsgrad | Bedeutung |
|---|---|---|---|---|
| 2 | Gleason-Score 2 | G1 | gute Prognose | |
| 46677009 | 3 | Gleason-Score 3 | G1 | gute Prognose |
| 18430005 | 4 | Gleason-Score 4 | G1 | gute Prognose |
| 74013009 | 5 | Gleason-Score 5 | G2 | gute Prognose |
| 84556003 | 6 | Gleason-Score 6 | G2 | gute Prognose |
| 57403001 | 7 | Gleason-Score 7 | G2-G3 | gute / schlechte Prognose |
| 7a | Gleason-Score 7a (ergibt sich aus 3+4) | G2 | gute Prognose | |
| 7b | Gleason-Score 7b (ergibt sich aus 4+3) | G3 | schlechte Prognose | |
| 33013007 | 8 | Gleason-Score 8 | G3 | schlechte Prognose |
| 58925000 | 9 | Gleason-Score 9 | G3 | schlechte Prognose |
| 24514009 | 10 | Gleason-Score 10 | G3 | schlechte Prognose |
Gleason: Gewebsmuster
Value Set OID todo.valueset.gleason002
Code System OID 1.2.276.0.76.5.402
| Code | Bedeutung |
|---|---|
| 1 | Muster 1 = scharf begrenzter Tumorknoten, Drüsen gleichförmig, dicht gepackt, mittelgroß. |
| 2 | Muster 2 = nicht ganz scharf begrenzter Tumorknoten, Drüsen locker und ungleichmäßig. |
| 3 | Muster 3 = unscharf begrenzter Knoten, Drüsen klein und ungleichmäßig, evtl. kleine kribriforme Strukturen. |
| 4 | Muster 4 = unscharf begrenzter Knoten, Drüsen z.T. ohne Lichtungen, kribriform, fusioniert. |
| 5 | Muster 5 = unscharf begrenzter Knoten, Verlust drüsiger Differenzierung, meist solide Formen, einzelzellig, oder hypernephroid. |
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Elston-Ellis-Score Entry (Template) (Nottingham-Grading)
|
|
Zu diesem Score-System existiert ein fertiger Value Set in cdaonk Assessment scales Elston-Ellis. Abgleich erforderlich G.H. |
| Template-Metadaten | |
| Template-Typ | Entry |
| Template ID | <OID für das Template> |
| generischeres Template | Assessment Scales |
| genutzte Templates | |
| nutzende Templates | Grading |
| abgeleitete Templates | |
| Generelle Beschreibung | Diese Seite beschreibt die Darstellung des Elston-Ellis-Scores |
| allg. Erläuterung | Scores_und_Assessments |
| Verhältnis zu IHE | neu |
| Ballotierungsstatus | in Arbeit |
| Erweiterbarkeit | geschlossen |
Übersicht
Die Elemente werden, sofern spezialisiert/abweichend vom übergeordneten Template, hier als Walk Through beschrieben:
| Lvl | RIM | Name | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|
| 1 | act | observation | 1..1 | M | Elston-Ellis-Score (Nottingham) | |
| 2 | act | code | CD CWE | 1..1 | F | @code: "396293007", @codeSystem: 2.16.840.1.113883.6.96 |
| 2 | act | value | CD CWE | 1..1 | M | Wert aus dem Scoresystem. Value Set: todo.valueset.xxy004 |
| 2 | rel | entryRelationship | 0..1 | O | 1. Komponente | |
| 3 | act | observation | 1..1 | O | Tubulusbildung (Tubule formation score) | |
| 4 | act | code | CD CWE | 1..1 | F | @code: "371470008", @codeSystem: 2.16.840.1.113883.6.96 |
| 4 | act | value | CD CWE | 1..1 | M | Value Set: todo.valueset.xxy005 |
| 2 | rel | entryRelationship | 0..1 | O | 2. Komponente | |
| 3 | act | observation | 1..1 | O | Kernpolymorphie (Nuclear pleomorphism score) | |
| 4 | act | code | CD CWE | 1..1 | F | @code: "371471007", @codeSystem: 2.16.840.1.113883.6.96 |
| 4 | act | value | CD CWE | 1..1 | M | Value Set: todo.valueset.xxy006 |
| 2 | rel | entryRelationship | 0..1 | O | 3. Komponente | |
| 3 | act | observation | 1..1 | O | Mitoserate (Number of mitoses per 10 high power fields) | |
| 4 | act | code | CD CWE | 1..1 | F | @code: "406094009", @codeSystem: 2.16.840.1.113883.6.96 |
| 4 | act | value | CD CWE | 1..1 | M | Value Set: todo.valueset.xxy007 |
Value Sets
Elston-Ellis-Score
Value Set OID: todo.valueset.xxy004
Code System OID: todo.codesystem.xxy004
Code: @code="44648-4" @codeSystem="2.16.840.1.113883.6.1"
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| 3 | Elston-Ellis-Score 3 | |
| 4 | Elston-Ellis-Score 4 | |
| 5 | Elston-Ellis-Score 5 | |
| 6 | Elston-Ellis-Score 6 | |
| 7 | Elston-Ellis-Score 7 | |
| 8 | Elston-Ellis-Score 8 | |
| 9 | Elston-Ellis-Score 9 |
Tubulusbildung
Value Set OID: todo.valueset.xxy005
Code System OID: 2.16.840.1.113883.6.96
in LOINC: Tubulusbildung=44644-3, 2.16.840.1.113883.6.1
| Score | Snomed CT Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|---|
| 1 | 369778004 | > 75% | Breast tubule formation: Majority of tumor >75% |
| 2 | 369779007 | 10-75% | Breast tubule formation: Moderate 10% to 75% |
| 3 | 369780005 | < 10% | Breast tubule formation: Minimal <10% |
Kernpolymorphie
Value Set OID: todo.valueset.xxy006
Code System OID: 2.16.840.1.113883.6.96
in LOINC: Kernpolymorphie=44645-0, 2.16.840.1.113883.6.1
| Score | Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|---|
| 1 | 384735004 | gering | Nuclear pleomorphism: small regular nuclei |
| 2 | 384737007 | mäßig | Nuclear pleomorphism: moderate increase in size, etc. |
| 3 | 384738002 | stark | Nuclear pleomorphism: marked variation in size, nucleoli, chromatin clumping, etc. |
Mitoserate
Die Grundlage dieses Scores (Anzahl Mitosen/10 HPF) ist von der Beobachtungssituation des Pathologen abhängig (Größe des Gesichtsfeldes). Daher wird hier nicht die hierauf fixierte Codierung aus SNOMED-CT verwendet.
Value Set OID: todo.valueset.xxy007
Code System OID: todo.codesystem.xxy007
in LOONC: Mitoserate*=44650-0, 2.16.840.1.113883.6.1
| Score | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| 1 | z.B. SFZ 25: 0-10 / SFZ 20: 0-8 | |
| 2 | z.B. SFZ 25: 11-20 / SFZ 20: 9-16 | |
| 3 | z.B. SFZ 25: > 20 / SFZ 20: > 16 |
Value Set (OID TODO - Code System 1.2.276.0.76.5.336??):
| ScoreItem | Code | CodeSystem | Kriterien | Value |
|---|---|---|---|---|
| Mitoserate* | 44650-0 | 2.16.840.1.113883.6.1 | 0-5/10 HPF | 1 |
| Mitoserate* | 44650-0 | 2.16.840.1.113883.6.1 | 6-11/10 HPF | 2 |
| Mitoserate* | 44650-0 | 2.16.840.1.113883.6.1 | >11/10 HPF | 3 |
- ... die Schwellwerte der Mitoserate sind abhängig vom verwendeten Objektiv (Gesichtsfeldgröße). Die hier angegebenen Werte gelten für Gesichtsfelddurchmesser von 0,44-0,45 mm.
unklar / doppelt ???
Aus den drei Scorewerten wird ein Summenscore für den Malignitätsgrad gebildet.
| InterpretationCode | ObservationRange | Interpretation |
|---|---|---|
| MG1 | 3-5 | grade 1 (gut differenziert) |
| MG2 | 6-7 | grade 2 (mäßig differenziert) |
| MG3 | 8-9 | grade 3 (schlecht differenziert) |
Beispiel
<observation moodCode='EVN' classCode='OBS'>
<templateID root='' />
<id root='' extension='' />
<code code='396293007' codeSystem='2.16.840.1.113883.6.96' />
<statusCode code='completed'/>
<value xsi:type='CD' code='7' codeSystem='todo.codesystem.xxy004'/>
<entryRelationship typeCode='COMP'>
<observation moodCode='EVN' classCode='OBS'>
<code code='371470008' codeSystem='2.16.840.1.113883.6.96' />
<statusCode code='completed'/>
<value xsi:type='CD' code='369780005' codeSystem='2.16.840.1.113883.6.96'/>
</observation>
</entryRelationship>
<entryRelationship typeCode='COMP'>
<observation moodCode='EVN' classCode='OBS'>
<code code='371471007' codeSystem='2.16.840.1.113883.6.96' />
<statusCode code='completed'/>
<value xsi:type='CD' code='384737007' codeSystem='2.16.840.1.113883.6.96'/>
</observation>
</entryRelationship>
<entryRelationship typeCode='COMP'>
<observation moodCode='EVN' classCode='OBS'>
<code code='406094009' codeSystem='2.16.840.1.113883.6.96' />
<statusCode code='completed'/>
<value xsi:type='CD' code='2' codeSystem='todo.codesystem.xxy007'/>
</observation>
</entryRelationship>
</observation>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Breslow-Index Entry (Template)
| Template-Metadaten | |
| Template-Typ | Entry |
| Template ID | <OID für das Template> |
| generischeres Template | cdaab2:Assessment_Scales_Entry_(Template) |
| genutztes Templates | |
| nutzende Templates | |
| abgeleitete Templates | |
| Schwester-Templates | cdaab2:Gleason-Score-Entry_(Template) |
| Generelle Beschreibung | Diese Seite beschreibt die Darstellung des Breslow-Indexes (Breslow-Levels) sowie der Tumordicke nach Breslow. |
| allg. Erläuterung | Scores_und_Assessments |
| Verhältnis zu IHE | neu |
| Ballotierungsstatus | in Arbeit |
| Erweiterbarkeit | offen |
Übersicht
Die Elemente werden, sofern spezialisiert/abweichend vom übergeordneten Template, hier als Walk Through beschrieben:
| Lvl | RIM | Name | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|
| 1 | act | observation | 1..1 | M | Breslow-Index | |
| 2 | act | code | CD CWE | 1..1 | F | @code: "106243009", @codeSystem: 2.16.840.1.113883.6.96 |
| 2 | act | value | CD CWE | 1..1 | M | Die entsprechenden Stages. Value Set: todo.valueset.xx001 |
| 2 | rel | entryRelationship | 0..1 | O | Komponente | |
| 3 | act | observation | 1..1 | M | genaue Tumordicke, die für Stages nach Breslow zugrunde liegt. | |
| 4 | act | code | CD CWE | 1..1 | F | @code: "385348009", @codeSystem: 2.16.840.1.113883.6.96 |
| 4 | act | value | PQ | 1..1 | R | @value: Tumordicke fix: @unit="mm" |
| 5 | rel | entryRelationship | 0..0 | NP | wir untersützten keinen Referenz Range |
Value Sets
Breslow-Index
|
|
Breslow-Index und Breslow-Dicke (Breslow depth) sind synonym gebraucht, wobei der B.-Index vom Erstbeschreiber so genannt wurde. Es ist ein reines Längenmaß, kein Score!. Insofern ist auch die Abbildung nicht zutreffend. Verschiedentlich wurden Schwellwerte genutzt, um Längen zu klassifizieren. Lange Zeit war 0,75 mm eine solche Schwelle. Ist heute bedeutungslos geworden. Wo stammen die Zahlen für den untenstehenden Value Set her? Vgl. z.B. Wikipedia [1] und die untenstehende Tabelle mit SNOMED-CT Codes! Zwei Value sets mit jeweils 3, z.T. überlappenden values! |
Value Set OID: todo.valueset.xx001
Code System OID: 1.2.276.0.76.3.1.131.1.5.26
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| I | <= 0,75 mm | |
| II | 0,76-1,50 mm | |
| III | 1,51-2,25 mm | |
| IV | 2,26-3,00 mm | |
| V | > 3,00 mm |
Originär lt. Breslow gilt folgende Einteilung:
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| <= 0,75 mm | ||
| 0,76-1,50 mm | ||
| > 1,50 mm |
Snomed CT hat wiederum folgende Einteilung:
| Snomed CT Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| 86069005 | <= 0,75 mm | |
| 17456000 | 0,76-1,75 mm | |
| 44815009 | > 1,75 mm |
{{WorkBox|
Diese drei Einteilungen sind zu verifizieren!
Ggf. müssen wir alle drei verwenden. Dann wäre die Graduierung dem zugeordneten Codesystem zu entnehmen.
|
|
Das Nachfolgende ist noch zu prüfen und genauer anzugeben. |
Snomed CT bietet auch ein paar Codes an, die allerdings zwei verschiedene Gradings repräsentieren:
|
|
SNOMED bildet die Breslow-Dicke als Staging-System allerdings in zwei verschiedenen Achsen unter teilweise verschiedenen (nichtoffiziellen) Bezeichnungen ab. Die Kodierung 385348009, die netterweise Value Sets einschließt, beschreibt leider nicht den Index, sondern nur Messergebnisse in drei Klassen (die wie der Breslow-Index zur Prognoseeinschätzung von Melanomen genutzt werden). In D wird das als Breslow-Dicke bezeichnet. Breslow-Index und Clark-Level sind heutzutage beide nicht mehr notwendige Bestandteile eines Melanom-Staging. Ihre Komponenten gehen teilweise in der TNM-Klassifikation auf. Der Schwerpunkt ist hier auf die Methode der Dickenmessung gelegt: Es wird genau angegeben, von wo bis wo die Dicke zu messen ist. Dieselbe Methode liegt aber eben auch dem Breslow-Index zugrunde. Das kann man aber in SNOMED CT nicht auf Anhieb erkennen. |
| fully specified Concept | Snomed CT | other description | hierarchy |
|---|---|---|---|
| Breslow depth staging for melanoma (observable entity) | (106243009) | Breslow depth staging for melanoma | Observable entity |
| Breslow system for melanoma staging (tumor staging) | (385346008) | Breslow system for melanoma staging | Staging and Scales |
| Breslow depth finding for melanoma (finding) | (385348009) | Breslow depth finding for melanoma | Clinical findings |
Für den letzten Eintrag stehen folgende Werte (Codes) zur Verüfung:
| fully specified Concept | Snomed CT | other description | hierarchy |
|---|---|---|---|
| Breslow measurement - depth less than 0.76 mm (finding) | (86069005) | Breslow measurement - depth less than 0.76 mm; Depth less than 0.76 mm | Clinical findings |
| Breslow measurement - depth from 0.76 to 1.75 mm (finding) | (17456000) | Depth from 0.76 to 1.75 mm; Breslow measurement - depth from 0.76 to 1.75 mm | Clinical findings |
| Breslow measurement - greater than 1.75 mm (finding) | (44815009) | Breslow measurement - greater than 1.75 mm; Greater than 1.75 mm | Clinical findings |
| Breslow measurement - Invasion 0.76 to 1.5mm from deep border of granular layer (finding) | (369714007) | Breslow measurement - Invasion 0.76 to 1.5mm from deep border of granular layer | Clinical findings |
| Breslow measurement - Invasion > 1.5mm from deep border of granular layer (finding) | (369715008) | Breslow measurement - Invasion > 1.5mm from deep border of granular layer | Clinical findings |
Beispiel
<observation moodCode='EVN' classCode='OBS'>
<templateID root='' />
<id root='' extension='' />
<!-- um welches Score-System handelt es sich -->
<code code='106243009' codeSystem='2.16.840.1.113883.6.96' />
<statusCode code='completed'/>
<!-- eigentlicher Scorewert -->
<value xsi:type='CD' code='1' codeSystem='1.2.276.0.76.3.1.131.1.5.26'/>
<!-- Messwert -->
<entryRelationship typeCode='COMP' inversionInd='false'>
<observation moodCode='EVN' classCode='OBS'>
<code code='385348009' codeSystem='2.16.840.1.113883.6.96' />
<statusCode code='completed'/>
<value xsi:type='PQ' value='1.25' unit='mm' />
</observation>
</entryRelationship>
</observation>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Section: Erkrankungsdaten
| Template-Metadaten | |
| Template-Typ | Section |
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.2 |
| generischeres Template | |
| genutztes Templates | |
| nutzende Templates | |
| abgeleitete Templates | |
| Generelle Beschreibung | In diesem Abschnitt werden die Daten zur Erkankung übermittelt. |
| allg. Erläuterung | |
| Verhältnis zu IHE | dt.Übersetzung oder Ergänzung oder neu |
| Ballotierungsstatus | |
| Erweiterbarkeit | offen oder geschlossen |
Modell
Abbildung: Erkrankungsdaten
Attribute
| Lvl | RIM | Name | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|
| 1 | act | section | 1..1 | required | ||
| 2 | act | @classCode | CS CNE | 1..1 | required | "DOCSECT" |
| 2 | act | @moodCode | CS CNE | 1..1 | required | "EVN" |
| 2 | act | templateID | II | 1..1 | required | Erkrankungsabschnitt |
| 3 | act | @root | 1..1 | fix | "1.2.276.0.76.3.1.131.1.10.2.2" | |
| 2 | act | code | CE CWE | 0..1 | optional | Code für Erkrankungsabschnitt |
| 3 | act | @code | 1..1 | fix | "54532-7" | |
| 3 | act | @codeSystem | 1..1 | fix | "2.16.840.1.113883.6.1" | |
| 2 | act | title | ST | 1..1 | fix | "Erkrankung" |
| 2 | act | text | ED | 1..1 | required | textliche Beschreibung des codierten Inhalts der Section, i.d.R. vom sendenden System automatisch generiert |
| 2 | rel | entry | 1..1 | required | ||
| 3 | act | observation | 1..1 | required | ||
| 4 | act | @classCode | CS CNE | 1..1 | fix | "OBS" |
| 4 | act | @moodCode | CS CNE | 1..1 | fix | "EVN" |
| 4 | act | id | II | 0..1 | optional | ID der Erkrankung |
| 5 | act | @root | 1..1 | optional | OID für Erkrankungen, OID des sendenden System, um Erkrankungen eindeutig zu identifizieren | |
| 5 | act | @extension | 1..1 | optional | ID der Erkrankung im sendenden System | |
| 4 | act | code | CD CWE | 1..1 | required | |
| 5 | act | @code | 1..1 | fix | "NEO" | |
| 5 | act | @codeSystem | 1..1 | fix | "1.2.276.0.76.5.342" | |
| 4 | act | text | ED | 1..1 | required | textliche Beschreibung des codierten Inhalts der Observation, i.d.R. vom sendenden System automatisch generiert |
| 4 | act | effectiveTime | TS | 1..1 | required | Zeitpunkt der ersten gesicherten Diagnose der Erkrankung |
| 4 | act | value | CD | 1..1 | required | Hauptdiagnose der Erkrankung |
| 5 | act | @code | 1..1 | required | eigentliche Hauptdiagnose, Codesystem: OID für die verwendete ICD-10-Version | |
| 5 | act | @codeSystem | 1..1 | required | OID der verwendeten ICD-10-Version | |
| 5 | act | originalText | ED | 0..1 | optional | Freitext-Beschreibung der Diagnose |
| 5 | act | qualifier | CR | 0..1 | optional | |
| 6 | act | name | CV | 1..1 | required | |
| 7 | act | @code | 1..1 | fix: Seitenlokalisation | "78615007" | |
| 7 | act | @codeSystem | 1..1 | fix: SNOMED CT | "2.16.840.1.113883.6.96" | |
| 6 | act | value | CV | 1..1 | required | Seitenlokalisation |
| 7 | act | @code | 1..1 | required | eigentliche Seitenangabe, Codesystem: 1.2.276.0.76.5.412 | |
| 7 | act | @codeSystem | 1..1 | fix | "1.2.276.0.76.5.412" | |
| 2 | rel | entryRelationship | 0..0 | nicht verwendet |
|
|
TODO: Adresse zum Erkrankungszeitpunkt? |
Beispiel
<section classCode="DOCSECT" moodCode="EVN">
<templateId root="1.2.276.0.76.3.1.131.1.10.2.2"/>
<code code="54532-7" codeSystem="2.16.840.1.113883.6.1"/>
<title>Erkrankungsdaten</title>
<text>
<paragraph>
Bösartige Neubildung: Oberer äußerer Quadrant der Brustdrüse links, diagnostiziert am 19.03.2011
</paragraph>
</text>
<entry>
<observation classCode="OBS" moodCode="EVN">
<id extension="tumor12345"
root="1.2.276.0.76.3.1.131.1.4.3.9999.9999.999904"/>
<code code="NEO"
codeSystem="1.2.276.0.76.5.342"/>
<text>
Bösartige Neubildung: Oberer äußerer Quadrant der Brustdrüse links, diagnostiziert am 19.03.2011
</text>
<effectiveTime value="20110319"/>
<value xsi:type="CD" code="C50.4"
codeSystem="1.2.276.0.76.5.409">
<originalText>Mammakarzinom links, oben außen</originalText>
<qualifier>
<name code="78615007"
codeSystem="2.16.840.1.113883.6.96"/>
<value xsi:type="CD" code="L"
codeSystem="1.2.276.0.76.5.412"/>
</qualifier>
</value>
</observation>
</entry>
</section>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Section: Phänomendaten
| Template-Metadaten | |
| Template-Typ | Section |
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.6 |
| generischeres Template | |
| genutztes Templates | |
| nutzende Templates | |
| abgeleitete Templates | |
| Generelle Beschreibung | In diesem Abschnitt werden die Daten zu Phänomenen übermittelt. |
| allg. Erläuterung | |
| Verhältnis zu IHE | dt.Übersetzung oder Ergänzung oder neu |
| Ballotierungsstatus | |
| Erweiterbarkeit | offen oder geschlossen |
| 21855-2 21920-4 |
O | Primary site Cancer Site of distant metastasis Cancer |
Modell
Abbildung: Phänomendaten
Attribute
| Lvl | RIM | Name | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|
| 1 | act | section | 1..1 | R | ||
| 2 | act | @classCode | CS CNE | 1..1 | F | "DOCSECT" |
| 2 | act | @moodCode | CS CNE | 1..1 | F | "EVN" |
| 2 | act | templateID | II | 1..1 | R | Phänomendaten-Abschnitt |
| 3 | act | @root | 1..1 | F | "1.2.276.0.76.3.1.131.1.10.2.6" | |
| 2 | act | code | CE CWE | 0..1 | O | Code für Phänomendatenabschnitt |
| 3 | act | @code | 1..1 | O | eigentlicher Code, abhängig von der Art des Phänomens, Value Set 1.2.276.0.76.3.1.131.1.11.4 | |
| 3 | act | @codeSystem | 1..1 | F | LOINC: "2.16.840.1.113883.6.1" | |
| 2 | act | title | ST | 1..1 | R | "Phänomen: Primärtumor" oder "Phänomen: Metastase", abhängig von der Art des Phänomens |
| 2 | act | text | ED | 1..1 | R | textliche Beschreibung des codierten Inhalts der Section, i.d.R. vom sendenden System automatisch generiert |
| 2 | rel | entry | 1..1 | R | ||
| 3 | act | observation | 1..1 | R | ||
| 4 | act | @classCode | CS CNE | 1..1 | F | "OBS" |
| 4 | act | @moodCode | CS CNE | 1..1 | F | "EVN" |
| 4 | act | id | II | 0..1 | O | ID des Phänomens |
| 5 | act | @root | 1..1 | O | OID für Phänomene, OID des sendenden System, um Phänomene eindeutig zu identifizieren | |
| 5 | act | @extension | 1..1 | O | ID des Phänomens im sendenden System | |
| 4 | act | code | CD CWE | 1..1 | R | |
| 5 | act | @code | 1..1 | R | eigentlicher Code, abhängig von der Art des Phänomens, Value Set 1.2.276.0.76.3.1.131.1.11.4 | |
| 5 | act | @codeSystem | 1..1 | F | LOINC: "2.16.840.1.113883.6.1" | |
| 4 | act | text | ED | 1..1 | R | textliche Beschreibung des codierten Inhalts der Observation, i.d.R. vom sendenden System automatisch generiert |
| 4 | act | effectiveTime | TS | 0..1 | O | Zeitpunkt der Feststellung des Phänomens, Das Datum, an dem das Phänomen festgestellt wurde |
| 4 | act | value | CD | 1..1 | R | Lokalisation |
| 5 | act | @code | 1..1 | R | eigentliche Lokalisation, zulässige Codesysteme: 2.16.840.1.113883.6.43.1 (ICD-O-3) | |
| 5 | act | @codeSystem | 1..1 | R | verwendetes Codesystem, vgl. @code | |
| 5 | act | originalText | ED | 0..1 | O | Freitext-Beschreibung der Lokalisation |
| 5 | act | qualifier | CR | 0..1 | O | |
| 6 | act | name | CV | 1..1 | R | |
| 7 | act | @code | 1..1 | F | Seitenlokalisation: "78615007" | |
| 7 | act | @codeSystem | 1..1 | F | SNOMED CT: "2.16.840.1.113883.6.96" | |
| 6 | act | value | CV | 1..1 | R | Seitenlokalisation |
| 7 | act | @code | 1..1 | R | eigentliche Seitenangabe, Codesystem 1.2.276.0.76.5.412 | |
| 7 | act | @codeSystem | 1..1 | F | "1.2.276.0.76.5.412" |
Es ist zu beachten, dass die Codierung der Lokalisationsangaben (Topographie) in ICD-O-3 zwar den ICD-10-Codes auf den ersten Blick stark ähnelt, jedoch grundlegende Unterschiede bestehen. Eine entsprechende Erläuterung findet sich im Abschnitt "Unterschiede zwischen der ICD-O und der ICD-10" der ICD-O-Klassifikation, die als PDF-Dokument bei DIMDI verfügbar ist. Dies gilt analog für den Tumorlokalisationsschlüssel, der auf einer früheren Version von ICD-O basiert.
Beispiel
Phänomen Primärtumor
Die Codierung erfolgt nach ICD-O-3 oder nach Tumorlokalisationsschlüssel. Eine Codierung nach TNM-Metastasenlokalisationen ist für Primärtumore nicht zulässig (sic!).
<section classCode="DOCSECT" moodCode="EVN">
<templateId root="1.2.276.0.76.3.1.131.1.10.2.6"/>
<code code="21855-2"
codeSystem="2.16.840.1.113883.6.1"/>
<title>Phänomen: Primärtumor</title>
<text>
<paragraph>
linke Brust, oben außen
</paragraph>
</text>
<entry>
<observation classCode="OBS" moodCode="EVN">
<id extension="prim12345"
root="1.2.276.0.76.3.1.131.1.4.3.9999.9999.999910"/>
<code code="21855-2"
codeSystem="2.16.840.1.113883.6.1"/>
<text>
linke Brust, oben außen
</text>
<effectiveTime value="20120525"/>
<value xsi:type="CD" code="C50.4"
codeSystem="2.16.840.1.113883.6.43.1">
<originalText>linke Brust, oben außen</originalText>
<qualifier>
<name code="78615007"
codeSystem="2.16.840.1.113883.6.96"/>
<value xsi:type="CD" code="L"
codeSystem="1.2.276.0.76.5.412"/>
</qualifier>
</value>
</observation>
</entry>
</section>
Phänomen Fernmetastase
Die Codierung kann hier nach jedem der oben genannten Codesysteme erfolgen.
<section classCode="DOCSECT" moodCode="EVN">
<templateId root="1.2.276.0.76.3.1.131.1.10.2.6"/>
<code code="21920-4"
codeSystem="2.16.840.1.113883.6.1"/>
<title>Phänomen: Fernmetastase</title>
<text>
<paragraph>
hepatisch
</paragraph>
</text>
<entry>
<observation classCode="OBS" moodCode="EVN">
<id extension="metast12345"
root="1.2.276.0.76.3.1.131.1.4.3.9999.9999.999910"/>
<code code="21920-4"
codeSystem="2.16.840.1.113883.6.1"/>
<text>
Fernmetastasen: HEP
</text>
<effectiveTime value="20120528"/>
<value xsi:type="CD" code="HEP"
codeSystem="1.2.276.0.76.5.401">
<originalText>
hepatisch
</originalText>
</value>
</observation>
</entry>
</section>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Section: Operation (operative Therapie)
| Template-Metadaten | |
| Template-Typ | Section |
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.7 ???? |
| generischeres Template | |
| genutztes Templates | |
| nutzende Templates | |
| abgeleitete Templates | |
| Generelle Beschreibung | In diesem Abschnitt werden die Daten über die Operation übermittelt. |
| allg. Erläuterung | |
| Verhältnis zu IHE | dt.Übersetzung oder Ergänzung oder neu |
| Ballotierungsstatus | |
| Erweiterbarkeit | offen oder geschlossen |
Beschreibung
Die Operation ist ein Eingriff zu therapeutischen (Beispiel: Ablation der Mamma) oder diagnostischen (Beispiel: Stanzbiopsie) Zwecken. Neben den durchgeführten Prozeduren sind hier auch die Intention der Therapie, die durchführenden Personen (Operateure) sowie die ggf. aufgetretenen Komplikationen von Bedeutung.
Eine operative Therapie kann aus mehreren Operationen bestehen. Hierbei wird für jede Operation ein Entry in der Section „Operative Therapie“ generiert.
Modell
Abbildung 23: Operation
Attribute
| Lvl | RIM | Name | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|
| 0 | act | section | 1..1 | required | Dieses Element enthält den Text. | |
| 1 | act | @classCode | ST | 1..1 | F | "DOCSECT" |
| 1 | act | @moodCode | ST | 1..1 | F | "EVN" |
| 1 | act | code | ST | 1..1 | required | Dieser Code definiert den Inhalt des Abschnittes. |
| 1 | act | title | ST | 1..1 | required | Dieses Element gibt die Überschrift des Abschnittes an. |
| 1 | act | text | ST | 1..1 | required | Dieses Element enthält den Text.
Hier werden alle strukturiert enthaltenen Daten der Section als Freitext übermittelt. Für Operationen ist zusätzlich die Übermittlung zusätzlicher unstrukturierter Informationen zulässig. |
| 1 | act | procedure | 1..1 | required | Dieses Element repräsentiert die gesamte (einzelne) Operation. | |
| 2 | act | @classCode | 1..1 | R | classCode <= PROC | |
| 2 | act | @moodCode | 1..1 | R | <= x_DocumentProcedureMood | |
| 2 | act | id | SET<II> | 1..* | required | Identifikation der Operation: Über dieses Element wird die Operation eindeutig identifiziert. Somit ist dieses Element required. Die Kombination der Attribute root und extension identifiziert die Operation systemübergreifend eindeutig. |
| 3 | act | @root | ST | 1..1 | required | Root-OID: Dieses Attribut ist ein eindeutiger Identifikator für Operationen innerhalb eines bestimmten sendenden Systems. |
| 3 | act | @extension | ST | 1..1 | required | Dieses Attribut ist innerhalb des sendenden Systems ein eindeutiger Identifikator für die Operation. |
| 3 | act | code | CD CWE | 0..1 | optional | durchgeführte Operation |
| 3 | act | text | ED | 0..1 | optional | Freitext |
| 3 | act | statusCode | ST | 1..1 | required | Über dieses Attribut wird der Status angegeben, in dem der beschriebene Act sich befindet. Ein Act kann z.B. den Status „new", „active" oder „cancelled" besitzen. Die Beobachtung einer Operation wird mit der Dokumentation als abgeschlossene Handlung betrachtet. Somit wird für das Attribut statusCode der feste Wert „completed" vorgegeben.<= completed |
| 3 | act | effectiveTime | IVL<TS> | 0..1 | optional | Durchführungszeitraum: Dieses Element gibt den Durchführungszeitraum der Therapie an. Benötigt wird Tagesgenauigkeit, bei einer Operation werden daher nicht Beginn und Ende angegeben, sondern das Datum im value-Attribut übermittelt. |
| 3 | rel | entryRelationship | 0..1 | optional | Intention: Ziel der Operation. | |
| 4 | act | Observation | 0..1 | optional | Intention: Ziel der Operation. | |
| 5 | act | code | CD CWE | 0..1 | Diese Klasse im Intent Mood drückt das Ziel aus. | |
| 5 | act | value | CD CWE | 0..1 | ||
| 3 | rel | entryRelationship | 0..1 | optional | Komplikation | |
| 4 | act | Observation | 0..1 | optional | Oft reicht in Bezug auf Komplikationen die einfache Information aus, ob diese aufgetreten sind. Dies wird als observation innerhalb einer entryRelationship übermittelt. | |
| 5 | act | code | 0..1 | optional | Der code für diese Observation ist fix. Es wird der entsprechende SNOMED-CT-Code verwendet. | |
| 5 | act | value | 0..1 | optional | Die Information, ob Komplikationen aufgetreten sind, wird als boolscher Wert übermittelt. Ist diese Information nicht bekannt, kann dies als nullFlavor="UNK" übermittelt werden. | |
| 3 | rel | entryRelationship | 0..1 | optional | Bestandteil: einzelne Prozedur | |
| 4 | act | procedure | 0..1 | optional | Einzelprozeduren: Eine Operation besteht häufig aus mehreren Einzelprozeduren. Jede Einzelprozedur wird als procedure in einer entryRelationship übermittelt. Beliebig viele entryRelationships sind zulässig. | |
| 5 | act | code | 0..1 | optional | Im code-Element werden die durchführten Einzelprozeduren übermittelt. Im Rahmen dieses Leitfadens wird der Operationen- und Prozedurenschlüssel (OPS) verwendet. Zulässig sind alle Fassungen von OPS. | |
| 3 | part | performer | 0..1 | optional | Ausführender: Hier werden die Angaben zu Operateur(en) übermittelt. Die Übermittlung mehrerer Operateure ist zulässig. | |
| 4 | role | AssignedEntity | 0..1 | optional | ausführender Arzt | |
| 5 | role | role | 0..1 | optional | Über dieses Element wird der Operateur eindeutig identifiziert. Somit ist dieses Element required. Die Kombination der Attribute root und extension identifiziert den Operateur systemübergreifend eindeutig. | |
| 5 | ent | person | 0..1 | optional | Arzt | |
| 6 | ent | name | PN.DE | 0..1 | optional | Name des Operateurs |
| 3 | part | participant | Teilnehmer | 0..1 | optional | ausführender Arzt |
| 4 | role | ParticipantRole | 0..1 | optional | sonstiger Teilnehmer | |
| 5 | ent | Person | 0..1 | optional | Teilnehmer |
Vokabular
| Lvl | Code | Bedeutung | Erläuterung |
|---|---|---|---|
| 1 | OP | Operation | 1), 2), 3), 4),bei Operativer Therapie Fixwert |
| 1 | RAD | Strahlentherapie | 1), 2), 3) |
| 2 | RAD-B | Brachytherapie | 4) ADT-Basisdatensatz unter¬scheidet hier in den Strahlen¬therapiedaten endokavitär und interstitiell |
| 2 | RAD-T | Teletherapie | 4) |
| 2 | RAD-S | sonstige Strahlentherapie | 4) |
| 1 | NUK | ||
| 2 | NUK-J | Radiojodtherapie | 4) |
| 2 | NUK-O | offene Radionuklide | 4) |
| 2 | NUK-S | sonstige Nuklearmedizinische Therapie | 4) |
| 1 | CHE | Chemotherapie | 1), 2), 3) |
| 1 | HOR | Hormontherapie / Antihormonelle Therapie | 1), 2), 3) |
| 1 | |||
| 2 | KNTR | Knochenmarktransplantation | 1), 2) |
| 2 | STAMM | Stammzelltransplantation | 2) |
| 3 | STAMM-L | Autologe Stammzelltransplantation | 4) |
| 3 | STAMM-G | Sonstige Stammzelltransplantation | 4) |
| 3 | STAMM-S | Allogene Stammzelltransplantation | 4) |
| 1 | IMM | Antikörper / Immuntherapie | 1), 2), 3) |
| 1 | SCHM | Schmerztherapie | 2) |
| 1 | PSY | Psychoonkologie | 2) |
| 1 | SM | Sonstige Medikamentöse Therapie | 4) |
| 1 | WS | Wait and see / Active surveillance | 4) Wait and see und Active Surveillance sind eigentlich deutlich verschieden und müssen in Prostata¬karzinom¬zentren unterschieden werden |
| 1 | ATH nullFlavour=OTH |
Sonstige / andere Therapie | 1), 2), 3) |
| 2 | ATH-HY | Hyperthermie | 4) |
Tabelle 23: Codesystem für Operationen
- GeKiD-Mindestdatensatz
- ADT Basisdatensatz „Verlaufsdaten" (HOR wurde dort vergessen, Hormontherapie ist aber auf dem Bogen)
- GKR (Gemeinsames Krebsregister)
- KRBW (Krebsregister Baden-Württemberg)
Problematisch ist:
- die unterschiedliche Tiefe , z.B. RAD allgemein vs. Differenzierung in unterschiedliche Subformen der Strahlen- und nuklearmedizinischen Therapie
- mangelnde Abbildung neuer medikamentöser (Zytokine, Signal-Transduktions-Inhibitoren, …) und bestimmter Untergruppen supportiver Therapie (z.B. Bisphosphonate)
- mangelnde Abbildung kombinierter Therapien, und damit die Diskussion prä- und post-koordinierter Ansatz .
KRBW: post-koordinierter Ansatz durch eine Kennzeichnung zusammengehöriger Therapien (MULTIMODALE_THERAPIE).
| Code | Bedeutung | Erläuterung |
|---|---|---|
| A | Abgelehnt | |
| V | vorgesehen | |
| T | Terminiert | |
| B | Begonnen | |
| R | regulär beendet | |
| vorzeitig beendet |
Tabelle 25: Codesystem für die Qualifier zu den Operationen
| Lvl | Code | Bedeutung | Erläuterung | |
|---|---|---|---|---|
| 1 | N | Nein | 1) (in der Wirklichkeit verbirgt sich hier eine große Zahl unterschiedlicher Negationen, die durchaus im Rahmen von Organzentren relevant sein können) | ??? |
| 2 | N-A | abgelehnt | 3) | rejected |
| 1 | V | vorgesehen | 2), 4) | statusCode=ActStatusNew |
| 2 | V-T | terminiert | 4) | moodCode=APT (Mood)??? |
| 1 | J | Ja | 1), 2) | statusCode=ActStatusNormal??? |
| 2 | J-B | begonnen | 4) (implizit aus leerem „Ende-Datum") | statusCode=ActStatusActive |
| 2 | J-E | regulär beendet | 3) | statusCode=ActStatusCompleted |
| 2 | J-A | vorzeitig beendet | 3), 5) (Abbruch wegen Nebenwirkungen) | statusCode=ActStatusAborted |
| 1 | U | Unbekannt | 1) | nullFlavor=UNK |
Tabelle 26: Codesystem für Operation statusCode
- GeKiD / GKR
- ADT- Basisdatensatz „Verlaufsdaten" (nur implizit)
- Information auf ADT- Basisdatensatz Systemisch und Strahlentherapie
- Information teilweise relevant für Kennzahlenberechnung in Organzentren
- KRBW
| Code | Bedeutung | Erläuterung |
|---|---|---|
| K | kurativ | |
| P | palliativ | |
| D | diagnostisch | nur bei Operationen |
| U | unbekannt bzw. nicht anwendbar / sonstige Therapie | besser nullFlavor |
Tabelle 27: Codesystem für Operation code
|
|
Diese Tabelle passt nicht zur Intention! |
Stellung der Therapie im Gesamtkonzept
| Code | Bedeutung | Erläuterung |
|---|---|---|
| N | neoadjuvant | |
| I | intraoperativ | (neuer Code, November 2010) |
| A | adjuvant | |
| U | unbekannt bzw. nicht anwendbar / sonstige Therapie | besser als nullFlavor |
Tabelle 28: Codesysstem für Operation code
|
|
Diese Tabelle passt nicht dazu! |
Beispeiel
<section>
<templateId root="1.2.276.0.76.3.1.131.1.10.2.7"/>
<code code="" codesystem="" />
<title>Operative Therapie</title>
<text>
Brusterhaltende Exzision der Mamma links am 31.03.2011, kurativ.<br>
Operateur: Dr. med. Max Meier PhD<br>
Partielle (brusterhaltende) Exzision der Mamma<br>
Sentinel-Lymphonodektomie mit Radionuklidmarkierung<br>
Komplikationen sind aufgetreten:<br>
<ul>
<il>subkutane Wundheilungsstörung, nicht revisionsbedürftig</il>
</ul>
</text>
<entry typeCode="DRIV">
<procedure classCode="PROC" moodCode="EVN">
<templateID root="??????"/>
<id extension="op12345"
root="1.2.276.0.76.3.1.131.1.4.3.9999.9999.999911"/>
<code code="OP" displayName="Operation"
codeSystem="1.2.276.0.76.3.1.131.1.5.14">
<qualifier>
<name code="IntentionOfTherapy"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="K"
codeSystem="1.2.276.0.76.3.1.131.1.5.18"/>
</qualifier>
</code>
<statusCode code="completed"/>
<effectiveTime value="20110331"/>
<performer typeCode="PRF">
<assignedEntity>
<id extension="54321"
root="1.2.276.0.76.3.1.131.1.4.3.9999.9999.999905"/>
<assignedPerson>
<name>
<prefix qualifier="AC">Dr. med.</prefix>
<given>Max</given>
<family>Meier</family>
<suffix qualifier="AC">PhD</suffix>
</name>
</assignedPerson>
</assignedEntity>
</performer>
<entryRelationship typeCode="COMP">
<procedure classCode="PROC" moodCode="EVN">
<code code="5-870.60" codeSystem="1.2.276.0.76.5.410"/>
</procedure>
</entryRelationship>
<entryRelationship typeCode="COMP">
<procedure classCode="PROC" moodCode="EVN">
<code code="5-401.11" codeSystem="1.2.276.0.76.5.410"/>
</procedure>
</entryRelationship>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="116224001" codeSystem="2.16.840.1.113883.6.96"/>
<value xsi:type="BL" value="true"/>
</observation>
</entryRelationship>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="116224001" codeSystem="2.16.840.1.113883.6.96"/>
<value xsi:type="CD" code="WSSnr"
codeSystem="1.2.276.0.76.3.1.131.1.5.15">
<originalText>
subkutane Wundheilungsstörung,
nicht revisionsbedürftig
</originalText>
</value>
</observation>
</entryRelationship>
</procedure>
</entry>
</section>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Section: Bestrahlung
| Template-Metadaten | |
| Template-Typ | Section |
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.12 |
| generischeres Template | |
| genutztes Templates | |
| nutzende Templates | |
| abgeleitete Templates | |
| Generelle Beschreibung | In diesem Abschnitt werden die Bestrahlungsdaten übermittelt. |
| allg. Erläuterung | |
| Verhältnis zu IHE | dt.Übersetzung oder Ergänzung oder neu |
| Ballotierungsstatus | |
| Erweiterbarkeit | offen oder geschlossen |
Beschreibung
 |
Ist das eine Section oder ein Entry? |
|
|
Maßnahmen &equal; Procedure Als Ergebnis oder als Maßnahme? |
Die Bestrahlung an sich ist eine Prozedur. Allerdings bestehen Beziehungen zu Phänomenen, die als Beobachtung kommuniziert werden.
Modell
Abbildung 24: Bestrahlung
Attribute
|
|
TODO |
| Lvl | RIM | Name | DT | Kard | Conf | Beschreibung | |
|---|---|---|---|---|---|---|---|
| 1 | act | observation | |||||
| 2 | act | templateID | |||||
| 2 | act | code | |||||
| 2 | act | value | |||||
| 2 | rel | ||||||
| 1 | part | ||||||
| 2 | role | ||||||
| 4 | ent |
Beispiel
|
|
falsches Beispiel !!! |
<observation classCode="OBS" moodCode="EVN">
<templateId root="2.16.840.1.113883.10.20.5.2.5.1"/>
<id root=".." extension=".." />
<code codeSystem="2.16.840.1.113883.6.1"
codeSystemName="LOINC"
code="41852-5"
displayName="Microorganism identified"/>
<value xsi:type="CD"
codeSystem="2.16.840.1.113883.6.96"
codeSystemName="SNOMED"
code="116197008"
displayName="Staphylococcus, coagulase negative (organism)"/>
<entryRelationship>
<observation>
<entryRelationship>
<observation>
</observation>
</entryRelationship>
</observation>
</entryRelationship>
</observation>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Section: Medikation
| Template-Metadaten | |
| Template-Typ | Section |
| Template ID | |
| generischeres Template | |
| genutztes Templates | |
| nutzende Templates | |
| abgeleitete Templates | |
| Generelle Beschreibung | In diesem Abschnitt wird die Daten zur Medikation übermittelt. |
| allg. Erläuterung | |
| Verhältnis zu IHE | dt.Übersetzung oder Ergänzung oder neu |
| Ballotierungsstatus | |
| Erweiterbarkeit | offen oder geschlossen |
|
|
Abgleich mit den anderen Medikation-Templates |
Modell
Abbildung 26: Medikation (gemäß IHE PCC)
|
|
Substance Administration Hinweis auf VHitG Addendum Medikation sowie epSOS!!! |
Attribute
| Lvl | RIM | Name | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|
| 1 | act | SubstanceAdministration | ||||
| 2 | act | @classCode | fix | SBADM | ||
| 2 | act | @moodCode | fix | ENV | ||
| 2 | act | templateID | fix | |||
| 2 | act | id | II | |||
| 2 | act | code | CD | |||
| 2 | part | consumable | ||||
| 2 | part | @typeCode | fix | CSM | ||
| 3 | role | manufacturedProduct | ||||
| 4 | ent | LabeledDrug | ||||
| 1 | act | |||||
| 2 | rel | |||||
| 1 | part | |||||
| 2 | role | |||||
| 4 | ent |
Beispiel
Aus dem VHitG-Arztbrief:
<substanceAdministration moodCode="EVN" classCode="SBADM" negationInd="true">
<templateId root="2.16.840.1.113883.10.20.5.6.42"/>
<consumable>
<manufacturedProduct classCode="MANU">
<manufacturedMaterial classCode="MMAT">
<code code="8611"
codeSystem="2.16.840.1.113883.6.88"
codeSystemName="RxNorm"
displayName="Povidone iodine"/>
</manufacturedMaterial>
</manufacturedProduct>
</consumable>
</substanceAdministration>
Aus IHE PCC TF 2:6.1.4.16.2
<substanceAdministration classCode="SBADM" moodCode="INT\|EVN">
<templateId root="2.16.840.1.113883.10.20.1.24"/>
<templateId root="1.3.6.1.4.1.19376.1.5.3.1.4.7"/>
<templateId root=""/>
<id root=\’\’ extension=\’\’/>
<code code=\’\’ codeSystem=\’\’ displayName=\’\’ codeSystemName=\’\’/>
<text><reference value=\’\#med-1\’/></text>
<statusCode code=\’completed\’/>
<effectiveTime xsi:type=\’IVL_TS\’>
<low value=\’\’/>
<high value=\’\’/>
</effectiveTime>
<effectiveTime operator=\’A\’ xsi:type=\’TS\|PIVL_TS\|EIVL_TS\|PIVL_PPD_TS\|SXPR_TS\’>
:
</effectiveTime>
<routeCode code=\’\’ codeSystem=\’\’ displayName=\’\’ codeSystemName=\’\’/>
<doseQuantity value=\’\’ unit=\’\’/>
<approachSiteCode code=\’\’ codeSystem=\’\’ displayName=\’\’ codeSystemName=\’\’/>
<rateQuantity value=\’\’ unit=\’\’/>
<consumable>
:
.
</consumable>
<!-- 0..\* entries describing the components -->
<entryRelationship typeCode=\’COMP\’ >
<sequenceNumber value=\’\’/>
</entryRelationship>
<!-- An optional entry relationship that indicates the the reason for use -->
<entryRelationship typeCode=\’RSON\’>
<act classCode=\’ACT\’ moodCode=\’EVN\’>
<templateId root=\’1.3.6.1.4.1.19376.1.5.3.1.4.4.1\’/>
<id root=\’\’ extension=\’\’/>
</act>
</entryRelationship>
<!-- An optional entry relationship that provides prescription activity -->
<entryRelationship typeCode=\’REFR\’>
<templateId root=\’1.3.6.1.4.1.19376.1.5.3.1.4.7.3\’/>
:
.
</entryRelationship>
<precondition>
<criterion>
<text><reference value=\’\’></text>
</criterion>
</precondition>
</substanceAdministation>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Status (Nachsorge und andere Follow-Up)
| Template ID | <OID für das Template> | |
| General Description | In diesem Abschnitt wird der Status zur Nachsorge und andere Follow-Ups übermittelt. | |
| LOINC Code | Opt. | Description |
| ???? | O | |
|
|
??? |
| Lvl | RIM | AE | Name | Desc | DT | Kard | Conf | Beschreibung
|
|---|---|---|---|---|---|---|---|---|
| 1 | act | elm | ||||||
| 2 | rel | elm | ||||||
| 1 | part | elm | ||||||
| 2 | role | elm | ||||||
| 4 | ent | elm |
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Section: Studiendaten
| Template-Metadaten | |
| Template-Typ | Section |
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.10 |
| generischeres Template | |
| genutztes Templates | |
| nutzende Templates | |
| abgeleitete Templates | |
| Schwester-Templates | |
| generelle Beschreibung | In diesem Abschnitt werden Studiendaten übermittelt. |
| allg. Erläuterung | |
| Verhältnis zu IHE | neu |
| Ballotierungsstatus | |
| Erweiterbarkeit | offen oder geschlossen |
|
|
Observation |
| Lvl | RIM | Name | Desc | DT | Kard | Conf | Beschreibung
|
|---|---|---|---|---|---|---|---|
| 1 | act | ||||||
| 2 | rel | ||||||
| 1 | part | ||||||
| 2 | role | ||||||
| 4 | ent |
Beispiel
|
|
Beispiel fehlt. |
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Section: Abschlussdaten
| Template-Metadaten | |
| Template-Typ | Section |
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.9 |
| generischeres Template | |
| genutztes Templates | |
| nutzende Templates | |
| abgeleitete Templates | |
| Schwester-Templates | |
| generelle Beschreibung | In diesem Abschnitt wird die Abschusdaten übermittelt |
| allg. Erläuterung | |
| Verhältnis zu IHE | neu |
| Ballotierungsstatus | |
| Erweiterbarkeit | offen oder geschlossen |
|
|
Section oder Entry? |
Attribute
|
|
Observation |
| Lvl | RIM | Name | Desc | DT | Kard | Conf | Beschreibung
|
|---|---|---|---|---|---|---|---|
| 1 | act | ||||||
| 2 | rel | ||||||
| 1 | part | ||||||
| 2 | role | ||||||
| 4 | ent |
Entry: Sterbedatum
|
|
TODO: Das Sterbedatum wird zur Zeit sowohl hier als auch im Abschnitt "Abschluss" vorgesehen. Es ist zu klären, was verwendet werden soll. Bis dahin wird dieser Entry nicht verwendet.
Eine Möglichkeit ist die Übernahme der Vorgabe aus IHE PCC. -> Prüfen Auf jeden Fall muss es als separates Entry-Level-Template ausgelagert und dann darauf referenziert werden. |
| Template ID | IHE PCC Health Status 1.3.6.1.4.1.19376.1.5.3.1.4.1.2 | |
| General Description | In diesem Abschnitt wird der Gesundheitszustand des Patienten übermittelt. | |
| LOINC Code | Opt. | Description |
| 11323-3 | O | Health Status |
Das Sterbedatum ist dann eine konkrete Beobachtung darin.
Vokabular
Die folgenden Snomed CT Codes (vgl. IHE PCC Vol.2 6.3.4.5.8) stehen zur Verfügung:
| Code | Description |
|---|---|
| 81323004 | Alive and well |
| 313386006 | In remission |
| 162467007 | Symptom free |
| 161901003 | Chronically ill |
| 271593001 | Severely ill |
| 21134002 | Disabled |
| 161045001 | Severely disabled |
| 419099009 | Deceased |
Beispiel
<entry>
<observation classCode='OBS' moodCode='EVN'>
<entryRelationship typeCode='REFR' inversionInd='false'>
<observation classCode='OBS' moodCode='EVN'>
<templateId root='2.16.840.1.113883.10.20.1.51'/>
<templateId root='1.3.6.1.4.1.19376.1.5.3.1.4.1.2'/>
<code code='11323-3' displayName='Health Status'
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC' />
<text><reference value='#hstatus-2'/></text>
<statusCode code='completed'/>
<value xsi:type='CE' code=' ' codeSystem='2.16.840.1.113883.6.96' codeSystemName='SNOMED CT'/>
</observation>
</entryRelationship>
</observation>
</entry>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Section: Todesursache
| Template ID | ????? | |
| General Description | In diesem Abschnitt wird die Todesursache übermittelt. | |
| LOINC Code | Opt. | Description |
| ???? | O | |
|
|
TODO: dieser Abschnitt muss noch überarbeitet werden. |
Die Übermittlung der Todesursache wird über eine separate Observation ausgedrückt, die aber in demselben Abschnitt (Section) untergebracht wird:
Abbildung 22: Diagnostik
| Lvl | RIM | Name | Desc | DT | Kard | Conf | Beschreibung
|
|---|---|---|---|---|---|---|---|
| 1 | act | Section | 0..1 | Dieser Abschnitt übermittelt Informationen über die Todesursache. | |||
| 2 | act | @classCode | DOCSECTC | CD CWE | 1..1 | M | |
| 2 | act | code | CD CWE | 1..1 | M | Dieser Code besagt, dass dieser Abschnitt über die Todesursache berichtet. | |
| 2 | act | title | 1..1 | M | |||
| 2 | act | text | 1..1 | M | |||
| 2 | rel | entry | |||||
| 3 | rel | @typeCode | DRIV | Der Text ist aus den Entry-Informationen abgeleitet. | |||
| 3 | act | Observation | Tod | ||||
| 4 | act | Observation | Tod durch Tumor | 1..1 | M | ||
| 5 | act | @code | Tod durch Tumor | CD CWE | 1..1 | M | Dieses Attribut drückt aus, dass diese Beobachtung Aufschluss über die Todesursache gibt. |
| 5 | act | @negationInd | Negation | BL | 0..1 | Wenn gesetzt drückt dieses Attribut aus, dass der Tumor nicht tumorbedingt ist/war. | |
| 5 | act | @value | Tod durch Tumor | CD CWE | 1..1 | M | Hier wird die Information (Code) zur Todesursache eingetragen: s.u. Codesystem zur Todesursache. |
| 4 | rel | entryRelationship | |||||
| 5 | rel | @typeCode | RSON | Es handelt sich um die Angabe der Todesursache (Grund). | |||
| 5 | act | Observation | Beruf | Spezialfall einiger EKR (z.B. GKR Gemeinsames Krebsregister, längster letzter Beruf, Dauer) => spätere Ausbauphase. Wird als Freitext und ggf. nach speziellem Berufe¬schlüssel übermittelt. | |||
| 6 | act | @code | Tod durch Beruf | CD CWE | 1..1 | M | Dieses Attribut drückt aus, dass diese Beobachtung angibt, ob der Tod ursächlich durch die Berufsausübung veranlasst ist. |
| 6 | act | @negationInd | Negation | BL | 0..1 | Wenn gesetzt drückt dieses Attribut aus, dass der Tumor nicht berufsbedingt ist/war. | |
| 6 | act | @value | Tod durch Beruf | BL | 1..1 | Ein boolescher Wert gibt an, ob der Krebs durch die Berufsausübung (Kap. 1.8.5) entstanden ist.
Wenn diese Aussage nicht gesichert (Verdacht) ist, dann wird dies über einen entsprechenden qualifier zum Ausdruck gebracht. Wenn nicht klar ist, ob die Berufsausübung ursächlich für den Krebs ist, dann ist dies durch einen nullFlavor auszudrücken. |
Vokabular
| Lvl | Code | Bedeutung | Erläuterung |
|---|---|---|---|
| 1 | J | Tod tumorbedingt, keine nähere Angabe | |
| 2 | T | Tod tumorbedingt durch das Tumorleiden, keine nähere Angabe | |
| 2 | P | Tod tumorbedingt durch Progression des primären Tumorgeschehens | |
| 2 | L | Tod tumorbedingt durch lokoregionäres Rezidiv | |
| 2 | M | Tod tumorbedingt durch Fernmetastasierung | |
| 2 | B | Tod tumorbedingt durch Behandlungskomplikation | |
| 1 | E | Entscheidung nicht möglich | |
| 1 | nullFlavor="NI" | „unbekannt" wird über einen nullFlavor zum Ausdruck gebracht. |
Tabelle 20: Vocabulary Domain für Todesursache (Kap 7.5)
(OID: ????)
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Section: Planung
| Template ID | ????? | |
| General Description | In diesem Abschnitt wird die Planung übermittelt. | |
| LOINC Code | Opt. | Description |
| ???? | O | |
Abbildung ??: Planungsdaten
|
|
Erklärung fehlt |
| Lvl | RIM | Name | Desc | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|---|
| 1 | act | ||||||
| 2 | rel | ||||||
| 1 | part | ||||||
| 2 | role | ||||||
| 4 | ent |
Beispiel
|
|
Beispiel fehlt |
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Anhang A: Diverses
|
|
Die meisten dieser Inhalte (d.h. Vokabularien aus Anhang B) sind noch als separate Templates aufzubereiten und dann zuerst auf die entsprechenden Seiten zu verschieben. Alternativ können diese als eigene Seiten ausgelagert werden! |
Offene Punkte
- vollständige Umsetzung
- Festlegung der Vokabularien
- Festlegung zur Übertragung (Transport) der Dokumente
- Validierung: BQS, Schemas, Schematron-Regeln, etc.
- Signatur
- Anonymisierung bzw. Pseudonymisierung (vgl. neue IHE-Profile)
- Abgleich mit IHE QRPH-ca (public reporting to cancer registries)
Referenzen/Literatur
| Kürzel | Inhalt |
|---|---|
| DIMDI, Alpha_Id | Alpha-ID - Die Identifikationsnummer |
| DIMDI, Verschl | Anleitung zur Verschlüsselung
http://www.dimdi.de/static/de/klassi/diagnosen/icd10/icdsgbv20.htm |
| DIMDI, FAQ OID | hilfreiche Antworten auf häufig auftretende Fragen zu OIDs. |
| DIMDI, Basis | Basiswissen Codieren, DIMDI 2004 |
| HL7 Datentypen | HL7 Version 3 Datentypen und CMETs für das Deutsche Gesundheitswesen, www.hl7.de (Publikationen) |
| CDAr2Arztbrief | Arztbrief auf Basis der HL7 Clinical Document Architecture Release 2 für das deutsche Gesundheitswesen, Version 1.50 vom 12.05.2006, herausgegeben vom VHitG, HL7 Deutschland und der Arbeitsgemeinschaft Sciphox, www.hl7.de (Publikationen) |
| Wiley, 2009 | TNM Classification of Malignant Tumours, 7th Edition
Editors: Sobin L, Gospodaarowicz M, Wittekind C Wiley-Blackwell, ISBN: 978-1-4443-3241-4, Dez. 2009 |
| Louis, 2007 | Louis et al. (2007) The 2007 WHO Classification of Tumours of the Central
Nervous System. Acta Neuropathol 114(2):97–110 |
Tabelle 29: Referenzen/Literatur
Glossar
| Abkürzung | Bedeutung |
|---|---|
| ADT | Arbeitsgemeinschaft Deutscher Tumorzentren e.V. |
| AQUA | Institut für angewandte Qualitätsförderung und Forschung im Gesund¬heitswesen GmbH |
| NCT | Nationales Centrum für Tumorerkrankungen Heidelberg |
| DKG | Deutsche Krebsgesellschaft e. V. |
| DOC | Deutsche Onkologie Centrum Holding GmbH |
| DokuData | DokuData Dokumentations- und Informationssysteme GmbH |
| GEKID | Gesellschaft der epidemiologischen Krebsregister in Deutschland e.V. |
| GTDS | Gießener Tumordokumentationssystem |
| HL7 | HL7 Benutzergruppe in Deutschland e.V. |
| ID | Information und Dokumentation im Gesundheitswesen GmbH & Co. KGa |
| IHE | IHE Deutschland e. V. |
| VHitG | Verband der Hersteller von IT-Lösungen für das Gesundheitswesen, e.V. |
Tabelle 30: Glossar
Detaillierte Änderungshistorie
| Version | Änderungen gegenüber Vorversion | Kapitel/Seitenzahl |
|---|---|---|
| 01 | Initialfassung mit grundlegender Struktur | Alle |
| 02 | weitere Ausarbeitungen | Alle |
| 03 | weitere Ausarbeitungen | alle |
| 04 | Einarbeitung der Kommentare von UA, BS, SF, CT | alle |
| 05 | weitere Ausarbeitung | alle |
| 06 | weitere Ausarbeitung | alle |
| 07 | weitere Ausarbeitung | alle |
| 08 | Überarbeitung gemäßt Beispiel | alle |
Tabelle 31: Änderungshistorie
Anhang B: Verzeichnisse
OID-Konzept der dt. Krebsgesellschaft
Codesysteme
Die OIDs für Codesysteme selbst sind auf einer separaten Seite zu finden. Dort werden zukünftig auch die entsprechenden Codelisten verfügbar sein. Bis dahin werden die in diesem Leitfaden verwendeten Codesysteme im Folgenden aufgeführt.
|
|
Ein Teil der verwendeten Codesysteme wird voraussichtlich zukünftig mit internationalen OIDs versehen sein. Es wird empfohlen, dies bei der Implementierung zu berücksichtigen. |
DKG-Labels
OID: 1.2.276.0.76.3.1.131.1.5.1
Dieses Codesystem unfasst die semantischen Identifikatoren für den Onkologie-Leitfaden. In der finalen Fassung werden voraussichtlich einige dieser Codes durch bereits existierende (z.B. LOINC Codes) ersetzt.
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| gekidMeldebegruendung | GEKID Meldebegründung | |
| gekidDiagnosesicherung | GEKID Diagnosesicherung | |
| gekidDiagnoseanlass | GEKID Diagnoseanlass | |
| gekidGrobstadium | GEKID Grobstadium | |
| fruehereChemotherapie | Hat eine frühere Chemotherapie stattgefunden? | Ja/nein |
| fruehereStrahlentherapie | Hat eine frühere Strahlentherapie stattgefunden? | Ja/nein |
| tnmT | TNM: T | Diagnoseleitfaden |
| tnmN | TNM: N | Diagnoseleitfaden |
| tnmM | TNM: M | Diagnoseleitfaden |
| tnmS | TNM: S | Diagnoseleitfaden |
| tnmL | TNM: L | Diagnoseleitfaden |
| tnmV | TNM: V | Diagnoseleitfaden |
| tnmPn | TNM: Pn | Diagnoseleitfaden |
| tnmStage | TNM: UICC-Stadium | Diagnoseleitfaden |
| tnmCp | TNM: c oder p (klinisch oder pathologisch) | Diagnoseleitfaden |
| tnmASymbol | TNM: a-Symbol | Diagnoseleitfaden |
| tnmRSymbol | TNM: r-Symbol | Diagnoseleitfaden |
| tnmYSymbol | TNM: y-Symbol | Diagnoseleitfaden |
| tnmCFactor | TNM: C-Faktor | Diagnoseleitfaden |
| tnmMSymbol | TNM: m-Symbol | Diagnoseleitfaden |
| tnmSn | TNM: Sentinel Node | Diagnoseleitfaden |
| tnmI | TNM: i | Diagnoseleitfaden |
| tnmMol | TNM: mol | Diagnoseleitfaden |
| tnmR | TNM: R | Diagnoseleitfaden |
| tnmRDomain | TNM: Gültigkeitsbereich der R-Klassifikation (lokal, gesamt) | |
| tnmRLocation | TNM: Lokalisation des Residualtumors | SnomedCT |
| SentinelNodesExamined | Anzahl untersuchter Sentinel-Lymphknoten | kein Kodesystem |
| SentinelNodesPositive | Anzahl befallener Sentinel-Lymphknoten | kein Kodesystem |
| Breslow | Breslow-Level (Tumordicke nach Breslow bei Hauttumoren) | Score |
| BreslowMM | Tumordicke nach Breslow in mm | kein Kodesystem |
| Enneking | Chirurgisches Staging nach Enneking 1986 (Weichteil-Sarkom) | Score |
| FIGO | FIGO-Klassifikation (gynäkologische Tumoren) | Score |
| Holoye | Holoye (Bronchialkarzinom) | |
| IGCCCG | IGCCCG-Prognose (Hodentumoren) | |
| Indiana | Indiana-Klassifikation (metastasierte Hodentumoren) | |
| INSS | INSS-Stadium (Neuroblastom) | |
| Lugano | Lugano-Klassifikation (Hodentumoren) | |
| Marburger | Marburger Klassifikation (kleinzelliges Bronchialkarzinom) | |
| WHOBrain | WHO-Stadium (Gehirntumoren) | |
| AnnArborStage | Ann Arbor Stadium (Hodgkin und Non-Hodgkin Lymphome) | Score |
| AnnArborBSymptoms | Ann Arbor B-Symptome (Hodgkin und Non-Hodgkin Lymphome) | Teil von Score |
| AnnArborExtranodal | Ann Arbor extranodaler Befall (Hodgkin und Non-Hodgkin Lymphome) | Teil von Score |
| Binet | Binet-Stadium (lymphatische Leukämien) | Score |
| DurieSalmon | Stadium nach Durie und Salmon (multiple Myelome) | Score |
| FAB | FAB-Klassifikation akuter myeloischer Leukämien | |
| Jansen | Stadium nach Jansen und Hermans (Haarzellleukämie) | Score |
| PhasenCML | Phasen für chronisch myeloische Leukämie (CML) | |
| Philadelphia | Philadelphia-Chromosom (CML) | |
| Radaszkiewicz | Stadium für Magenlymphome nach Radaszkiewicz | |
| RAI | RAI-Stadium (CLL) | Score |
| Robson | Stadium nach Robson (Nierenzellkarzinom) | Score |
| Murphy | Stadium nach Murphy (kindliches und jugendliches NHL) | Score |
| Kernohan | Kernohan-Klassifikation (Astrozytom) | |
| NWTS | NWTS-Klassifikation (Wilms-Tumor, Nephroblastom) | |
| Dukes | Dukes-Klassifikation (kolorektale Karzinome) | |
| VALG | VALG-Stadium (kleinzellige Bronchialkarzinome) | |
| RiskC58 | Risikokategorien bei trophoblastären Schwangerschaftstumoren (C58) | |
| Bismuth | Klassifikation der zentralen Gallengangskarzinome nach Bismuth und Corlette (1975) | |
| Borrmann | Borrmann-Klassifikation (Magenkarzinom) | |
| Fuhrman | Kerngrad nach Fuhrman (Nierenzellkarzinom) | |
| Hannover | Hannover-Klassifikation der Akustikusneurinome | |
| Helpap | Grading nach Helpap (Prostatakarzinom) | |
| Siewert | Siewert-Einteilung (Kardiakarzinom) | |
| VanNuysIndex | Van Nuys Prognoseindex (Mammakarzinom, DCIS) | |
| HistologieEinsendenummer | Einsendernummer des Histologiebefundes | |
| TypeOfEncounter | Art des Aufenthalts | |
| ReasonOfEncounter | Grund des Aufenthalts / der Untersuchung | |
| RemissionState | Remissionsstatus | |
| RemissionStateUnknownReason | Grund für unbekannten Remissionsstatus | |
| PrimarySiteStatus | Status des Primärtumors | |
| RegionalStatus | Status der regionären Lymphknoten | |
| DistantMetastasisStatus | Status der Fernmetastasen | |
| Clark | Clark-Level (Hauttumoren) | |
| TypeOfRadiationTherapy | Applikationsart der Strahlentherapie | |
| TotalDoseOfRadiationTherapy | Strahlentherapie: Gesamtdosis | |
| FractionDoseOfRadiationTherapy | Strahlentherapie: Einzeldosis | |
| IntentionOfTherapy | Therapieintention | |
| FinalStatusOfTherapy | Beendigungsstatus der Therapie | |
| Toxicity | Nebenwirkung | |
| ToxicityGrade | Nebenwirkungsgrad | |
| TherapyRegime | Therapieschema | |
| NumberOfPlannedTherapyCycles | Anzahl geplanter Therapiezyklen | |
| NumberOfAppliedTherapyCycles | Anzahl durchgeführter Therapiezyklen | |
| DoseReduction | Dosisreduktion | |
| ComplicationOccurred | Komplikation(en) ist/sind aufgetreten | |
| SurgeryRelationOfTherapy | zeitliche Relation der Therapie zur Operation | |
| StudyParticipation | Studienteilnahme des Patienten | |
| tnmMiSymbol | TNM mi-Symbol | |
| tnmMucosaSymbol | TNM Mucosa-Befall | |
| tnmSubmucosaSymbol | TNM Submucosa-Befall | |
| tnmMultiplicity | TNM Multiplizität (numerisch) | |
| residualIs | R-Klassifikation Suffix "(is)" | |
| residualCy+ | R-Klassifikation Suffix "(cy+)" | |
| residualUn | R-Klassifikation Suffix "(un)" | |
| residualGt1mm | R-Klassifikation Suffix ">1mm" | |
| residualLt1mm | R-Klassifikation Suffix "<1mm" | |
| residualDir | R-Klassifikation Suffix "-dir" |
GEKID Meldebegründung
OID: 1.2.276.0.76.3.1.131.1.5.2
Die genauen Formulierungen sind von Bundesland zu Bundesland verschieden. Die folgende Tabelle deckt aber alle Erfordernisse ab. Spezifische Teilmengen für die einzelnen Bundesländer sind über die entsprechenden Value Sets realisiert.
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| E | Patient ist informiert und hat eingewilligt | |
| I | Patient ist informiert und hat einer Kontaktaufnahme nicht widersprochen | |
| W | Patient ist informiert und hat einer Kontaktaufnahme widersprochen | |
| A | Ausnahme: Patient ist nicht informiert wegen möglicher gesundheitlicher Nachteile | |
| D | Meldung durch diagnostisch tätigen Arzt ohne unmittelbaren Patientenkontakt | |
| V | Patientenunterrichtung ist entfallen, da der Patient bereits verstorben ist |
GEKID Diagnosesicherung
OID: 1.2.276.0.76.3.1.131.1.5.3
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| 0 | Obduktion | |
| 1 | klinisch ohne spez. Diagnostik | |
| 2 | klinische Diagnostik | |
| 3 | Todesbescheinigung (DCO) | |
| 4 | spez. Tumormaker | |
| 5 | Zytologie | |
| 6 | Histologie Metastase | |
| 7 | Histologie Primärtumor | |
| 8 | Sonstige | |
| 9 | Unbekannt |
GEKID Diagnoseanlass
OID: 1.2.276.0.76.3.1.131.1.5.4
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| E | Eigenuntersuchung (Selbstuntersuchung) | |
| F | gesetzliche Früherkennung | |
| C | spezifische Screeningmaßnahme | |
| V | nicht gesetzliche Vorsorge | |
| A | Anamnese | |
| N | Nachsorge | |
| T | Tumorsymptomatik | |
| U | Unbekannt |
GEKID Grobstadium
OID: 1.2.276.0.76.3.1.131.1.5.5
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| I | in situ | |
| L | lokal | |
| R | regional | |
| F | Fernmetastasen | |
| S | Systemerkrankung | |
| U | unbekannt |
Art des Krankenhausaufenthalts
OID: 1.2.276.0.76.3.1.131.1.5.6
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| A | ambulant | |
| S | stationär |
Allgemeiner Leistungszustand, ECOG
OID: 1.2.276.0.76.3.1.131.1.5.7
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| 0 | Normale, uneingeschränkte körperliche Aktivität | |
| 1 | Mäßig eingeschränkte körperliche Aktivität u. Arbeitsfähigkeit, nicht bettlägerig | |
| 2 | Arbeitsunfähig, meist selbstständige Lebensführung, wachsendes Ausmaß an Pflege und Unterstützung notwendig, weniger als 50 % bettlägerig | |
| 3 | Weitgehend unfähig, sich selbst zu versorgen, kontinuierliche Pflege oder Hospitalisierung notwendig, rasche Progredienz des Leidens, mehr als 50 % bettlägerig | |
| 4 | 100 % bettlägerig, völlig pflegebedürftig | |
| 5 | Tod |
TLS - Tumorlokalisationsschlüssel (ICD-O-2 deutsche Erweiterung)
OID: 1.2.276.0.76.3.1.131.1.5.8
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| C00.0 | Oberlippe, Lippenrot (ohne Haut der Oberlippe, = C44.01) | |
| C00.1 | Unterlippe, Lippenrot (ohne Haut der Unterlippe, = C44.02) | |
| C00.2 | Lippenrot, o.n.A. | |
| C00.3 | Schleimhaut der Oberlippe | |
| C00.4 | Schleimhaut der Unterlippe | |
| C00.5 | Lippenschleimhaut o.n.A. | |
| C00.6 | Lippenkommissur, Mundwinkel | |
| C00.8 | Lippen (mehrere Teilbereiche überlappend) | |
| C00.9 | Lippe o.n.A. (ohne Haut der Lippen, = C44.0) | |
| C01.9 | Zungengrund (hinteres Zungendrittel, Zungenwurzel) | |
| C02.0 | Zungenrücken (vordere 2/3 des Zungenrückens) | |
| C02.1 | Zungenrand, Zungenspitze | |
| C02.2 | Zungenunterfläche, Zungenfrenulum | |
| C02.3 | Zunge, vordere 2/3, o.n.A. | |
| C02.4 | Zungentonsille | |
| C02.8 | Zunge (mehrere Teilbereiche, Verbindungszone) | |
| C02.9 | Zunge o.n.A. | |
| C03.0 | Oberkieferschleimhaut (Mundschleimhaut) | |
| C03.01 | Gingiva im Oberkiefer (Mundschleimhaut) | |
| C03.02 | Schleimhaut des Alveolarfortsatzes im Oberkiefer (Mundschleimhaut) | |
| C03.03 | Alveole im Oberkiefer (Mundschleimhaut) | |
| C03.1 | Unterkieferschleimhaut (Mundschleimhaut) | |
| C03.11 | Gingiva im Unterkiefer (Mundschleimhaut) | |
| C03.12 | Schleimhaut des Alveolarfortsatzes im Unterkiefer (Mundschleimhaut) | |
| C03.13 | Alveole im Unterkiefer (Mundschleimhaut) | |
| C03.8 | Mundschleimhaut (mehrere Teilbereiche) | |
| C03.9 | Mundschleimhaut o.n.A., parodontales Gewebe | |
| C04.0 | Vorderer Mundboden | |
| C04.1 | Seitlicher Mundboden | |
| C04.8 | Mundboden (mehrere Teilbereiche überlappend) | |
| C04.9 | Mundboden o.n.A. | |
| C05.0 | Harter Gaumen | |
| C05.1 | Weicher Gaumen (ohne Nasopharynx-Anteil,= C11.3) | |
| C05.2 | Uvula | |
| C05.8 | Gaumen (mehrere Teilbereiche überlappend) | |
| C05.9 | Gaumen o.n.A. | |
| C06.0 | Wangenschleimhaut | |
| C06.1 | Vestibulum oris | |
| C06.11 | Sulcus alveolaris | |
| C06.12 | Sulcus buccomaxillaris | |
| C06.13 | Sulcus buccomandibularis | |
| C06.2 | Retromolare Zone, Trigonum retromolare | |
| C06.8 | Mundhöhle (mehrere Teilbereiche überlappend) | |
| C06.9 | Mund o.n.A., Mundhöhle o.n.A., kleine Speicheldrüsen o.n.A. | |
| C07.9 | Parotis (einschl. Stensen-Gang) | |
| C08.0 | Gl. submandibularis (einschl. Wharton-Gang) | |
| C08.1 | Gl. sublingualis (mit Ausführungsgang) | |
| C08.8 | Große Speicheldrüsen (mehrere Teilbereiche überlappend) | |
| C08.9 | Große Speicheldrüsen o.n.A. | |
| C09.0 | Tonsillennische | |
| C09.1 | Gaumenbogen | |
| C09.8 | Tonsille (mehrere Teilbereiche überlappend) | |
| C09.9 | Gaumentonsille, Tonsille o.n.A. | |
| C10.0 | Vallecula epiglottica (ohne Zungengrund, = C01.9) | |
| C10.1 | Vordere, linguale Epiglottisfläche | |
| C10.2 | Oropharynx, Seitenwand (auch Glossotonsillarfurche) | |
| C10.3 | Oropharynx, Hinterwand | |
| C10.4 | Branchiogene Fistel | |
| C10.8 | Oropharynx (mehrere Teilbereiche überlappend) | |
| C10.9 | Oropharynx o.n.A. | |
| C11.0 | Nasopharynx, Dach | |
| C11.1 | Nasopharynx, Hinterwand (einschl. Rachentonsille) | |
| C11.2 | Nasopharynx, Seitenwand (einschl. Rosenmüller-Grube) | |
| C11.3 | Nasopharynx,Vorderwand (einschl. Choanen und nasopharyngeale Fläche des Gaumens) | |
| C11.8 | Nasopharynx (mehrere Teilbereiche überlappend) | |
| C11.9 | Nasopharynx o.n.A. | |
| C12.9 | Sinus piriformis | |
| C13.0 | Postkrikoidbezirk, Krikoid o.n.A. | |
| C13.1 | Aryepiglottische Falte (ohne Larynx-Anteil, = C32.1) | |
| C13.2 | Hypopharynxhinterwand | |
| C13.8 | Hypopharynx (mehrere Teilbereiche überlappend) | |
| C13.9 | Hypopharynx o.n.A. | |
| C14.0 | Pharynx o.n.A., Retropharynx | |
| C14.1 | Laryngopharynx | |
| C14.2 | Waldeyer-Ring | |
| C14.8 | Mundbereich (mehrere Teilbereiche überlappend) | |
| C15.0 | Zervikaler Oesophagus | |
| C15.1 | Thorakaler Oesophagus | |
| C15.2 | Abdominaler Oesophagus | |
| C15.3 | Oesophagus, oberes intrathorakales Drittel | |
| C15.4 | Oesophagus, mittleres intrathorakales Drittel | |
| C15.5 | Oesophagus, unteres intrathorakales Drittel | |
| C15.8 | Oesophagus (mehrere Teilbereiche überlappend) | |
| C15.9 | Oesophagus o.n.A. | |
| C16.0 | Kardia (oesophago-kardialer Übergang) | |
| C16.1 | Fundus, Magen | |
| C16.11 | Fundus (kleine Kurvatur), Magen | |
| C16.12 | Fundus (Vorderwand), Magen | |
| C16.13 | Fundus (große Kurvatur), Magen | |
| C16.14 | Fundus (Hinterwand), Magen | |
| C16.2 | Korpus, Magen | |
| C16.21 | Korpus (kleine Kurvatur), Magen | |
| C16.22 | Korpus (Vorderwand), Magen | |
| C16.23 | Korpus (große Kurvatur), Magen | |
| C16.24 | Korpus (Hinterwand), Magen | |
| C16.3 | Antrum | |
| C16.31 | Antrum (kleine Kurvatur, Angulus) | |
| C16.32 | Antrum (Vorderwand) | |
| C16.33 | Antrum (große Kurvatur) | |
| C16.34 | Antrum (Hinterwand) | |
| C16.4 | Pylorus | |
| C16.42 | Pylorus (Vorderwand) | |
| C16.44 | Pylorus (Hinterwand) | |
| C16.5 | Kleine Kurvatur o.n.A. | |
| C16.6 | große Kurvatur o.n.A. | |
| C16.8 | Magen (mehrere Teilbereiche überlappend) | |
| C16.9 | Magen o.n.A. | |
| C17.0 | Duodenum | |
| C17.01 | Duodenum, Pars superior | |
| C17.02 | Duodenum, Pars descendens | |
| C17.03 | Duodenum, Pars horizontalis (inferior) | |
| C17.04 | Duodenum, Pars ascendens | |
| C17.1 | Jejunum | |
| C17.2 | Ileum (ohne Ileozaekalklappe, = C18.0) | |
| C17.3 | Meckel-Divertikel | |
| C17.8 | Dünndarm (mehrere Teilbereiche überlappend) | |
| C17.9 | Dünndarm o.n.A. | |
| C18.0 | Zaekum (einschl.Ileozaekalklappe) | |
| C18.1 | Appendix | |
| C18.2 | Colon ascendens | |
| C18.3 | Flexura hepatica | |
| C18.4 | Colon transversum | |
| C18.41 | Colon transversum, rechtes Drittel | |
| C18.42 | Colon transversum, mittleres Drittel | |
| C18.43 | Colon transversum, linkes Drittel | |
| C18.5 | Flexura lienalis | |
| C18.6 | Colon descendens | |
| C18.7 | Colon sigmoideum (ohne Rektosigmoid, Übergang, = C19.9) | |
| C18.8 | Dickdarm (mehrere Teilbereiche überlappend) | |
| C18.9 | Dickdarm o.n.A. | |
| C19.9 | Rektosigmoid, Übergang sowie Kolon und Rektum | |
| C20.9 | Rektum o.n.A. (Ampulle) | |
| C20.91 | Rektum 4 bis < 7,5 cm Höhe | |
| C20.92 | Rektum 7,5 bis < 12 cm Höhe | |
| C20.93 | Rektum 12 cm und mehr aufwärts | |
| C21.0 | Anus o.n.A. (ohne Anus, äußere Haut = C44.5) | |
| C21.1 | Analkanal, Analsphinkter | |
| C21.2 | Kloakenregion | |
| C21.8 | Mehrere Teilbereiche von Anus und Analkanal | |
| C22.0 | Leber | |
| C22.01 | Rechter Leberlappen | |
| C22.02 | Linker Leberlappen | |
| C22.1 | Intrahepatische Gallengänge (Gallenkanälchen) | |
| C23.9 | Gallenblase | |
| C23.91 | Gallenblasenhals | |
| C23.92 | Gallenblasenkörper | |
| C23.93 | Gallenblasenfundus | |
| C24.0 | Extrahepatische Gallengänge | |
| C24.01 | Rechter Ductus hepaticus | |
| C24.02 | Linker Ductus hepaticus | |
| C24.03 | Ductus hepaticus communis | |
| C24.04 | Ductus choledochus | |
| C24.05 | Ductus cysticus | |
| C24.1 | Ampulla Vateri | |
| C24.8 | Gallengänge (mehrere Teilbereiche überlappend) | |
| C24.9 | Gallengänge o.n.A. | |
| C25.0 | Pankreaskopf | |
| C25.1 | Pankreaskörper | |
| C25.2 | Pankreasschwanz | |
| C25.3 | Ductus pankreaticus | |
| C25.4 | Langerhans-Inseln | |
| C25.7 | Andere Teile des Pankreas | |
| C25.8 | Pankreas (mehrere Teilbereiche überlappend) | |
| C25.9 | Pankreas o.n.A. | |
| C26.0 | Darm o.n.A. | |
| C26.8 | Tumoren der Verdauungsorgane, deren Ursprungsort keiner d. Rubriken C15-C26.0 zugeordnet werden kann | |
| C26.9 | Verdauungstrakt o.n.A. | |
| C30.0 | Innere Nase, Nasenhöhle (einschl. Knorpel, Schleimhaut,...) | |
| C30.1 | Mittel- und Innenohr (einschl. Mastoid, Tube u. Paukenhöhle) | |
| C31.0 | Kieferhöhle | |
| C31.1 | Siebbeinhöhle | |
| C31.2 | Stirnhöhle | |
| C31.3 | Keilbeinhöhle | |
| C31.8 | Nebenhöhlen (mehrere Teilbereiche überlappend) | |
| C31.9 | Nebenhöhlen o.n.A. | |
| C32.0 | Glottis, Stimmband, Kommissur | |
| C32.1 | Supraglottis, Taschenbänder | |
| C32.2 | Subglottis | |
| C32.3 | Larynxknorpel | |
| C32.8 | Larynx (mehrere Teilbereiche überlappend) | |
| C32.9 | Larynx o.n.A. | |
| C33.9 | Trachea | |
| C34.0 | Hauptbronchus | |
| C34.01 | Carina | |
| C34.02 | Zwischenbronchus | |
| C34.1 | Lungenoberlappen (einschl. Lingula u. Oberlappenbronchus) | |
| C34.2 | Lungenmittellappen (einschl.Mittellappenbronchus) | |
| C34.3 | Lungenunterlappen (einschl. Unterlappenbronchus) | |
| C34.8 | Lunge (mehrere Teilbereiche überlappend) | |
| C34.9 | Lunge o.n.A, Bronchus o.n.A. | |
| C37.9 | Thymus | |
| C38.0 | Herz | |
| C38.1 | Vorderes Mediastinum | |
| C38.2 | Hinteres Mediastinum | |
| C38.3 | Mediastinum o.n.A. | |
| C38.4 | Pleura o.n.A. | |
| C38.41 | Pleura parietalis | |
| C38.42 | Pleura visceralis | |
| C38.8 | Herz, Mediastinum und Pleura (mehrere Teilber.überlappend) | |
| C39.0 | Oberer Respirationstrakt o.n.A. | |
| C39.8 | Tumoren des Respirationstraktes, deren Ausgangspunkt nicht C30 - C39.0 zugeordnet werden kann | |
| C39.9 | Respirationstrakt o.n.A. | |
| C40.0 | Lange Knochen von Arm und Schulter und zugehörige Gelenke | |
| C40.01 | Scapula | |
| C40.02 | Humerus | |
| C40.03 | Radius | |
| C40.04 | Ulna | |
| C40.05 | Schultergelenk | |
| C40.06 | Ellenbogengelenk | |
| C40.07 | Radioulnargelenk | |
| C40.08 | Akromioklavikurlargelenk | |
| C40.1 | Kurze Knochen der oberen Extremitäten und zugehörige Gelenke | |
| C40.11 | Carpalia | |
| C40.12 | Metacarpalia | |
| C40.13 | Phalangen der Finger | |
| C40.14 | Handgelenke | |
| C40.15 | Daumengrundgelenk | |
| C40.16 | Fingergrundgelenk | |
| C40.17 | Mittel- und Endgelenke der Finger | |
| C40.2 | Lange Knochen der unteren Extremitäten und zugehörige Gelenke | |
| C40.21 | Femur | |
| C40.22 | Tibia | |
| C40.23 | Fibula | |
| C40.24 | Tibiofibulargelenk | |
| C40.25 | Kniegelenk | |
| C40.26 | Meniskus, medialer | |
| C40.27 | Meniskus, lateraler | |
| C40.3 | Kurze Knochen der unteren Extremitäten und zugehörige Gelenke | |
| C40.31 | Calcaneus | |
| C40.32 | Andere Tarsalia (Tarsus) | |
| C40.33 | Metatarsalia (Metatarsus) | |
| C40.34 | Zehenphalangen | |
| C40.35 | Patella | |
| C40.36 | Sprunggelenke | |
| C40.37 | Andere Fußwurzelgelenke | |
| C40.38 | Tarsometatarsalgelenke | |
| C40.39 | Zehengelenke | |
| C41.0 | Knochen des Hirn- und Gesichtsschädels (ausschließl. Mandibula, = C41.1) | |
| C41.01 | Knochen des Hirnschädels | |
| C41.02 | Schädelbasis | |
| C41.03 | Knochen des Gesichtsschädels | |
| C41.04 | Nasenbein | |
| C41.05 | Maxilla | |
| C41.06 | Jochbein (Os zygomaticum) | |
| C41.07 | Siebbein (Os ethmoidale) | |
| C41.08 | Keilbein (Os sphenoidale) | |
| C41.09 | Zungenbein (Os hyoidale) | |
| C41.1 | Mandibula | |
| C41.11 | Kiefergelenk | |
| C41.2 | Wirbelsäule (ohne Kreuzbein, = C41.45 und Steißbein, = C41.46) | |
| C41.21 | Halswirbel | |
| C41.22 | Brustwirbel | |
| C41.23 | Lendenwirbel | |
| C41.24 | Halswirbelsäule, Diskus | |
| C41.25 | Brustwirbelsäule, Diskus | |
| C41.26 | Lendenwirbelsäule, Diskus | |
| C41.3 | Thoraxskelett mit Klavikula und zugehörige Gelenke | |
| C41.31 | Rippen, knöcherner Anteil | |
| C41.32 | Rippenknorpel | |
| C41.33 | Sternum | |
| C41.34 | Klavikula | |
| C41.35 | Kostovertebralgelenk | |
| C41.36 | Sternokostalgelenk | |
| C41.37 | Sternoklavikulargelenk | |
| C41.4 | Beckenknochen, Kreuzbein, Steißbein und zugehörige Gelenke | |
| C41.41 | Os ilium | |
| C41.42 | Os pubis | |
| C41.43 | Os ischii | |
| C41.44 | Os sacrum | |
| C41.45 | Os coccygeum | |
| C41.46 | Acetabulum, Hüftgelenk | |
| C41.47 | Iliosakralgelenk | |
| C41.48 | Symphysis pubica | |
| C41.8 | Knochen, Gelenke und Gelenkknorpel, die nicht C40-C41.4 zuordenbar | |
| C41.9 | Knochen, Gelenke und Gelenkknorpel o.n.A. | |
| C42.0 | Blut | |
| C42.1 | Knochenmark | |
| C42.2 | Milz | |
| C42.3 | Retikuloendotheliales System o.n.A. | |
| C42.4 | Haematopoetisches System o.n.A. | |
| C44.0 | Lippenhaut | |
| C44.01 | Oberlippe, äußere Haut | |
| C44.02 | Unterlippe, äußere Haut | |
| C44.03 | Mundwinkel, Aussenseite (Haut, Innenseite = C00.6) | |
| C44.1 | Augenlid (Haut) | |
| C44.11 | Oberlid (Haut) | |
| C44.12 | Unterlid (Haut) | |
| C44.13 | Innerer Augenwinkel (Haut) | |
| C44.14 | Äußerer Augenwinkel (Haut) | |
| C44.15 | Meibom-Drüse (Haut) | |
| C44.2 | Äußeres Ohr (Haut) | |
| C44.21 | Ohrmuschel (Haut) | |
| C44.22 | Äußerer Gehörgang (Haut) | |
| C44.3 | Andere Teile der Gesichtshaut | |
| C44.31 | Nase (Haut) (ohne Nasenschleimhaut, = C30.0) | |
| C44.32 | Wange (Haut) (ohne Wangenschleimhaut, = C06.0) | |
| C44.33 | Stirn, Augenbrauen (Haut) | |
| C44.34 | Schläfe (Haut) | |
| C44.35 | Kinn (Haut) | |
| C44.36 | Kieferwinkel (Haut) | |
| C44.4 | Haut von behaartem Kopf und Hals (einschl. supraklavikulärer Region) | |
| C44.41 | Behaarter Kopf (Haut) | |
| C44.42 | Nacken (Haut) | |
| C44.43 | Hals (Haut) | |
| C44.44 | Supraklavikulaere Region (Haut) | |
| C44.5 | Haut des Stammes | |
| C44.51 | Vordere und seitliche Brustwand (Haut) | |
| C44.52 | Bauchhaut (einschl. Haut des Nabels) | |
| C44.53 | Rückenhaut | |
| C44.54 | Gesäßhaut | |
| C44.55 | Analrand (Perianale Haut) | |
| C44.56 | Leistenbeuge (Haut) | |
| C44.57 | Genitokrural-Beuge (Haut) | |
| C44.58 | Damm (Haut) | |
| C44.6 | Haut von Arm und Schulter | |
| C44.61 | Schulter (Haut) | |
| C44.62 | Axilla (Haut) | |
| C44.63 | Oberarm (Haut) | |
| C44.64 | Ellenbogen, Ellenbeuge (Haut) | |
| C44.65 | Unterarm (Haut) | |
| C44.66 | Handrücken (Haut) | |
| C44.67 | Handinnenfläche (Haut) | |
| C44.68 | Finger (Haut) | |
| C44.69 | Subungualgegend (Hand) | |
| C44.7 | Haut von Bein und Hüfte | |
| C44.71 | Hüfte (Haut) | |
| C44.72 | Oberschenkel (Haut) | |
| C44.73 | Knie, Kniekehle (Haut) | |
| C44.74 | Unterschenkel (Haut) | |
| C44.75 | Fußrücken (Haut) | |
| C44.76 | Fußsohle (Haut) | |
| C44.77 | Ferse (Haut) | |
| C44.78 | Zehen (Haut) | |
| C44.79 | Subungualgegend (Fuß) | |
| C44.8 | Haut (mehrere Regionen überlappend) | |
| C44.9 | Haut o.n.A. | |
| C47.0 | Periphere Nerven u. autonomes Nervensystem im Kopf-Hals-Bereich | |
| C47.1 | Periphere Nerven u. autonomes Nervensystem der oberen Extremität und der Schulter | |
| C47.2 | Periphere Nerven u. autonomes Nervensystem der unteren Extremität und der Hüfte | |
| C47.3 | Periphere Nerven u. autonomes Nervensystem des Thorax | |
| C47.4 | Periphere Nerven u. autonomes Nervensystem des Abdomens | |
| C47.5 | Periphere Nerven u. autonomes Nervensystem des Beckens | |
| C47.6 | Periphere Nerven u. autonomes Nervensystem des Stamms | |
| C47.8 | Periphere Nerven u. autonomes Nervensystem (mehrere Teilbereiche überlappend) | |
| C47.9 | Autonomes Nervensystem o.n.A. | |
| C48.0 | Retroperitoneum | |
| C48.01 | Periadrenales Gewebe | |
| C48.02 | Perirenales Gewebe | |
| C48.03 | Peripankreatisches Gewebe | |
| C48.04 | Retrozaekales Gewebe | |
| C48.1 | Peritoneum | |
| C48.11 | Peritoneum parietale | |
| C48.12 | Peritoneum viscerale | |
| C48.13 | Omentum majus | |
| C48.14 | Dünndarm-Mesenterium | |
| C48.15 | Appendix-Mesenterium | |
| C48.16 | Mesokolon | |
| C48.17 | Douglas-Raum | |
| C48.2 | Peritonealhöhle o.n.A. | |
| C48.8 | Peritoneum und Retroperitoneum (mehrere Teilbereiche überlappend) | |
| C49.0 | Bindegewebe, Subkutane und andere Weichteilgewebe von Kopf und Hals | |
| C49.01 | Kopfschwarte (Bindegewebe, Weichteile) | |
| C49.02 | Schläfe (Bindegewebe, Weichteile) | |
| C49.03 | Stirn (Bindegewebe, Weichteile) | |
| C49.04 | Mittelgesicht (Bindegewebe, Weichteile) | |
| C49.05 | Kinn (Bindegewebe, Weichteile) | |
| C49.06 | Hals und Nacken (Bindegewebe, Weichteile) | |
| C49.07 | Supraklavikulaere Region (Bindegewebe, Weichteile) | |
| C49.1 | Bindegewebe, subkutane und andere Weichteilgewebe der oberen Extremität und Schulter | |
| C49.11 | Schulter (Bindegewebe, Weichteile) | |
| C49.12 | Oberarm (Bindegewebe, Weichteile) | |
| C49.13 | Ellenbogen (Bindegewebe, Weichteile) | |
| C49.14 | Ellenbeuge (Bindegewebe, Weichteile) | |
| C49.15 | Unterarm (Bindegewebe, Weichteile) | |
| C49.16 | Handgelenk (Bindegewebe, Weichteile) | |
| C49.17 | Mittelhand (Bindegewebe, Weichteile) | |
| C49.18 | Finger (Bindegewebe, Weichteile) | |
| C49.2 | Bindegewebe, subkutane und andere Weichteilgewebe der unteren Extremitaet und Hüfte | |
| C49.21 | Hüfte (Bindegewebe, Weichteile) | |
| C49.22 | Oberschenkel (Bindegewebe, Weichteile) | |
| C49.23 | Knie (Bindegewebe, Weichteile) | |
| C49.24 | Kniekehle (Bindegewebe, Weichteile) | |
| C49.25 | Unterschenkel (Bindegewebe, Weichteile) | |
| C49.26 | Sprunggelenk (Bindegewebe, Weichteile) | |
| C49.27 | Fuß (Bindegewebe, Weichteile) | |
| C49.28 | Zehen (Bindegewebe, Weichteile) | |
| C49.3 | Bindegewebe, subkutane und andere Weichteilgewebe des Thorax | |
| C49.31 | Aorta thoracalis (Bindegewebe, Weichteile) | |
| C49.32 | Vena cava superior (Bindegewebe, Weichteile) | |
| C49.33 | Axilla (Bindegewebe, Weichteile) | |
| C49.34 | Diaphragma | |
| C49.36 | Muskeln im Thoraxbereich | |
| C49.37 | Gefäße im Thoraxbereich (außer Aorta u. V.cava sup.) | |
| C49.38 | Ductus thoracicus | |
| C49.4 | Bindegewebe, subkutane und andere Weichteilgewebe des Bauches | |
| C49.41 | Aorta abdominalis | |
| C49.42 | Vena cava inferior, Vena cava o.n.A. | |
| C49.43 | Arterien des Bauchraumes | |
| C49.44 | Venen des Bauchraumes | |
| C49.45 | Nabel (Bindegewebe) | |
| C49.46 | Bauchdeckenmuskulatur | |
| C49.5 | Bindegewebe, subkutane und andere Weichteilgewebe des Beckens | |
| C49.51 | Iliakalarterien | |
| C49.52 | Iliakalvenen | |
| C49.54 | Bindegewebe der Leistengegend | |
| C49.55 | Bindegewebe des Perineums | |
| C49.56 | Bindegewebe des Gesäßes | |
| C49.57 | Bindegewebe der Steißregion | |
| C49.58 | M. gluteus maximus (Muskel) | |
| C49.6 | Bindegewebe, subkutane und andere Weichteilgewebe des Stamms | |
| C49.61 | Rücken (Bindegewebe, Weichteile) | |
| C49.62 | Flanke (Bindegewebe, Weichteile) | |
| C49.63 | Stamm (Bindegewebe, Weichteile) | |
| C49.8 | Bindegewebe, subkutane und andere Weichteilgewebe mehrerer Regionen überlappend | |
| C49.9 | Bindegewebe und Weichteilgewebe o.n.A. | |
| C49.91 | Bindegewebe o.n.A. | |
| C49.92 | Sehnen, Bänder, Faszien, Aponeurosen o.n.A. | |
| C49.93 | Schleimbeutel, Gelenkkapseln, Sehnenscheiden o.n.A. | |
| C49.94 | Arterien o.n.A. | |
| C49.95 | Venen o.n.A. | |
| C49.96 | Muskeln o.n.A. | |
| C49.97 | Fettgewebe o.n.A. | |
| C49.98 | Lymphatisches Gewebe o.n.A., Lymphgefäße o.n.A. | |
| C50.0 | Mamille (Brust, Mamma) | |
| C50.1 | Zentraler Drüsenkörper (Brust, Mamma) | |
| C50.2 | Oberer innerer Quadrant (Brust, Mamma) | |
| C50.3 | Unterer innerer Quadrant (Brust, Mamma) | |
| C50.4 | Oberer äußerer Quadrant (Brust, Mamma) | |
| C50.5 | Unterer äußerer Quadrant (Brust, Mamma) | |
| C50.6 | Axillaere Ausläufer (Brust, Mamma) | |
| C50.7 | [Aberrierende Mamma (Brust, Mamma) Vorschlag org.spez.Tudok.] | |
| C50.8 | Brust (Mamma) (mehrere Teilregionen überlappend) | |
| C50.9 | Brust (Mamma) o.n.A. | |
| C51.0 | Labia Majora (einschl. Bartholini-Drüsen) | |
| C51.1 | Labia Minora | |
| C51.2 | Klitoris | |
| C51.8 | Vulva (mehrere Teilbereiche überlappend) | |
| C51.9 | Vulva o.n.A. | |
| C52.9 | Vagina o.n.A. | |
| C52.91 | Oberes Drittel der Vagina | |
| C52.92 | Vorderes Scheidengewölbe | |
| C52.93 | Hinteres Scheidengewölbe | |
| C52.94 | Mittleres Drittel der Vagina | |
| C52.95 | Äußeres Drittel der Vagina | |
| C52.96 | Hymenalsaum | |
| C53.0 | Endozervix | |
| C53.01 | Innerer Muttermund | |
| C53.02 | Zervixschleimhaut | |
| C53.1 | Ektozervix | |
| C53.11 | Äußerer Muttermund | |
| C53.12 | Portioepithel | |
| C53.8 | Zervix (mehrere Teilbereiche überlappend) | |
| C53.9 | Cervix uteri o.n.A. | |
| C54.0 | Isthmus uteri | |
| C54.1 | Endometrium (Stroma und Drüsen) | |
| C54.2 | Myometrium | |
| C54.3 | Fundus uteri | |
| C54.8 | Corpus uteri (mehrere Teilbereiche überlappend) | |
| C54.9 | Corpus uteri o.n.A. | |
| C55.9 | Uterus o.n.A. | |
| C56.9 | Ovar | |
| C57.0 | Tuba uterina | |
| C57.1 | Ligamentum latum, Mesovarium, Parovarialgewebe | |
| C57.2 | Ligamentum rotundum | |
| C57.3 | Parametrium | |
| C57.4 | Adnexe o.n.A. | |
| C57.7 | Wolffscher Gang | |
| C57.8 | Mehrere Teile d.weibl.Genitalorgane sowie Tumoren, deren Ausgangspkt.n.C51-C57.7 u. C58.9 zuordenbar | |
| C57.9 | Weibliche Genitalorgane o.n.A. | |
| C58.9 | Plazenta, foetale Membranen | |
| C60.0 | Praeputium, Vorhaut | |
| C60.1 | Glans Penis | |
| C60.2 | Penissschaft, Corpus cavernosum | |
| C60.8 | Penis (mehrere Teilbereiche überlappend) | |
| C60.9 | Penis o.n.A. | |
| C61.9 | Prostata o.n.A. | |
| C61.91 | Prostata, lateraler Lappen | |
| C61.92 | Prostata, Mittelappen | |
| C61.93 | Prostata, Isthmus | |
| C61.94 | Prostata, Apex | |
| C62.0 | Hodenhochstand, dystoper Hoden | |
| C62.1 | Hoden im Skrotum | |
| C62.9 | Hoden o.n.A. | |
| C63.0 | Nebenhoden, Epididymis | |
| C63.01 | Nebenhoden, Kopf | |
| C63.02 | Nebenhoden, Körper | |
| C63.03 | Nebenhoden, Schwanz | |
| C63.1 | Samenstrang (einschl. Ductus deferens) | |
| C63.2 | Skrotum o.n.A., Skrotalhaut | |
| C63.7 | Andere Teile der männl. Genitalorgane (z.B.Samenblasen, Tunica vaginalis testis) | |
| C63.8 | Männliche Genitalorgane (mehr. Teilber.) sowie Tumoren, deren Ausgangspkt.n. C60-C63.7 zuordenbar | |
| C63.9 | Männliche Genitalorgane o.n.A. | |
| C64.9 | Niere | |
| C64.91 | Oberes Drittel (einschl. oberer Pol) | |
| C64.92 | Mittleres Drittel | |
| C64.93 | Unteres Drittel (einschl. unterer Pol) | |
| C65.9 | Nierenbecken | |
| C65.91 | Nierenkelche | |
| C65.92 | Nierenbeckenauslaß | |
| C66.9 | Ureter | |
| C67.0 | Trigonum vesicae | |
| C67.1 | Fundus (Harnblase) | |
| C67.2 | Laterale Blasenwand | |
| C67.3 | Vordere Blasenwand | |
| C67.4 | Hintere Blasenwand | |
| C67.5 | Blasenhals | |
| C67.6 | Ureterostien | |
| C67.7 | Urachus | |
| C67.8 | Harnblase (mehrere Teilbereiche überlappend) | |
| C67.9 | Harnblase o.n.A. | |
| C68.0 | Urethra (inkl. Cowper-Drüse) | |
| C68.1 | Paraurethrale Drüsen | |
| C68.8 | Harnorgane (meh.Teilber.überlappend), sowie Tumoren, deren Ausgangsp. nicht in C64-C68.1 zuordenbar | |
| C68.9 | Harnorgane o.n.A. | |
| C69.0 | Konjunktiva | |
| C69.01 | Lidkonjunktiva | |
| C69.02 | Fornix der Konjunktiva | |
| C69.03 | Conjunctiva bulbi | |
| C69.1 | Cornea, Limbus corneae | |
| C69.2 | Retina (einschl.Sehnervenpapille) | |
| C69.3 | Chorioidea | |
| C69.4 | Bulbus oculi (Auge) | |
| C69.41 | Sklera (Auge) | |
| C69.42 | Iris (Auge) | |
| C69.43 | Ziliarkörper | |
| C69.44 | Uvealtrakt | |
| C69.45 | Linse | |
| C69.5 | Tränendrüse, Tränengang | |
| C69.51 | Papilla lacrimalis | |
| C69.52 | Tränen-Nasen-Gang | |
| C69.53 | Tränensack | |
| C69.6 | Orbita | |
| C69.61 | Capsula bulbi | |
| C69.62 | Fettgewebe (Corpus adiposum orbitae) | |
| C69.63 | Muskelgewebe (Auge) | |
| C69.64 | Gefäßgewebe (Auge) | |
| C69.65 | Lymphgewebe (Spatium circumbulbare) | |
| C69.66 | Retrobulbaeres Gewebe | |
| C69.8 | Auge (mehrere Teile überlappend) (ohne Augenlid, = C44.1) | |
| C69.9 | Auge o.n.A. | |
| C70.0 | Zerebrale Hirnhäute | |
| C70.01 | Kraniale Dura mater | |
| C70.02 | Kraniale Arachnoidea | |
| C70.03 | Kraniale Pia mater | |
| C70.04 | Falx cerebri | |
| C70.05 | Falx cerebelli | |
| C70.06 | Tentorium cerebelli | |
| C70.1 | Spinale Hirnhäute | |
| C70.11 | Spinale Dura mater | |
| C70.12 | Spinale Arachnoidea | |
| C70.13 | Spinale Pia mater | |
| C70.9 | Hirnhäute o.n.A. | |
| C71.0 | Großhirn | |
| C71.01 | Großhirnrinde | |
| C71.02 | Weisse Hirnsubstanz | |
| C71.03 | Basalganglien (Corpus Striatum, Putamen, Pallidum, Thalamus) | |
| C71.04 | Hypothalamus, Zwischenhirn | |
| C71.05 | Insula | |
| C71.06 | Operculum | |
| C71.07 | Septum pellucidum | |
| C71.08 | Capsula interna | |
| C71.1 | Frontallappen | |
| C71.2 | Temporallappen | |
| C71.21 | Hippocampus | |
| C71.22 | Uncus | |
| C71.3 | Parietallappen | |
| C71.4 | Okzipitallappen | |
| C71.5 | Hirnventrikel | |
| C71.51 | Seitenventrikel (mit Vorderhorn) | |
| C71.52 | Foramen Monroi | |
| C71.53 | III. Ventrikel | |
| C71.54 | Aquaeductus Sylvii | |
| C71.55 | Ependym | |
| C71.56 | Plexus chorioideus (Ventrikel I-III) | |
| C71.6 | Kleinhirn (Cerebellum) | |
| C71.61 | Oberwurm | |
| C71.62 | Unterwurm | |
| C71.63 | Hemisphaere | |
| C71.64 | Brückenwinkel | |
| C71.65 | Tonsilla cerebelli | |
| C71.7 | Hirnstamm | |
| C71.71 | Mittelhirn (und Lamina quadrigemina) | |
| C71.72 | Brücke (Pons) | |
| C71.73 | Medulla oblongata | |
| C71.74 | Olive, Pyramide | |
| C71.75 | Foramen Magendii | |
| C71.76 | Foramen Luschkae | |
| C71.77 | IV. Ventrikel (einschl.Plexus chorioideus) | |
| C71.78 | Hirnstamm (mehrere Teilbereiche) | |
| C71.8 | Andere Teile des Gehirns (z.B. Corpus callosum, Tapetum) | |
| C71.9 | Gehirn o.n.A. | |
| C72.0 | Rückenmark | |
| C72.01 | Zervikalmark | |
| C72.02 | Thorakalmark | |
| C72.03 | Lumbalmark | |
| C72.04 | Sakralmark | |
| C72.05 | Conus medullaris | |
| C72.06 | Filum terminale | |
| C72.1 | Cauda equina | |
| C72.2 | N. olfactorius | |
| C72.3 | N. opticus, Chiasma | |
| C72.4 | N. acusticus | |
| C72.5 | Andere Hirnnerven | |
| C72.51 | N. abducens | |
| C72.52 | N. facialis | |
| C72.53 | N. glossopharyngeus | |
| C72.54 | N. hypoglossus | |
| C72.55 | N. oculomotorius | |
| C72.56 | N. trigeminus | |
| C72.57 | N. trochlearis | |
| C72.58 | N. vagus | |
| C72.59 | N. accessorius | |
| C72.8 | Gehirn und zentrales Nervensystem (mehr.Teilber.)sowie Tumoren, d.nicht C70-C72.5 zuordenbar | |
| C72.9 | Nervensystem, zentrales o.n.A. | |
| C73.9 | Schilddrüse | |
| C73.91 | Seitenlappen | |
| C73.92 | Isthmus | |
| C73.93 | Lobus pyramidalis | |
| C73.94 | Ductus thyreoglossus | |
| C73.95 | Dystope Schilddrüse | |
| C74.0 | Nebennierenrinde | |
| C74.1 | Nebennierenmark | |
| C74.9 | Nebenniere o.n.A. | |
| C75.0 | Nebenschilddrüsen (Epithelkörperchen) | |
| C75.1 | Hypophyse | |
| C75.11 | Adenohypophyse | |
| C75.12 | Neurohypophyse | |
| C75.13 | Sella turcica | |
| C75.2 | Ductus craniopharyngealis | |
| C75.3 | Glandula pinealis, Zirbeldrüse | |
| C75.4 | Glomus caroticum | |
| C75.5 | Andere Paraganglien | |
| C75.51 | Glomus paraaorticum (Zuckerkandl-Organ) | |
| C75.52 | Glomus coccygicum | |
| C75.53 | Glomus jugulare | |
| C75.8 | Mehrere endokrine Drüsen | |
| C75.9 | Endokrine Drüsen o.n.A. | |
| C76.0 | Kopf, Gesicht, Hals o.n.A. | |
| C76.1 | Thorax o.n.A. | |
| C76.2 | Bauch o.n.A. | |
| C76.3 | Becken o.n.A. | |
| C76.4 | Obere Extremitaet o.n.A. | |
| C76.5 | Untere Extremitaet o.n.A. | |
| C76.7 | Andere mangelhaft bezeichnete Lokalisationen (z.B. Rücken, Flanke, Stamm) | |
| C76.8 | Mangelhaft bezeichnete Lokalisationen (überlappende Bereiche) | |
| C77.0 | Lymphknoten des Kopfes und Halses | |
| C77.01 | Ln. occipitales (retroauriculares), Ln. mastoidei | |
| C77.02 | Ln. parotidei, Ln. faciales | |
| C77.03 | Ln. submandibulares, Ln. submentales | |
| C77.04 | Ln. cervicales anteriores | |
| C77.05 | Ln. cervicales laterales superficiales | |
| C77.06 | Ln. cervicales laterales profundi | |
| C77.07 | Ln. supraclaviculares | |
| C77.08 | Ln. retropharyngeales | |
| C77.09 | Sonstige Lymphknoten im Kopf-Hals-Bereich | |
| C77.1 | Lymphknoten, thorakale und intrathorakale | |
| C77.11 | Ln. parasternales | |
| C77.12 | Ln. paratracheales | |
| C77.13 | Ln. pericardiales | |
| C77.14 | Ln. tracheobronchiales sup. et inf. (einschl. subkarinale) | |
| C77.15 | Lymphknoten des Mediastinums (einschl. infraklavikuläre) | |
| C77.16 | Ln. praevertebrales | |
| C77.17 | Ln. intercostales | |
| C77.18 | Ln. an A. mammaria interna | |
| C77.19 | Sonstige intrathorakale Lymphknoten | |
| C77.2 | Lymphknoten, intraabdominale | |
| C77.21 | Ln. coeliaci | |
| C77.22 | Lymphknoten, perigastrische | |
| C77.23 | Lymphknoten, peripankreatische | |
| C77.24 | Ln. splenici (lienales) | |
| C77.25 | Ln. hepatici | |
| C77.26 | Ln. mesenterici, Ln. mesocolici | |
| C77.27 | Ln. ileocolici | |
| C77.28 | Ln. paraaortales abdominales | |
| C77.29 | Ln. lumbales, Ln. retroperitoneales | |
| C77.3 | Lymphknoten der Achseln und oberen Gliedmassen | |
| C77.31 | Ln. cubitales | |
| C77.32 | Ln. brachiales | |
| C77.33 | Ln. pectorales | |
| C77.34 | Ln. subscapulares | |
| C77.35 | Ln. axillares | |
| C77.36 | Ln. sub- und infraclaviculares | |
| C77.4 | Lymphknoten der unteren Gliedmassen und inguinale Ln. | |
| C77.41 | Ln. inguinales (femorales) superficiales | |
| C77.42 | Ln. inguinales profundi | |
| C77.43 | Ln. popliteales | |
| C77.48 | Sonstige inguinale Lymphknoten (z.B. Cloquet, Rosenmüller) | |
| C77.5 | Lymphknoten des Beckens | |
| C77.51 | Ln. iliaci interni (hypogastrici) | |
| C77.52 | Ln. iliaci externi | |
| C77.53 | Ln. iliaci communes | |
| C77.54 | Ln. paravaginales, Ln. parauterini | |
| C77.55 | Ln. paravesiculares, Ln. pararectales | |
| C77.58 | Sonstige Lymphknoten des Beckens | |
| C77.8 | Lymphknoten mehrerer Regionen | |
| C77.9 | Lymphknoten in nicht naeher bezeichneter Region | |
| C80.9 | Unbekannte Primärlokalisation | |
| C00 | Lippen | |
| C01 | Zunge | |
| C02 | Zunge | |
| C03 | Mundschleimhaut | |
| C04 | Mundboden | |
| C05 | Gaumen | |
| C06 | Mund , Mundhöhle , kleine Speicheldrüsen | |
| C07 | Parotis | |
| C08 | Große Speicheldrüsen | |
| C09 | Tonsille | |
| C10 | Oropharynx | |
| C11 | Nasopharynx | |
| C12 | Sinus piriformis | |
| C13 | Hypopharynx | |
| C14 | Andere und ungenau bezeichnete Lokalisationen im Mundbereich | |
| C15 | Ösophagus | |
| C16 | Magen | |
| C17 | Dünndarm | |
| C18 | Dickdarm | |
| C19 | Rektosigmoid | |
| C20 | Rektum | |
| C21 | Anus und Analkanal | |
| C22 | Leber und intrahepatische Gallengänge | |
| C23 | Gallenblase | |
| C24 | Andere und nicht näher bezeichnete Teile der Gallenwege | |
| C25 | Pankreas | |
| C26 | Verdauungsorgane o.n.A. | |
| C30 | Nasenhöhle und Mittelohr | |
| C31 | Nebenhöhlen | |
| C32 | Larynx | |
| C33 | Trachea | |
| C34 | Bronchien und Lunge | |
| C37 | Thymus | |
| C38 | Herz, Mediastinum, Pleura | |
| C39 | Ungenaue Lokalisationen im Respirationssystem | |
| C40 | Knochen, Gelenke und Gelenkknorpel der Extremitäten | |
| C41 | Knochen, Gelenke und Gelenkknorpel anderer und nicht näher bezeichneter Lokalisationen | |
| C42 | Hämatopoetisches und retikuloendotheliales System | |
| C44 | Haut | |
| C47 | Peripher Nerven und autonomes Nervensystem | |
| C48 | Retroperitoneum und Peritoneum | |
| C49 | Bindegewebe, subkutane und andere Weichteilgewebe | |
| C50 | Brust | |
| C51 | Vulva | |
| C52 | Vagina | |
| C53 | Cervix uteri | |
| C54 | Corpus uteri | |
| C55 | Corpus uteri | |
| C56 | Ovar | |
| C57 | Andere und nicht näher bezeichnete Bereiche der weiblichen Genitalorgane | |
| C58 | Plazenta | |
| C60 | Penis | |
| C61 | Prostata | |
| C62 | Hoden | |
| C63 | Andere und nicht näher bezeichnete männliche Genitalorgane | |
| C64 | Niere | |
| C65 | Nierenbecken | |
| C66 | Ureter | |
| C67 | Harnblase | |
| C68 | Andere und nicht näher bezeichnete Organe des Harntraktes | |
| C69 | Auge und Adnexe | |
| C70 | Hirnhäute | |
| C71 | Gehirn | |
| C72 | Rückenmark, Hirnnerven und andere Teile des ZNS | |
| C73 | Schilddrüse | |
| C74 | Nebennieren | |
| C75 | Andere endokrine Drüsen | |
| C76 | Andere und mangelhaft bezeichnete Lokalisationen | |
| C77 | Lymphknoten | |
| C80 | Unbekannte Primärlokalisation |
Untersuchungsanlass
OID: 1.2.276.0.76.3.1.131.1.5.9
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| N | Nachsorge | |
| T | Therapie | |
| B | palliative Betreuung | |
| O | ärztliche Betreuung/onkologische Beratung | |
| S | Symptome | |
| D | Diagnostik | |
| Z | Zweitmeinung | |
| P | Planung |
Remissionsstatus
OID: 1.2.276.0.76.3.1.131.1.5.10
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| O | Postoperativ R0, Tumormarker nicht berücksichtigt oder nicht bekannt | |
| CR | Vollremission / Tumorfrei (complete remission, CR) | |
| PR | Teilremission / mindestens 50% Rückgang des Tumors (partial remission, PR) | |
| NC | Keine Änderung (no change, NC) | |
| PD | Progression | |
| U | Beurteilung unmöglich | |
| FT | Postoperativ R0 (free of tumor, FT), Tumormarker 4 Monate nach Operation bzw. anschließender Radio- und/oder Chemotherapie negativ (R0a nach [39 in BD5]) | |
| M | Postoperativ R0, anhaltend erhöhte Tumormarker oder Markeranstieg in den ersten 4 Monaten nach Operation bzw. Abschluss einer anschließender Radio- und/oder Chemotherapie (R0b nach [39 in BD5]) | |
| CRr | Vollremission mit residualen Auffälligkeiten (CRr) | |
| MR | Klinische Besserung des Zustandes, Kriterien für Teilremission jedoch nicht erfüllt (minimal response, MR) | |
| D | Divergentes Geschehen | |
| E | Entfällt, da Behandlung im Rahmen eines multimodalen Konzepts |
Status Primärtumor
OID: 1.2.276.0.76.3.1.131.1.5.11
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| T | Tumornachweis | |
| K | kein Tumornachweis | |
| R | Rezidiv | |
| F | fraglicher Befund | |
| E | Primärtumor vor Ersttherapie |
Status regionär
OID: 1.2.276.0.76.3.1.131.1.5.12
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| T | Tumornachweis | |
| K | kein Tumornachweis | |
| R | Rezidiv | |
| F | fraglicher Befund | |
| E | Lymphknoten vor Ersttherapie | |
| B | verbliebene und neue Metastasen |
Status Fernmetastasen
OID: 1.2.276.0.76.3.1.131.1.5.13
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| T | Tumornachweis | |
| K | kein Tumornachweis | |
| R | Rezidiv | |
| F | fraglicher Befund | |
| E | Fernmetastasen vor Ersttherapie | |
| B | verbliebene und neue Metastasen |
Therapiekategorien
OID: 1.2.276.0.76.3.1.131.1.5.14
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| RAD | Strahlentherapie o.n.A. | |
| RAD-B | Brachytherapie | |
| RAD-T | Teletherapie | |
| RAD-S | sonstige Strahlentherapie | |
| NUK-J | Radiojodtherapie | |
| NUK-O | offene Radionuklide | |
| NUK-S | sonstige Nuklearmedizinische Therapie | |
| CHE | Chemotherapie | |
| HOR | Hormontherapie / Antihormonelle Therapie | |
| KNTR | Knochenmarktransplantation | |
| STAMM | Stammzelltransplantation o.n.A. | |
| STAMM-L | Autologe Stammzelltransplantation | |
| STAMM-G | Sonstige Stammzelltransplantation | |
| STAMM-S | Allogene Stammzelltransplantation | |
| IMM | Antikörper / Immuntherapie | |
| SCHM | Schmerztherapie | |
| PSY | Psychoonkologie | |
| SM | Sonstige Medikamentöse Therapie | |
| WS | Wait and see / Active surveillance | |
| ATH | Sonstige / andere Therapie o.n.A. | |
| ATH-HY | Hyperthermie |
Komplikationen
OID: 1.2.276.0.76.3.1.131.1.5.15
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| ABD | Abszess in einem Drainagekanal | |
| ABS | Abszess, intraabdominaler oder intrathorakaler (z.B. LeberAbszess, subphrenischer Abszess) | |
| AEE | Anastomoseninsuffizienz einer Enterostomie | |
| AEP | Alkoholentzugspsychose | |
| ALR | Allergische Reaktion ohne Schocksymptomatik | |
| ANI | Akute Niereninsuffizienz | |
| ANS | Anaphylaktischer Schock | |
| API | Apoplektischer Insult | |
| ASF | Abszess, subfaszialer | |
| BIF | Biliäre Fistel | |
| BOE | Bolusverlegung eines Endotubus | |
| BOG | Blutung, obere gastrointestinale (z.B "Stressulkus") | |
| BSI | Bronchusstumpfinsuffizienz | |
| CHI | Cholangitis | |
| DAI | Darmanastomoseninsuffizienz | |
| DEP | Drogenentzugspsychose | |
| DIC | Disseminierte intravasale Koagulopathie | |
| DLU | Druck- und Lagerungsschäden, z.B. Dekubitalulzera | |
| DPS | Darmpassagestörungen (z.B. protrahierte Atonie, Subileus, Ileus) | |
| DSI | Duodenalstumpfinsuffizienz | |
| ENF | Enterale Fistel | |
| GER | Gerinnungsstörung | |
| HAE | Hämorrhagischer Schock | |
| HEM | Hämatemesis | |
| HFI | Harnfistel | |
| HNA | Hirnnervenausfälle | |
| HNK | Hautnekrose im Operationsbereich | |
| HOP | Hirnorganisches Psychosyndrom (z.B. "Durchgangssyndrom") | |
| HRS | Herzrhythmusstörungen | |
| HUR | Hämaturie | |
| HYB | Hyperbilirubinämie | |
| HYF | Hypopharynxfistel | |
| HZI | Herzinsuffizienz | |
| IFV | Ileofemorale Venenthrombose | |
| KAS | Kardiogener Schock | |
| KDS | Kurzdarmsyndrom | |
| KES | Komplikationen einer Stomaanlage | |
| KIM | Komplikation eines Implantates (Gefäßprothese, Totalendoprothese, Katheter), z.B. Dislokation | |
| KRA | Krampfanfall | |
| LEV | Leberversagen | |
| LOE | Lungenödem | |
| LYE | Lymphozele | |
| LYF | Lymphfistel | |
| MAT | Mesenterialarterien- oder -venenthrombose | |
| MED | Mediastinitis | |
| MES | Magenentleerungsstörung | |
| MIL | Mechanischer Ileus | |
| MYI | Myokardinfarkt | |
| NAB | Nachblutung, nicht revisionsbedürftig, anderweitig nicht erwähnt | |
| NIN | Nahtinsuffizienz, anderweitig nicht erwähnt | |
| OES | Ösophagitis | |
| OSM | Osteitis, Osteomyelitis | |
| PAB | Peranale Blutung | |
| PAE | Pulmonalarterienembolie | |
| PAF | Pankreasfistel | |
| PAV | Peripherer arterieller Verschluß (Embolie, Thrombose) | |
| PDA | Protrahierte Darmatonie (paralytischer Ileus) | |
| PER | Peritonitis | |
| PEY | Pleuraempyem | |
| PIT | Pankreatitis | |
| PLB | Platzbauch | |
| PLE | Pleuraerguss | |
| PMN | Pneumonie | |
| PNT | Pneumothorax | |
| PPA | Periphere Parese | |
| RIN | Respiratorische Insuffizienz | |
| RNB | Nachblutung, revisionsbedürftig, anderweitig nicht erwähnt | |
| RPA | Rekurrensparese | |
| SES | Septischer Schock | |
| SFH | Störungen des Flüssigkeits-, Elektrolyt- und Säurebasenhaushaltes | |
| SKI | Septische Komplikation eines Implantates | |
| SON | sonstige Komplikationen | |
| STK | Stomakomplikation (z.B. Blutung, Nekrose, Stenose) | |
| TIA | TIA (transitorische ischämische Attacke) oder RIND (reversibles ischämisches neurologisches Defizit) | |
| TRZ | Transfusionszwischenfall | |
| TZP | Thrombozytopenie | |
| WSS | Wundheilungsstörung, subkutane | |
| WSSnr | Wundheilungsstörung, subkutane, nicht revisionsbedürftig | |
| WSSr | Wundheilungsstörung, subkutane, revisionsbedürftig | |
| WUH | Wundhämatom (konservativ therapiert) |
Zielgebiet der Strahlentherapie (Zielgebietsschlüssel, BDT 5. Aufl. - Version B4)
OID: 1.2.276.0.76.3.1.131.1.5.16
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| 1. | ZNS | |
| 1.1 | Hirnschädel | |
| 1.2 | Kleinhirn | |
| 1.3 | Hypophyse | |
| 1.4 | Siebbein | |
| 1.5 | Orbita | |
| 1.6 | Retrobulbärregion | |
| 1.7 | Myelon | |
| 2. | Kopf / Hals | |
| 2.1 | Wange | |
| 2.2 | Tonsille | |
| 2.3 | Schilddrüse | |
| 2.4 | Nasopharynx | |
| 2.5 | Oropharynx | |
| 2.6 | Hypopharynx | |
| 2.7 | Oberkiefer | |
| 2.8 | Unterkiefer | |
| 2.9 | Mundboden | |
| 2.10 | Zunge | |
| 2.11 | Lippe | |
| 2.12 | Larynx | |
| 2.13 | Hals | |
| 2.14 | Ohr | |
| 2.15 | Nase | |
| 2.16 | Nasennebenhöhlen | |
| 2.17 | Gesichtsschädel | |
| 3. | Thorax | |
| 3.1 | Mediastinum | |
| 3.2 | Hilus+Mediastinum | |
| 3.3 | Hilus | |
| 3.4 | Ösophagus | |
| 3.5 | Lunge o.n.A. | |
| 3.6 | Lunge zentral | |
| 3.7 | Lunge peripher | |
| 4. | Abdomen | |
| 4.1 | Abdomen | |
| 4.2 | Leber | |
| 4.3 | Leberhilus | |
| 4.4 | Milz | |
| 4.5 | Milzstiel | |
| 4.6 | Magen | |
| 4.7 | Pankreas | |
| 5. | Kleines Becken | |
| 5.1 | kleines Becken | |
| 5.2 | Beckenwand | |
| 5.3 | Beckenschaufel | |
| 5.4 | Rektum | |
| 5.5 | Sakralhöhle | |
| 5.6 | Analfeld | |
| 6. | Urogenitalsystem | |
| 6.1 | Niere | |
| 6.2 | Nebenniere | |
| 6.3 | Blase | |
| 6.4 | Prostata | |
| 6.5 | Hoden | |
| 7. | Wirbelsäule | |
| 7.1 | Halswirbelsäule | |
| 7.2 | Brustwirbelsäule | |
| 7.3 | Lendenwirbelsäule | |
| 7.4 | Paravertebralgebiet | |
| 7.5 | Sonstige Knochen | |
| 8. | Brust | |
| 8.1 | Brustwand | |
| 8.2 | Sternum | |
| 8.3 | Mamma | |
| 8.4 | Axilla | |
| 9. | Extremitäten | |
| 9.1 | Hüfte | |
| 9.2 | Oberschenkel | |
| 9.3 | Unterschenkel | |
| 9.4 | Fuß | |
| 9.5 | Schulter/Hals-Region | |
| 9.6 | Oberarm | |
| 9.7 | Unterarm | |
| 9.8 | Hand | |
| 10. | Weibl. Genitale | |
| 10.1 | Cervix uteri | |
| 10.2 | Corpus uteri | |
| 10.3 | Parametrien | |
| 10.4 | Ovar | |
| 10.5 | Vagina | |
| 10.6 | Vulva | |
| 11. | Haut | |
| 12. | Spezielle Felder | |
| 12.1 | Ganzkörper | |
| 12.2 | Halbkörper supradiaphragmal | |
| 12.3 | Halbkörper infradiaphragmal | |
| 13. | Lymphknoten | |
| 13.1 | Inguinale LK | |
| 13.2 | Iliakale LK | |
| 13.3 | Paraaortale LK | |
| 13.4 | Subphrenische LK | |
| 13.5 | Submentale LK | |
| 13.6 | Mantelfeld | |
| 13.7 | Y-Feld | |
| 13.8 | Zervikale LK | |
| 13.9 | Supraklavikul. LK | |
| 13.a | Axilläre LK | |
| 13.b | Sternale und retrosternale LK | |
| 13.c | Beckenlymphabfl. | |
| 14. | Sonstige Gebiete | |
| X. | Fehlende Angabe |
Applikationsart der Strahlentherapie
OID: 1.2.276.0.76.3.1.131.1.5.17
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| P | Teletherapie (perkutane Therapie) | |
| K | Endokavitäre Kontakttherapie | |
| I | Interstitielle Kontakttherapie | |
| M | Therapie mit offenen Radionukliden (metabolische Therapie) | |
| A | Andere Kontakttherapie |
Therapieintention
OID: 1.2.276.0.76.3.1.131.1.5.18
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| K | kurativ | |
| P | palliativ | |
| D | diagnostisch |
Beendigungsstatus der Therapie
OID: 1.2.276.0.76.3.1.131.1.5.19
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| A | Abbruch wegen Nebenwirkungen | |
| E | reguläres Ende | |
| S | sonstiger Abbruchgrund | |
| V | Patient verweigert weitere Therapie |
WHO-ART 1999 (Basisdokumentation für Tumorkranke, 5. Auflage)
OID: 1.2.276.0.76.3.1.131.1.5.21
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| HGL | Hämoglobin | |
| LEU | Leukozyten (1000/mm3) | |
| GRA | Granulozyten(1000/mm3) | |
| THR | Plättchen (1000/mm3) | |
| BLU | Hämorrhagie | |
| BIL | Bilirubin | |
| TRA | Transaminasen (SGOT/SGPT) | |
| ALK | Alkalische Phosphatase | |
| ORA | Oral | |
| UBL | Übelkeit/Erbrechen | |
| DIA | Diarrhö | |
| KRE | Kreatinin | |
| PRO | Proteinurie | |
| HUR | Hämaturie | |
| LUN | Lunge | |
| FIE | Fieber nach Medikamenten | |
| ALL | Allergie | |
| HAU | Haut | |
| HAA | Haare | |
| INF | Infektion (Herd angeben) | |
| RHY | Herzrhythmus | |
| FUN | Herzfunktion | |
| PER | Perikarditis | |
| BEW | Bewußtseinszustand | |
| NER | Periphere Nerven | |
| OBS | Obstipation | |
| DOL | Schmerz | |
| HAR | Blutharnstoff | |
| XXX | Weitere Befunde von klinischer Relevanz |
RTOG Toxicity Criteria
OID: 1.2.276.0.76.3.1.131.1.5.22
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| 1 | AJCC-/ECOG- Skala: Karnofsky-Index (KI in %) | |
| 2a | Leukozyten (N/µl) | |
| 2b | Neutrophile (N/µl) | |
| 2c | Thrombozyten (x 10³/µl) | |
| 2d | Hämoglobin (g/dl) | |
| 2e | Hämatokrit (Vol.-%) | |
| 2f | Blutung (klinisch) | |
| 2g | Infektion | |
| 3a | Haut | |
| 3b | Unterhautgewebe | |
| 4 | Schleimhäute | |
| 5 | Speicheldrüsen | |
| 6 | (Pharynx und) Speiseröhre | |
| 7 | Kehlkopf | |
| 8 | Lunge | |
| 9 | Herz | |
| 10 | Oberer GI-Trakt | |
| 11 | Unterer GI-Trakt (Dünn- und Dickdarm) | |
| 12 | Leber | |
| 13a | Niere | |
| 13b | Hämaturie | |
| 14 | Harnleiter und Blase | |
| 15 | Knochen | |
| 16 | Gelenke | |
| 17 | Gehirn (ZNS) | |
| 18 | Rückenmark (Periphere Nerven) | |
| 19 | Augen | |
| 20 | Ohren |
WHO-ART 1999 (Basisdokumentation für Tumorkranke, 5. Auflage) - Grad
OID: 1.2.276.0.76.3.1.131.1.5.24
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| 0 | WHO-Grad 0 | |
| 1 | WHO-Grad 1 | |
| 2 | WHO-Grad 2 | |
| 3 | WHO-Grad 3 | |
| 4 | WHO-Grad 4 |
RTOG Toxicity Criteria - Grad
OID: 1.2.276.0.76.3.1.131.1.5.25
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| 0 | RTOG-Grad 0 | |
| 1 | RTOG-Grad 1 | |
| 2 | RTOG-Grad 2 | |
| 3 | RTOG-Grad 3 | |
| 4 | RTOG-Grad 4 | |
| 5 | RTOG-Grad 5 |
Risikokategorien bei trophoblastären Schwangerschaftstumoren (C58)
OID: 1.2.276.0.76.3.1.131.1.5.53
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| N | niedrig | |
| H | hoch |
Boolsche Werte für Datentyp CD
OID: 1.2.276.0.76.3.1.131.1.5.61
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| true | true, ja | |
| false | false, nein |
zeitliche Relation der Therapie zur Operation
OID: 1.2.276.0.76.3.1.131.1.5.62
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| N | neoadjuvant | |
| I | intraoperativ | |
| A | adjuvant | |
| T | alleinige Therapie (ohne vorausgehende oder folgende operative Therapie) |
Gültigkeitsbereich der Patientenzustimmung
OID: 1.2.276.0.76.3.1.131.1.5.63
Diese Tabelle wird ggf. weiter ergänzt bzw. bundeslandspezifisch verfeinert.
| Code | Bedeutung (Kurztext) | Bemerkung |
|---|---|---|
| EKR | epidemiologisches Krebsregister | |
| KKR | klinisches Krebsregister | |
| AUD | Auditor für Organzentren/onkologische Zentren | derzeit nicht verwendet |
| BEN | Benchmarking-Stelle | derzeit nicht verwendet |
| QS | Qualitätssicherung §137a SGB V | derzeit nicht verwendet |
Value Sets
Dokument-Arten
OID: 1.2.276.0.76.3.1.131.1.11.1
| Code | Codesystem | Codesystem Name | Bedeutung |
|---|---|---|---|
| 34806-0 | 2.16.840.1.113883.6.1 | LOINC | Oncology Note |
| x-physician-cancer-rep | 2.16.840.1.113883.6.1 | LOINC | Report to Cancer Registries |
Vertraulichkeit bei onkologischen Dokumenten
OID: 1.2.276.0.76.3.1.131.1.11.2
| Code | Codesystem | Codesystem Name | Bedeutung |
|---|---|---|---|
| N | 2.16.840.1.113883.5.25 | HL7 Confidentiality Code | normal |
bundeslandspezifische Meldebegründung
Die Meldebegründung ist bundeslandspezifisch unterschiedlich. Die Codierung erfolgt im Codesystem 1.2.276.0.76.3.1.131.1.5.2. Die für die einzelnen Bundesländer gültigen Werte sind nachfolgend definiert. Wenn zwei Bundesländer dieselbe Wertemenge vorgeben, so wird dafür dieselbe Value Set OID verwendet. Bei Bundesländern, welche diese Meldebegründungen nicht entgegennehmen, wurde ein "-" vergeben.
| Bundesland, Code | E | A | I | V | D | W | ValueSet OID |
|---|---|---|---|---|---|---|---|
| Baden-Württemberg | X | X | X | X | X | X | 1.2.276.0.76.3.1.131.1.11.3.6 |
| Bayern, Saarland |
- | X | X | - | X | - | 1.2.276.0.76.3.1.131.1.11.3.1 |
| Berlin, Brandenburg, Mecklenburg-Vorpommern, Sachsen-Anhalt, Sachsen, Thüringen |
- | - | - | - | - | - | - |
| Bremen | - | X | X | X | X | - | 1.2.276.0.76.3.1.131.1.11.3.2 |
| Hamburg, Niedersachsen |
X | X | - | X | - | - | 1.2.276.0.76.3.1.131.1.11.3.3 |
| Hessen | - | - | - | - | - | - | - |
| Nordrhein-Westfalen | - | - | X | - | - | X | 1.2.276.0.76.3.1.131.1.11.3.4 |
| Rheinland-Pfalz | - | - | - | - | - | - | - |
| Saarland | - | X | X | - | X | - | 1.2.276.0.76.3.1.131.1.11.3.5 |
Phänomen-Arten
OID: 1.2.276.0.76.3.1.131.1.11.4
| Code | Codesystem | Codesystem Name | Bedeutung |
|---|---|---|---|
| 21855-2 | 2.16.840.1.113883.6.1 | LOINC | Primary site Cancer |
| 21920-4 | 2.16.840.1.113883.6.1 | LOINC | Site of distant metastasis Cancer |
Organe bei extranodalem Befall, Ann Arbor Stadium
OID: 1.2.276.0.76.3.1.131.1.11.5
| Code | Codesystem | Codesystem Name | Bedeutung |
|---|---|---|---|
| 127230005 | 2.16.840.1.113883.6.96 | SNOMED CT | Milz |
| 206892013 | 2.16.840.1.113883.6.96 | SNOMED CT | Knochen |
| 417127010 | 2.16.840.1.113883.6.96 | SNOMED CT | Knochenmark |
| 280107018 | 2.16.840.1.113883.6.96 | SNOMED CT | Lunge |
| 280201012 | 2.16.840.1.113883.6.96 | SNOMED CT | Leber |
| 384461017 | 2.16.840.1.113883.6.96 | SNOMED CT | Gehirn |
| 280743016 | 2.16.840.1.113883.6.96 | SNOMED CT | Pleura |
| 26155015 | 2.16.840.1.113883.6.96 | SNOMED CT | Peritoneum |
| 279965012 | 2.16.840.1.113883.6.96 | SNOMED CT | Nebenniere |
| 280523012 | 2.16.840.1.113883.6.96 | SNOMED CT | Haut |
| 258348004 | 2.16.840.1.113883.6.96 | SNOMED CT | anderes Organ |
OIDs für Templates
Diese Seite listet alle Kodesysteme auf, für die es eine gewisse Relevanz gibt wohl wissend, dass es für diesen Zweck auf sog. OID-Registries gibt:
|
|
Für die Verwendung in FHIR getroffene Festlegungen zu den Kodesystemen sind in den Deutschen Basisprofilen zu finden. |
|
|
Die Verweise (Hyperlinks) sind noch einzubauen.Hier darf man sich noch austoben...Für alle Templates des Pathologiebefundes sollten die OID von IHE PAT verwendet werden, wo solche fehlen, ist die Registrierung durch IHE PAT anzustreben. GH |
| Vokabeldomäne/ Codiersystem | OID | Kurzbezeichnung (codesystem in HL7 v2.x) |
Typ des Kodesystems | Sonstiges |
|---|---|---|---|---|
| ICD-10-GM | ||||
| ICD-10 GM Version 2017 | 1.2.276.0.76.5.463 | icd10gm2017 | Diagnosen | |
| ICD-10 GM Version 2016 | 1.2.276.0.76.5.430 | icd10gm2016 | Diagnosen | |
| ICD-10 GM Version 2015 | 1.2.276.0.76.5.424 | icd10gm2015 | Diagnosen | |
| ICD-10 GM Version 2014 | 1.2.276.0.76.5.417 | icd10gm2014 | Diagnosen | |
| ICD-10 GM Version 2013 | 1.2.276.0.76.5.413 | icd10gm2013 | Diagnosen | |
| ICD-10 GM Version 2012 | 1.2.276.0.76.5.409 | icd10gm2012 | Diagnosen | |
| ICD-10 GM Version 2011 | 1.2.276.0.76.5.388 | icd10gm2011 | Diagnosen | |
| ICD-10 GM Version 2010 | 1.2.276.0.76.5.384 | icd10gm2010 | Diagnosen | |
| ICD-10 GM Version 2009 | 1.2.276.0.76.5.356 | icd10gm2009 | Diagnosen | |
| ICD-10 GM Version 2008 | 1.2.276.0.76.5.330 | icd10gm2008 | Diagnosen | |
| ICD-10 GM Version 2007 | 1.2.276.0.76.5.318 | icd10gm2007 | Diagnosen | |
| ICD-10 GM Version 2006 | 1.2.276.0.76.5.311 | icd10gm2006 | Diagnosen | |
| ICD-10 GM Version 2005 | 1.2.276.0.76.5.304 | icd10gm2005, I10-2005 | Diagnosen | |
| ICD-10 GM Version 2004 | 1.2.276.0.76.5.302 | icd10gm2004, I10-2004 | Diagnosen | |
| ICD-10 Version 2.0 | n.def. | I10-20 | Diagnosen | |
| ICD-10 Version 1.3 | n.def. | I10-13 | Diagnosen | |
| Alpha-ID | ||||
| Alpha-ID 2017 | 1.2.276.0.76.5.465 | alphaid2017 | Diagnosen | |
| Alpha-ID 2016 | 1.2.276.0.76.5.432 | alphaid2016 | Diagnosen | |
| Alpha-ID 2015 | 1.2.276.0.76.5.426 | alphaid2015 | Diagnosen | |
| Alpha-ID 2014 | 1.2.276.0.76.5.419 | alphaid2014 | Diagnosen | |
| Alpha-ID 2013 | 1.2.276.0.76.5.415 | alphaid2013 | Diagnosen | |
| Alpha-ID 2012 | 1.2.276.0.76.5.408 | alphaid2012 | Diagnosen | |
| Alpha-ID 2011 | 1.2.276.0.76.5.387 | alphaid2011 | Diagnosen | |
| Alpha-ID 2010 | 1.2.276.0.76.5.383 | alphaid2010 | Diagnosen | |
| Alpha-ID 2009 | 1.2.276.0.76.5.355 | alphaid2009 | Diagnosen | |
| Alpha-ID 2008 | 1.2.276.0.76.5.329 | alphaid2008 | Diagnosen | |
| Alpha-ID 2007 | 1.2.276.0.76.5.316 | alphaid2007 | ||
| Alpha-ID 2006 | 1.2.276.0.76.5.309 | alphaid2006 | ||
| ICD-O | ||||
| ICD-O-3 | 2.16.840.1.113883.6.43.1 | icd-o-3 | ||
| ICD-O-DA-1978 | n.a. | |||
| ICD-O-DA-2002 | n.a. | |||
| TNM: Tumordokumentation | ||||
| TNM 5. Auflage (UICC) | 2.16.840.1.113883.15.8 | tnm5 | ||
| TNM 6. Auflage (UICC) | 2.16.840.1.113883.15.7 | tnm6 | ||
| TNM 7. Auflage (UICC) | 2.16.840.1.113883.15.6 | tnm7 | ||
| TNM 8. Auflage (UICC) | tnm8 | |||
| TNM: C-Faktor | 1.2.276.0.76.5.341 | c-faktor-tumor | ||
| TNM-Qualifier | 1.2.276.0.76.5.340 | tnm-qualifier | ||
| TNM: Metastasen | 1.2.276.0.76.5.339 | metastasen-tnm | ||
| TNM: Nodus | 1.2.276.0.76.5.338 | nodus-tnm | ||
| TNM: Ausdehnung | 1.2.276.0.76.5.337 | ausdehnung-tnm | ||
| TNM: Differenzierung/Grading (KLÄREN!) | 1.2.276.0.76.5.336 | diff-grading-tumor | ||
| TNM: Dignität | 1.2.276.0.76.5.335 | dignitaet-tumor | ||
| TNM: Tumordiagnosen | 1.2.276.0.76.5.334 | tumordiagnosen | ||
| Zell-Typ (KLÄREN!) | OID bereits durch ICD belegt!!!! | |||
| Gültigkeitsbereich R-Klassifikation | ||||
| Vorhandensein Residualtumor | ||||
| Metastasen-Lokalisation | 1.2.276.0.76.5.401 | OID nicht registriert! | ||
| Typisierung-diagnose | 1.2.276.0.76.5.342 | typisierung-diagnose | ||
| OPS | ||||
| OPS 2017 | 1.2.276.0.76.5.464 | ops2017 | Maßnahmen | |
| OPS 2016 | 1.2.276.0.76.5.431 | ops2016 | Maßnahmen | |
| OPS 2015 | 1.2.276.0.76.5.425 | ops2015 | Maßnahmen | |
| OPS 2014 | 1.2.276.0.76.5.418 | ops2014 | Maßnahmen | |
| OPS 2013 | 1.2.276.0.76.5.414 | ops2013 | Maßnahmen | |
| OPS 2012 | 1.2.276.0.76.5.410 | ops2012 | Maßnahmen | |
| OPS 2011 | 1.2.276.0.76.5.389 | ops2011 | Maßnahmen | |
| OPS 2010 | 1.2.276.0.76.5.385 | ops2010 | Maßnahmen | |
| OPS 2009 | 1.2.276.0.76.5.357 | ops2009 | Maßnahmen | |
| OPS 2008 | 1.2.276.0.76.5.331 | ops2008 | Maßnahmen | |
| OPS 2007 | 1.2.276.0.76.5.317 | ops2007, O301-2007 | Maßnahmen | 2. Bez. wird bei HL7v2 genutzt |
| OPS 2006 | 1.2.276.0.76.5.310 | ops2006, O301-2006 | Maßnahmen | 2. Bez. wird bei HL7v2 genutzt |
| OPS 2005 | 1.2.276.0.76.5.303 | ops2005, O301-2005 | Maßnahmen | 2. Bez. wird bei HL7v2 genutzt |
| OPS-301 2004 | 1.2.276.0.76.5.301 | ops301v2004, O301-2004 | Maßnahmen | 2. Bez. wird bei HL7v2 genutzt |
| OPS-301 Version 2.1 | n.def. | OPS301-21 | Maßnahmen | |
| OPS-301 Version 2.0 | n.def. | OPS301-01 | Maßnahmen | |
| OPS-301 Version 1.0 | n.def. | OPS301-10 | Maßnahmen | |
| ATC | ||||
| ATC GM 2017 | 1.2.276.0.76.5.468 | atcgm2017 | ||
| ATC GM 2016 | 1.2.276.0.76.5.435 | atcgm2016 | ||
| ATC GM 2015 | 1.2.276.0.76.5.427 | atcgm2015 | ||
| Pflege- Interventionen | ||||
| International Classification of Nursing Practice (ICNP) | 2.16.840.1.113883.6.97 | Diagnose, Lokalisation | ||
| NANDA-I Nursing Diagnosis | 2.16.840.1.113883.6.204 | Diagnose | ||
| International Classification of Functioning, Disability and Health (ICF) | 2.16.840.1.113883.6.254 | Diagnose, Lokalisation | ||
| Clinical Care Classification (CCC) | 2.16.840.1.113883.6.236 | |||
| apenio | 1.2.276.0.76.3.1.125.1 | Diagnose | ||
| European Nursing care pathways | 1.2.276.0.76.5.407 | Diagnose | ||
| lep-nursing-3 | 1.2.276.0.76.5.391 | |||
| Scores | ||||
| Gleason-Score | ||||
| Gleason-Score: Entdifferenzierungsgrad | 1.2.276.0.76.5.402 | |||
| Gleason-Score: Wachstumsmuster | 1.2.276.0.76.5.403 | |||
| Gleason-Score: Grading | 1.2.276.0.76.5.404 | |||
| Ann-Arbor | 1.2.276.0.76.5.405 | |||
| Ann-Arbor: Allgemeinsymptome | 1.2.276.0.76.5.xxx | |||
| Ann-Arbor: Organbefall | 1.2.276.0.76.5.xxx | |||
| Papanikolaou: Grading | 1.2.276.0.76.5.406 | |||
| sonstige | ||||
| MeSH | ||||
| MeSH (deutsch) | 2.16.840.1.113883.6.177.5 | MSHGER | Diagnose, Lokalisation | |
| Kodiersysteme | ||||
| LOINC | 2.16.840.1.113883.6.1 | loinc | Diagnose, Prozeduren, Befunde | |
| Snomed CT | 2.16.840.1.113883.6.96 | SNOMED CT | Diagnose, Maßnahmen, Befunde, ... | [2] |
| ID Macs | 1.2.276.0.76.5.305 | id_macs | Diagnose, Maßnahmen, Befunde, ... | [3] |
| .. | ||||
| 1.2.276.0.76.5.342 | Typisierung-diagnose | Diagnose, Lokalisation |
Tabelle 5: Kodierschemata
ICD-Kodes
Diese Seite enthält auch die Beschlüsse des TC Konformität und Zertifizierung vom Dezember 2003, März 2005 und November 2007.
Ab dem 01. Januar 2004 wechselt das DIMDI die Bezeichnung für die unterschiedlichen Versionen bei den ICD-10 und OPS-301, hierzu werden die Kennzeichnungen gemäß nachfolgender Tabelle in HL7 verwendet. Die Systematik des DIMDI zur Bezeichnung der ICD-10 Kataloge lautet von 2004 bis 2007: I10-<Jahr>.
Am 29.11.2007 hat das TC beschlossen, ab 2008 auf die neuen Bezeichner zu wechseln. Damit ist die Namenskonvention wieder synchron mit den Vorgaben von HL7 USA:
OPS-Kodes
Die Systematik des DIMDI zur Bezeichnung der OPS301 Kataloge lautet von 2004 bis 2007: O301-<Jahr>.
Besonderheit ab Version 2.6
Mit der Einführung von Version 2.6 hat HL7 International die Benutzung von Sonderzeichen wie bspw. das Minuszeichen in der HL7 Tabelle 0396 - Coding System ausgeschlossen. Zu dieser Vorgabe, die eine Rückwärtskompatibilität der deutschen Entscheidung für die Identifikation der Kodiersysteme OPS301 und ICD-10 beeinträchtigt, wurde eine erneute Entscheidung durch das TC Konformität und Zertifizierung getroffen:
In Abweichung von der internationalen Spezifikation ist es in Deutschland nach wie vor erlaubt, dass Sonderzeichen in dem Bezeichner des Kodiersystems vorkommen dürfen. (Beschluss vom 17.März 2005)
Dies bedeutet das die bisherige Systematik der Bezeichnung für die Kodiersysteme auch in den Versioonen 2.6 und folgende beibehalten wird.
Auf Anfrage hat das TC am 29.11.2007 beschlossen, ab 2008 doch auf das neue Schema zu wechseln und damit den Konflikt zu lösen.
Für die Zukunft ...
Wie aus dem Beschluss des TCs hervorgeht wird diese Systematik bis auf weiteres fortgeführt.