TNM-Klassifikation-Entry (Template)
Slang (Diskussion | Beiträge) (→Fernmetastasen) |
(→Darstellung der TNM-Klassifikation in HL7) |
||
| (41 dazwischenliegende Versionen von 4 Benutzern werden nicht angezeigt) | |||
| Zeile 4: | Zeile 4: | ||
{| class="hl7table" | {| class="hl7table" | ||
| − | |bgcolor="ddddff"| | + | |bgcolor="ddddff"| || bgcolor="ddddff"| Template-Metadaten |
|- | |- | ||
| − | |bgcolor="ddddff"| | + | |bgcolor="ddddff"|Template-Typ|| Entry |
|- | |- | ||
| − | |bgcolor="ddddff"| | + | |bgcolor="ddddff"|Template ID|| 1.2.276.0.76.3.1.131.1.10.3.5 ????? |
|- | |- | ||
| − | | | + | |bgcolor="ddddff"|generischeres Template || <Verweis auf das Template> |
| + | |- | ||
| + | |bgcolor="ddddff"|genutztes Templates || R-Klassifikation | ||
| + | |- | ||
| + | |bgcolor="ddddff"|abgeleitete Templates || [[cdapath:TNM-Klassifikation-Entry (Template)]] | ||
| + | [[cdaonk:TNM-Klassifikation-Entry (Template)]] | ||
| + | |- | ||
| + | |bgcolor="ddddff"|Generelle Beschreibung|| Hiermit wird auf Entry-Level die Übermittlung von Tumordiagnosedaten (UICC TNM-Klassifikation) definiert. | ||
| + | |- | ||
| + | |bgcolor="ddddff"|allg. Erläuterung|| | ||
| + | |- | ||
| + | |bgcolor="ddddff"|Verhältnis zu IHE|| neu: Abstimmung mit QRPH und AP | ||
| + | |- | ||
| + | |bgcolor="ddddff"|Ballotierungsstatus|| in Arbeit | ||
| + | |- | ||
| + | |bgcolor="ddddff"|Erweiterbarkeit|| geschlossen | ||
|} | |} | ||
| Zeile 24: | Zeile 39: | ||
{| class="hl7table" | {| class="hl7table" | ||
!TNM-Symbol!!Bedeutung | !TNM-Symbol!!Bedeutung | ||
| − | |||
| − | |||
|- | |- | ||
|y||nach neoadjuvanter Therapie | |y||nach neoadjuvanter Therapie | ||
|- | |- | ||
|r||Beurteilung eines Rezidivs | |r||Beurteilung eines Rezidivs | ||
| + | |- | ||
| + | |a||Beurteilung erstmals aus Anlass einer Autopsie | ||
|- | |- | ||
|T||(Primär-)Tumor | |T||(Primär-)Tumor | ||
| Zeile 56: | Zeile 71: | ||
!TNM-Symbol!!Bedeutung!!gültig für | !TNM-Symbol!!Bedeutung!!gültig für | ||
|- | |- | ||
| − | |c/p||klinisch/pathologisch festgestellt||T, N, M | + | |c/u/ct/mr/p||klinisch (Ultraschall, CT, MRT)/pathologisch festgestellt||T, N, M |
|- | |- | ||
|C||Certainty Factor||T, N, M | |C||Certainty Factor||T, N, M | ||
|- | |- | ||
| − | |m||multipler Tumor (unquantifiziert) | + | |m||multipler Tumor (unquantifiziert)<br/>bzw. Angabe der Anzahl der Tumore||T |
| − | |||
| − | |||
| − | |||
| − | |||
|- | |- | ||
|sn||Sentinel Nodes||N | |sn||Sentinel Nodes||N | ||
| Zeile 83: | Zeile 94: | ||
|(kein)||Multiplizität (Anzahl der Tumorherde, numerisch)||T | |(kein)||Multiplizität (Anzahl der Tumorherde, numerisch)||T | ||
|- | |- | ||
| − | |i||immunhistochemisch nachgewiesene isolierte Tumorzellen (ITC)||N, M | + | |i-||immunhistochemisch nachgewiesene isolierte Tumorzellen (ITC)||N, M |
|- | |- | ||
| − | | | + | |i+||immunhistochemisch ausgeschlossene isolierte Tumorzellen (ITC)||N, M |
|- | |- | ||
| − | |(kein)||Anzahl entfernter und befallener Lymphknoten||N | + | |mol+||molekularpathologisch nachgewiesene isolierte Tumorzellen (ITC)||N, M |
| + | |- | ||
| + | |mol-||molekularpathologisch ausgeschlossene isolierte Tumorzellen (ITC)||N, M | ||
| + | |- | ||
| + | |(kein)||Anzahl entfernter und befallener Lymphknoten<br/>wird angegeben als RTO aus befallenen zu entfernten Lymphknoten||N | ||
|} | |} | ||
| − | Isolierte Tumorzellen sind einzeln oder in Clustern von bis zu 200 Mikrometern | + | Isolierte Tumorzellen sind einzeln oder in Clustern von bis zu 200 Zellen oder bis zu 200 Mikrometern Clusterdurchmesser vorkommende Tumorzellen, die bis auf das Melanom mit N0 kodiert werden. |
Mikrometastasen in Lymphknoten sind größer als 200 Mikrometer und kleiner als 2 mm, in aller Regel nur durch mikroskopische Untersuchung sichtbar. | Mikrometastasen in Lymphknoten sind größer als 200 Mikrometer und kleiner als 2 mm, in aller Regel nur durch mikroskopische Untersuchung sichtbar. | ||
| − | [[file:diag_tnm_v2.gif|TNM-Klassifikation]] | + | |
| + | [[file:diag_tnm_v2.gif|Modell für die TNM-Klassifikation]] | ||
Abbildung: Darstellung der TNM-Klassifikation | Abbildung: Darstellung der TNM-Klassifikation | ||
| + | |||
| + | {{AlertBox| | ||
| + | TODO: Abgleich mit TNM-Template aus APSR 2.0 erforderlich! | ||
| + | }} | ||
Anmerkung: Die Begriffe "davor" bzw. "dahinter" in obiger Grafik geben an, ob dieser Qualifier in der Darstellung als Tumorformel vor oder hinter der entsprechenden Kategorie (T, N oder M) aufgeführt wird. In der XML-Darstellung spielt dies jedoch keine Rolle. | Anmerkung: Die Begriffe "davor" bzw. "dahinter" in obiger Grafik geben an, ob dieser Qualifier in der Darstellung als Tumorformel vor oder hinter der entsprechenden Kategorie (T, N oder M) aufgeführt wird. In der XML-Darstellung spielt dies jedoch keine Rolle. | ||
| Zeile 102: | Zeile 122: | ||
+- UICC-Stadium, Tumorformel | +- UICC-Stadium, Tumorformel | ||
| − | |||
+- y | +- y | ||
+- r | +- r | ||
| − | +- T-Kategorie (c/p, C, m | + | +- a |
| − | + | +- T-Kategorie (c/u/ct/mr/p, C, m, m1/m2/m3, sm1/sm2/sm3) | |
| − | +- N-Kategorie (c/p, C | + | +- N-Kategorie (c/u/ct/mr/p, C, sn) |
| − | | +- | + | | +- (i+/i-: ITC durch Immunhistochemie nur für p) |
| − | + | | +- (mol+/mol-: ITC durch Molekularpathologie nur für p) | |
| − | | +- ITC durch Molekularpathologie nur für p | ||
| +- Anzahl der untersuchten und positiv bewerteten Knoten | | +- Anzahl der untersuchten und positiv bewerteten Knoten | ||
| − | +- M-Kategorie (c/p, C, Lokalisationscodes) | + | +- M-Kategorie (c/u/ct/mr/p, C, Lokalisationscodes) |
| − | | +- ITC durch Immunhistochemie | + | | +- (i+/i-: ITC durch Immunhistochemie) |
| − | | +- ITC durch Molekularpathologie | + | | +- (mol+/mol-: ITC durch Molekularpathologie) |
+- S (Serumtumormarker) | +- S (Serumtumormarker) | ||
+- L (Lymphgefäßinvasion) | +- L (Lymphgefäßinvasion) | ||
| Zeile 134: | Zeile 152: | ||
! Conf | ! Conf | ||
! Beschreibung | ! Beschreibung | ||
| + | |||
| + | |- | ||
| + | | | ||
| + | |bgcolor="ff8888"| | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | ! <h4>TNM-Klassifikation UICC-Stadien)</h4> | ||
|- | |- | ||
| Zeile 141: | Zeile 168: | ||
| | | | ||
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | | | + | | @classCode="OBS", @moodCode="EVN" |
|- | |- | ||
| Zeile 150: | Zeile 177: | ||
| II | | II | ||
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | | | + | | @root="1.2.276.0.76.3.1.131.1.10.3.5" |
| + | |||
|- | |- | ||
| 4 | | 4 | ||
| Zeile 185: | Zeile 213: | ||
| CD CWE | | CD CWE | ||
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | | | + | | @code="<b>tnmStage</b>", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
|- | |- | ||
| Zeile 212: | Zeile 240: | ||
|CD | |CD | ||
|1..1 | |1..1 | ||
| − | | | + | |R |
|Code für das UICC-Stadium<br> | |Code für das UICC-Stadium<br> | ||
| − | Value Set: 1.2.276.0.76.11.5 | + | Value Set: 1.2.276.0.76.11.5<br> |
| + | Ist dieses für eine Erkrankung nicht relevant, jedoch bestimmte TNM-Unterinformationen, wird @nullFlavor="NA" gesetzt.<br> | ||
| + | Ist es grundsätzlich für die Erkrankung relevant, jedoch nicht bekannt bzw. nicht vollständig ermittelbar, wird @nullFlavor="UNK" gesetzt.<br> | ||
|- | |- | ||
| Zeile 224: | Zeile 254: | ||
| optional | | optional | ||
| fix: @typeCode="SPRT" | | fix: @typeCode="SPRT" | ||
| + | |||
| + | |- | ||
| + | | | ||
| + | |bgcolor="ff8888"| | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | ! <h4>TNM y-Symbol (Präfix)</h4> | ||
|- | |- | ||
| Zeile 231: | Zeile 270: | ||
| | | | ||
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | | | + | | Stadium nach neoadjuvanter Therapie<br/> |
| − | + | @classCode="OBS", @moodCode="EVN" | |
|- | |- | ||
| Zeile 241: | Zeile 280: | ||
| CD CWE | | CD CWE | ||
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | | | + | | @code="tnmYSymbol", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
|- | |- | ||
| Zeile 270: | Zeile 309: | ||
| optional | | optional | ||
| fix: @typeCode="SPRT" | | fix: @typeCode="SPRT" | ||
| + | |||
| + | |- | ||
| + | | | ||
| + | |bgcolor="ff8888"| | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | ! <h4>TNM r-Symbol (Präfix)</h4> | ||
|- | |- | ||
| Zeile 277: | Zeile 325: | ||
| | | | ||
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | | | + | | Stadium eines Rezidive nach tumorfreiem Intervall<br/> |
| − | + | classCode="OBS", @moodCode="EVN" | |
|- | |- | ||
| Zeile 288: | Zeile 336: | ||
| 1..1 | | 1..1 | ||
| required | | required | ||
| − | | fix: @code=" | + | | fix: @code="tnmRSymbol", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
|- | |- | ||
| Zeile 296: | Zeile 344: | ||
|ED | |ED | ||
|1..1 | |1..1 | ||
| − | | | + | |R |
|textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert | |textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert | ||
| Zeile 305: | Zeile 353: | ||
|BL | |BL | ||
|1..1 | |1..1 | ||
| − | | | + | |R |
| | | | ||
| Zeile 314: | Zeile 362: | ||
| | | | ||
| 0..1 | | 0..1 | ||
| − | | | + | | F |
| − | | | + | | @typeCode="SPRT" |
| + | |||
| + | |- | ||
| + | | 5 | ||
| + | |bgcolor="ff8888"| | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | ! <h4>TNM a-Symbol (Präfix)</h4> | ||
|- | |- | ||
| Zeile 323: | Zeile 380: | ||
| | | | ||
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | | | + | | Beurteilung erstmals aus Anlass einer Autopsie<br/> |
| − | + | classCode="OBS", @moodCode="EVN" | |
|- | |- | ||
| Zeile 334: | Zeile 391: | ||
| 1..1 | | 1..1 | ||
| required | | required | ||
| − | | fix: @code=" | + | | fix: @code="tnmASymbol", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
|- | |- | ||
| Zeile 342: | Zeile 399: | ||
|ED | |ED | ||
|1..1 | |1..1 | ||
| − | | | + | |R |
|textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert | |textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert | ||
| Zeile 351: | Zeile 408: | ||
|BL | |BL | ||
|1..1 | |1..1 | ||
| − | | | + | |R |
| | | | ||
| Zeile 360: | Zeile 417: | ||
| | | | ||
| 0..1 | | 0..1 | ||
| − | | | + | | F |
| − | | | + | | @typeCode="SPRT" |
| + | |||
| + | |- | ||
| + | | 5 | ||
| + | |bgcolor="ff8888"| | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | ! <h4>T: Tumor</h4> | ||
|- | |- | ||
| Zeile 369: | Zeile 435: | ||
| | | | ||
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | | | + | | @classCode="OBS", @moodCode="EVN" |
| − | |||
|- | |- | ||
| Zeile 379: | Zeile 444: | ||
| CD CWE | | CD CWE | ||
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | | | + | | @code="<b>tnmT</b>", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
|- | |- | ||
| Zeile 407: | Zeile 472: | ||
|0..1 | |0..1 | ||
|optional | |optional | ||
| − | |Art der Sicherung (klinisch, pathologisch) | + | |<h4>Art der Sicherung (qualifier)</h4> |
| − | + | (klinisch (Ultraschall, CT, MRT), pathologisch) | |
|- | |- | ||
| 8 | | 8 | ||
| Zeile 415: | Zeile 480: | ||
| CV | | CV | ||
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | | | + | | @code="tnmCp", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
|- | |- | ||
| Zeile 424: | Zeile 489: | ||
|CV | |CV | ||
|1..1 | |1..1 | ||
| − | | | + | |R |
|Value Set: 1.2.276.0.76.11.9 | |Value Set: 1.2.276.0.76.11.9 | ||
| Zeile 433: | Zeile 498: | ||
|CR | |CR | ||
|0..1 | |0..1 | ||
| − | | | + | |O |
| − | |C-Faktor (Certainty) | + | |<h4>C-Faktor (Certainty) (qualifier)</h4> |
|- | |- | ||
| Zeile 442: | Zeile 507: | ||
| CV | | CV | ||
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | | | + | | @code="tnmCFactor", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
|- | |- | ||
| Zeile 460: | Zeile 525: | ||
|CR | |CR | ||
|0..1 | |0..1 | ||
| − | | | + | |O |
| − | |m-Symbol: Multiplizität | + | |<h4>m-Symbol: Multiplizität (multiple tumor) (qualifier)</h4> |
|- | |- | ||
| Zeile 469: | Zeile 534: | ||
| CV | | CV | ||
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | | | + | | @code="<b>tnmMSymbol</b>", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
|- | |- | ||
| Zeile 476: | Zeile 541: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|value | |value | ||
| − | | | + | |BL |
|1..1 | |1..1 | ||
| − | | | + | |R |
| − | | | + | | |
|- | |- | ||
| − | |7 | + | | 7 |
| − | |bgcolor="ff8888"|act | + | |bgcolor="ff8888"| act |
| − | |qualifier | + | | qualifier |
| − | |CR | + | | CR |
| − | |0..1 | + | | 0..1 |
| − | | | + | | required |
| − | | | + | | <h4>Multiplizität (numerisch): Anzahl der Tumorherde (qualifier)</h4> |
|- | |- | ||
| 8 | | 8 | ||
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| − | | | + | | code |
| CV | | CV | ||
| 1..1 | | 1..1 | ||
| required | | required | ||
| − | | fix: @code=" | + | | fix: @code="<b>tnmMultiplicity</b>", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
|- | |- | ||
| Zeile 503: | Zeile 568: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|value | |value | ||
| − | | | + | |INT.NONNEG |
|1..1 | |1..1 | ||
|required | |required | ||
| − | | | + | | |
|- | |- | ||
| Zeile 514: | Zeile 579: | ||
|CR | |CR | ||
|0..1 | |0..1 | ||
| − | | | + | |O |
| − | | | + | |<h4>Mucosainfiltration (qualifier)</h4> |
|- | |- | ||
| Zeile 523: | Zeile 588: | ||
| CV | | CV | ||
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | | | + | | @code="<b>tnmMucosaSymbol</b>", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
|- | |- | ||
| Zeile 541: | Zeile 606: | ||
|CR | |CR | ||
|0..1 | |0..1 | ||
| − | | | + | |O |
| − | | | + | |<h4>Submucosainfiltration (qualifier)</h4> |
|- | |- | ||
| Zeile 550: | Zeile 615: | ||
| CV | | CV | ||
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | | | + | | @code="<b>tnmSubmucosaSymbol</b>", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
|- | |- | ||
| Zeile 563: | Zeile 628: | ||
|- | |- | ||
| − | | | + | | 4 |
|bgcolor="ffaaaa"| rel | |bgcolor="ffaaaa"| rel | ||
| entryRelationship | | entryRelationship | ||
| Zeile 572: | Zeile 637: | ||
|- | |- | ||
| − | | | + | | |
| + | |bgcolor="ff8888"| | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | ! <h4>N: Nodalstatus</h4> | ||
| + | |||
| + | |- | ||
| + | | 5 | ||
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| observation | | observation | ||
| | | | ||
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | | | + | | @classCode="OBS", @moodCode="EVN" |
| − | |||
|- | |- | ||
| − | | | + | | 6 |
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| code | | code | ||
| Zeile 588: | Zeile 661: | ||
| 1..1 | | 1..1 | ||
| required | | required | ||
| − | | fix: @code=" | + | | fix: @code="tnmN", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
|- | |- | ||
| − | | | + | |6 |
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
| − | | | + | |text |
| − | | | + | |ED |
|1..1 | |1..1 | ||
|required | |required | ||
| − | | | + | |textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert |
| + | |||
| + | |- | ||
| + | |6 | ||
| + | |bgcolor="ff8888"|act | ||
| + | |value | ||
| + | |CD | ||
| + | |1..1 | ||
| + | |required | ||
| + | |Value Set: 1.2.276.0.76.11.2 | ||
|- | |- | ||
| − | | | + | |7 |
| − | |bgcolor=" | + | |bgcolor="ff8888"|act |
| − | | | + | |qualifier |
| − | | | + | |CR |
| − | | 0..1 | + | |0..1 |
| − | | optional | + | |optional |
| − | | | + | |<h4>Art der Sicherung (qualifier)</h4> |
| + | Art der Sicherung (klinisch (Ultraschall, CT, MRT), pathologisch) | ||
|- | |- | ||
| − | | | + | | 8 |
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| − | | | + | | name |
| − | | | + | | CV |
| 1..1 | | 1..1 | ||
| required | | required | ||
| − | | | + | | fix: @code="<b>tnmCp</b>", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| − | fix: @ | ||
|- | |- | ||
| − | | | + | |8 |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
| − | | | + | |value |
| − | | | + | |CV |
|1..1 | |1..1 | ||
|required | |required | ||
| − | | | + | |Value Set: 1.2.276.0.76.11.9 |
|- | |- | ||
| − | | | + | |7 |
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
| − | + | |qualifier | |
| − | + | |CR | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |qualifier | ||
| − | |CR | ||
|0..1 | |0..1 | ||
|optional | |optional | ||
| − | | | + | | <h4>C-Faktor (Certainty)</h4> |
|- | |- | ||
| Zeile 661: | Zeile 725: | ||
| 1..1 | | 1..1 | ||
| required | | required | ||
| − | | fix: @code=" | + | | fix: @code="tnmCFactor", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
|- | |- | ||
| Zeile 670: | Zeile 734: | ||
|1..1 | |1..1 | ||
|required | |required | ||
| − | |Value Set: 1.2.276.0.76. | + | |TODO: Value Set OID; fix: @codeSystem="1.2.276.0.76.5.341" |
|- | |- | ||
| Zeile 679: | Zeile 743: | ||
|0..1 | |0..1 | ||
|optional | |optional | ||
| − | | | + | |sn-Symbol (Sentinel Nodes) |
|- | |- | ||
| Zeile 688: | Zeile 752: | ||
| 1..1 | | 1..1 | ||
| required | | required | ||
| − | | fix: @code=" | + | | fix: @code="<b>tnmSn</b>", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
|- | |- | ||
| Zeile 697: | Zeile 761: | ||
|1..1 | |1..1 | ||
|required | |required | ||
| − | |TODO: Value Set OID; fix: @codeSystem=" | + | |TODO: Value Set OID; fix: @codeSystem="2.16.840.1.113883.15.6" |
|- | |- | ||
| − | |7 | + | | 7 |
| − | |bgcolor="ff8888"|act | + | |bgcolor="ff8888"| act |
| − | |qualifier | + | | qualifier |
| − | | | + | | |
| − | | | + | | 1..1 |
| − | | | + | | required |
| − | | | + | | <h4>i (Suffix): ITC durch Immunhistochemie (qualifier)</h4> <br/> |
| − | + | ||
| − | |- | + | |- |
| − | | 8 | + | | 8 |
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| name | | name | ||
| − | | | + | | CD CWE |
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | | | + | | @code="<b>tnmI</b>", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
|- | |- | ||
| Zeile 721: | Zeile 785: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|value | |value | ||
| − | | | + | |CD |
|1..1 | |1..1 | ||
|required | |required | ||
| − | |TODO: Value Set OID; fix: @codeSystem="2.16.840.1.113883.15.6" | + | |TODO: Value Set OID (Code "i+" oder "i-"); fix: @codeSystem="2.16.840.1.113883.15.6" |
|- | |- | ||
| − | |7 | + | | 7 |
| − | |bgcolor="ff8888"|act | + | |bgcolor="ff8888"| act |
| − | |qualifier | + | | qualifier |
| − | | | + | | |
| − | | | + | | 1..1 |
| − | | | + | | required |
| − | | | + | | <h4>mol (Suffix): ITC durch Molekularpathologie</h4> <br/> |
| + | fix: @classCode="OBS", @moodCode="EVN" | ||
|- | |- | ||
| Zeile 739: | Zeile 804: | ||
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| name | | name | ||
| − | | | + | | CD CWE |
| 1..1 | | 1..1 | ||
| required | | required | ||
| − | | fix: @code=" | + | | fix: @code="<b>tnmMol</b>", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
|- | |- | ||
| Zeile 748: | Zeile 813: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|value | |value | ||
| − | | | + | |CD |
|1..1 | |1..1 | ||
|required | |required | ||
| − | |TODO: Value Set OID; fix: @codeSystem="2.16.840.1.113883.15.6" | + | |TODO: Value Set OID (Code "mol+" oder "mol-"); fix: @codeSystem="2.16.840.1.113883.15.6" |
|- | |- | ||
| Zeile 769: | Zeile 834: | ||
| 1..1 | | 1..1 | ||
| required | | required | ||
| − | | | + | | <h4>Anzahl Lymphknoten</h4> |
| − | + | Diese Observation kann auch standalone bzw. in anderem Kontext verwendet werden. | |
|- | |- | ||
| Zeile 779: | Zeile 844: | ||
| 1..1 | | 1..1 | ||
| required | | required | ||
| − | | fix: @code=" | + | | für die Angabe von Lymphknoten allgemein fix: @code="21894-1", @codeSystem="2.16.840.1.113883.6.1"<br> |
| + | für die Angabe von Sentinel Nodes fix: @code="SentinelNodes", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" | ||
|- | |- | ||
| Zeile 785: | Zeile 851: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|value | |value | ||
| − | | | + | |RTO_PQ_PQ |
| + | |1..1 | ||
| + | |required | ||
| + | | | ||
| + | |||
| + | |- | ||
| + | |9 | ||
| + | |bgcolor="ff8888"|act | ||
| + | |numerator | ||
| + | |PQ | ||
| + | |1..1 | ||
| + | |required | ||
| + | |@value: Anzahl der befallenen Lymphknoten | ||
| + | fix: @unit="1" | ||
| + | |||
| + | |- | ||
| + | |9 | ||
| + | |bgcolor="ff8888"|act | ||
| + | |denominator | ||
| + | |PQ | ||
|1..1 | |1..1 | ||
|required | |required | ||
| − | | | + | |@value: Anzahl der entfernten (untersuchten) Lymphknoten |
| + | fix: @unit="1" | ||
|- | |- | ||
| − | | | + | | 4 |
|bgcolor="ffaaaa"| rel | |bgcolor="ffaaaa"| rel | ||
| entryRelationship | | entryRelationship | ||
| Zeile 800: | Zeile 886: | ||
|- | |- | ||
| − | | | + | | |
| + | |bgcolor="ff8888"| | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | ! <h4>M: Metastasen</h4> | ||
| + | |||
| + | |- | ||
| + | | 5 | ||
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| observation | | observation | ||
| | | | ||
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | | | + | | @classCode="OBS", @moodCode="EVN" |
| − | |||
|- | |- | ||
| − | | | + | | 6 |
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| code | | code | ||
| Zeile 816: | Zeile 910: | ||
| 1..1 | | 1..1 | ||
| required | | required | ||
| − | | fix: @code=" | + | | fix: @code="tnmM", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| + | |||
| + | |- | ||
| + | |6 | ||
| + | |bgcolor="ff8888"|act | ||
| + | |text | ||
| + | |ED | ||
| + | |1..1 | ||
| + | |required | ||
| + | |textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert | ||
|- | |- | ||
| − | | | + | |6 |
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|value | |value | ||
| Zeile 825: | Zeile 928: | ||
|1..1 | |1..1 | ||
|required | |required | ||
| − | | | + | |Value Set: 1.2.276.0.76.11.3 |
|- | |- | ||
| − | | | + | |7 |
| − | |bgcolor=" | + | |bgcolor="ff8888"|act |
| − | | | + | |qualifier |
| − | | | + | |CR |
| − | | 0..1 | + | |0..1 |
| − | | optional | + | |optional |
| − | | | + | |<h4>Art der Sicherung (qualifier)</h4> |
| + | Art der Sicherung (klinisch (Ultraschall, CT, MRT), pathologisch) | ||
|- | |- | ||
| − | | | + | | 8 |
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| − | | | + | | name |
| − | | | + | | CV |
| 1..1 | | 1..1 | ||
| required | | required | ||
| − | | | + | | fix: @code="tnmCp", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| − | |||
|- | |- | ||
| − | | | + | |8 |
| − | |bgcolor=" | + | |bgcolor="ff8888"|act |
| − | | | + | |value |
| − | | | + | |CV |
| − | | | + | |1..1 |
| − | | | + | |required |
| − | | | + | |Value Set: 1.2.276.0.76.11.9 |
|- | |- | ||
| − | | | + | |7 |
| − | |bgcolor="ff8888"| act | + | |bgcolor="ff8888"|act |
| − | | | + | |qualifier |
| − | | | + | |CR |
| − | | | + | |0..1 |
| − | | | + | |optional |
| − | | | + | |<h4>C-Faktor (Certainty)</h4> |
| − | |||
|- | |- | ||
| − | | | + | | 8 |
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| − | | | + | | name |
| − | | | + | | CV |
| 1..1 | | 1..1 | ||
| required | | required | ||
| − | | fix: @code=" | + | | fix: @code="tnmCFactor", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
|- | |- | ||
| − | | | + | |8 |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|value | |value | ||
| − | | | + | |CV |
|1..1 | |1..1 | ||
|required | |required | ||
| − | |Value Set: 1.2.276.0.76. | + | |TODO: Value Set OID; fix: @codeSystem="1.2.276.0.76.5.341" |
|- | |- | ||
| Zeile 897: | Zeile 990: | ||
|qualifier | |qualifier | ||
|CR | |CR | ||
| − | |0.. | + | |0..* |
|optional | |optional | ||
| − | | | + | |Lokalisationscodes |
|- | |- | ||
| Zeile 908: | Zeile 1.001: | ||
| 1..1 | | 1..1 | ||
| required | | required | ||
| − | | fix: @code=" | + | | fix: @code="21920-4", @codeSystem="2.16.840.1.113883.6.1" |
|- | |- | ||
| Zeile 917: | Zeile 1.010: | ||
|1..1 | |1..1 | ||
|required | |required | ||
| − | |Value Set: 1.2.276.0.76. | + | |TODO: Value Set OID; fix: @codeSystem="1.2.276.0.76.5.401" |
|- | |- | ||
| − | |7 | + | | 7 |
| − | |bgcolor="ff8888"|act | + | |bgcolor="ff8888"| act |
| − | |qualifier | + | | qualifier |
| − | |CR | + | | CR |
| − | | | + | | 1..1 |
| − | | | + | | required |
| − | | | + | | <h4>i (Suffix): ITC durch Immunhistochemie (qualifier)</h4> |
| + | fix: @classCode="OBS", @moodCode="EVN" | ||
|- | |- | ||
| Zeile 932: | Zeile 1.026: | ||
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| name | | name | ||
| − | | | + | | CD CWE |
| 1..1 | | 1..1 | ||
| required | | required | ||
| − | | fix: @code=" | + | | fix: @code="tnmI", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
|- | |- | ||
| Zeile 941: | Zeile 1.035: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|value | |value | ||
| − | | | + | |CD |
|1..1 | |1..1 | ||
|required | |required | ||
| − | |TODO: Value Set OID; fix: @codeSystem=" | + | |TODO: Value Set OID (Code "i+" oder "i-"); fix: @codeSystem="2.16.840.1.113883.15.6" |
|- | |- | ||
| − | |7 | + | | 7 |
| − | |bgcolor="ff8888"|act | + | |bgcolor="ff8888"| act |
| − | |qualifier | + | | qualifier |
| − | |CR | + | | CR |
| − | | | + | | 1..1 |
| − | | | + | | required |
| − | | | + | | <h4>mol (Suffix): RT-PCR (ITC durch Molekularpathologie) (qualifier)</h4> |
| + | fix: @classCode="OBS", @moodCode="EVN" | ||
|- | |- | ||
| Zeile 959: | Zeile 1.054: | ||
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| name | | name | ||
| − | | | + | | CD CWE |
| 1..1 | | 1..1 | ||
| required | | required | ||
| − | | fix: @code=" | + | | fix: @code="tnmMol", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
|- | |- | ||
| Zeile 968: | Zeile 1.063: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|value | |value | ||
| − | | | + | |CD |
|1..1 | |1..1 | ||
|required | |required | ||
| − | |TODO: Value Set OID; fix: @codeSystem=" | + | |TODO: Value Set OID (Code "mol+" oder "mol-"); fix: @codeSystem="2.16.840.1.113883.15.6" |
|- | |- | ||
| − | | | + | | 4 |
|bgcolor="ffaaaa"| rel | |bgcolor="ffaaaa"| rel | ||
| entryRelationship | | entryRelationship | ||
| Zeile 983: | Zeile 1.078: | ||
|- | |- | ||
| − | | | + | | |
| + | |bgcolor="ff8888"| | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | ! <h4>S: Serumtumormarker</h4> | ||
| + | |||
| + | |- | ||
| + | | 5 | ||
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| observation | | observation | ||
| | | | ||
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | | | + | | @classCode="OBS", @moodCode="EVN" |
| − | |||
|- | |- | ||
| − | | | + | | 6 |
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| code | | code | ||
| Zeile 999: | Zeile 1.102: | ||
| 1..1 | | 1..1 | ||
| required | | required | ||
| − | | fix: @code=" | + | | fix: @code="tnmS", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
|- | |- | ||
| − | | | + | |6 |
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
| − | |value | + | |text |
| + | |ED | ||
| + | |1..1 | ||
| + | |required | ||
| + | |textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert | ||
| + | |||
| + | |- | ||
| + | |6 | ||
| + | |bgcolor="ff8888"|act | ||
| + | |value | ||
|CD | |CD | ||
|1..1 | |1..1 | ||
|required | |required | ||
| − | |TODO | + | |TODO Value Set OID |
|- | |- | ||
| − | | | + | | 4 |
|bgcolor="ffaaaa"| rel | |bgcolor="ffaaaa"| rel | ||
| entryRelationship | | entryRelationship | ||
| Zeile 1.020: | Zeile 1.132: | ||
|- | |- | ||
| − | | | + | | |
| + | |bgcolor="ff8888"| | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | ! <h4>L: Lymphgefäßinvasion</h4> | ||
| + | |||
| + | |- | ||
| + | | 5 | ||
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| observation | | observation | ||
| | | | ||
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | | | + | | @classCode="OBS", @moodCode="EVN" |
| − | |||
|- | |- | ||
| − | | | + | | 6 |
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| code | | code | ||
| Zeile 1.036: | Zeile 1.156: | ||
| 1..1 | | 1..1 | ||
| required | | required | ||
| − | | fix: @code=" | + | | fix: @code="tnmL", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| + | |||
| + | |- | ||
| + | |6 | ||
| + | |bgcolor="ff8888"|act | ||
| + | |text | ||
| + | |ED | ||
| + | |1..1 | ||
| + | |required | ||
| + | |textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert | ||
|- | |- | ||
| − | | | + | |6 |
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|value | |value | ||
| Zeile 1.045: | Zeile 1.174: | ||
|1..1 | |1..1 | ||
|required | |required | ||
| − | | | + | |Value Set: 1.2.276.0.76.11.7 |
|- | |- | ||
| Zeile 1.055: | Zeile 1.184: | ||
| optional | | optional | ||
| fix: @typeCode="SPRT" | | fix: @typeCode="SPRT" | ||
| + | |||
| + | |- | ||
| + | | | ||
| + | |bgcolor="ff8888"| | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | ! <h4>V: Veneninvasion</h4> | ||
|- | |- | ||
| Zeile 1.062: | Zeile 1.200: | ||
| | | | ||
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | | | + | | @classCode="OBS", @moodCode="EVN" |
| − | |||
|- | |- | ||
| Zeile 1.073: | Zeile 1.210: | ||
| 1..1 | | 1..1 | ||
| required | | required | ||
| − | | fix: @code=" | + | | fix: @code="tnmV", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
|- | |- | ||
| Zeile 1.091: | Zeile 1.228: | ||
|1..1 | |1..1 | ||
|required | |required | ||
| − | | | + | |Value Set: 1.2.276.0.76.11.6 |
|- | |- | ||
| Zeile 1.101: | Zeile 1.238: | ||
| optional | | optional | ||
| fix: @typeCode="SPRT" | | fix: @typeCode="SPRT" | ||
| + | |||
| + | |- | ||
| + | | | ||
| + | |bgcolor="ff8888"| | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | ! <h4>Pn: Perineuralinvasion</h4> | ||
|- | |- | ||
| Zeile 1.108: | Zeile 1.254: | ||
| | | | ||
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | | | + | | @classCode="OBS", @moodCode="EVN" |
| − | |||
|- | |- | ||
| Zeile 1.119: | Zeile 1.264: | ||
| 1..1 | | 1..1 | ||
| required | | required | ||
| − | | fix: @code=" | + | | fix: @code="tnmPn", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
|- | |- | ||
| Zeile 1.137: | Zeile 1.282: | ||
|1..1 | |1..1 | ||
|required | |required | ||
| − | |Value Set: 1.2.276.0.76.11. | + | |Value Set: 1.2.276.0.76.11.8 |
|- | |- | ||
| Zeile 1.147: | Zeile 1.292: | ||
| optional | | optional | ||
| fix: @typeCode="SPRT" | | fix: @typeCode="SPRT" | ||
| + | |||
| + | |- | ||
| + | | | ||
| + | |bgcolor="ff8888"| | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | !<h4>G: histopathologisches Grading</h4> | ||
|- | |- | ||
| Zeile 1.155: | Zeile 1.309: | ||
| 1..1 | | 1..1 | ||
| required | | required | ||
| − | | | + | | siehe [[cdaab2:Grading-Entry (Template)|Template Grading]] |
| − | |||
|- | |- | ||
| − | + | | 4 | |
| − | + | |bgcolor="ffaaaa"| rel | |
| − | + | | entryRelationship | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | | 4 | ||
| − | |bgcolor="ffaaaa"| rel | ||
| − | | entryRelationship | ||
| | | | ||
| 0..1 | | 0..1 | ||
| optional | | optional | ||
| fix: @typeCode="SPRT" | | fix: @typeCode="SPRT" | ||
| + | |||
| + | |- | ||
| + | | | ||
| + | |bgcolor="ff8888"| | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | | | ||
| + | ! <h4>R: Residualtumor</h4> | ||
|- | |- | ||
| Zeile 1.201: | Zeile 1.336: | ||
| 1..1 | | 1..1 | ||
| required | | required | ||
| − | | | + | | siehe [[cdaab2:Residualtumor-Entry_(Template)|Template]] |
| − | |||
| − | | | + | |} |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | ===Beispiel === | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | Das folgende Beispiel stellt das Maximum der möglichen Angaben dar, wohl wissend, dass dies aus medizinisch-fachlicher Sicht nicht korrekt ist. | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | + | <syntaxhighlight lang="XML"> | |
| − | + | <observation classCode="OBS" moodCode="EVN"> | |
| − | + | <templateId root="1.2.276.0.76.3.1.131.1.10.3.5"/> | |
| − | + | <id extension="tnm12345" | |
| − | + | root="1.2.276.0.76.3.1.131.1.4.3.9999.9999.999909"/> | |
| − | + | <code code="tnmStage" | |
| − | + | codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | |
| − | + | <text> | |
| + | Tumorklassifikation vom 26.03.2011: ayr UICC IV | ||
| + | </text> | ||
| + | <effectiveTime value="20110326"/> | ||
| + | <value xsi:type="CD" code="IV" | ||
| + | codeSystem="2.16.840.1.113883.15.6" /> | ||
| − | + | <entryRelationship typeCode="SPRT"> | |
| − | + | <observation classCode="OBS" moodCode="EVN"> | |
| − | + | <code code="tnmYSymbol" | |
| − | + | codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | |
| − | + | <text> | |
| − | + | y | |
| − | + | </text> | |
| − | + | <value xsi:type="BL" value="true" /> | |
| − | + | </observation> | |
| + | </entryRelationship> | ||
| − | + | <entryRelationship typeCode="SPRT"> | |
| − | + | <observation classCode="OBS" moodCode="EVN"> | |
| − | + | <code code="tnmRSymbol" | |
| − | + | codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | |
| − | + | <text> | |
| − | + | r | |
| − | + | </text> | |
| − | + | <value xsi:type="BL" value="true" /> | |
| + | </observation> | ||
| + | </entryRelationship> | ||
| − | + | <!-- T value --> | |
| − | + | <entryRelationship typeCode="SPRT"> | |
| − | + | <observation classCode="OBS" moodCode="EVN"> | |
| − | + | <code code="tnmT" | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | <entryRelationship typeCode=" | ||
| − | <observation classCode="OBS" moodCode="EVN"> | ||
| − | <code code="tnmT" | ||
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | ||
<text> | <text> | ||
| − | + | pT3am(4)(m1)(sm2)C4 | |
</text> | </text> | ||
<value xsi:type="CD" code="T3a" | <value xsi:type="CD" code="T3a" | ||
| Zeile 1.326: | Zeile 1.400: | ||
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | ||
<value xsi:type="CD" code="C4" | <value xsi:type="CD" code="C4" | ||
| − | codeSystem="2. | + | codeSystem="1.2.276.0.76.5.341"/> |
</qualifier> | </qualifier> | ||
<qualifier> | <qualifier> | ||
| Zeile 1.332: | Zeile 1.406: | ||
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | ||
<value xsi:type="CD" code="m" | <value xsi:type="CD" code="m" | ||
| + | codeSystem="2.16.840.1.113883.15.6"/> | ||
| + | </qualifier> | ||
| + | <qualifier> | ||
| + | <code code="tnmMultiplicity" | ||
| + | codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | ||
| + | <value xsi:type="INT.NONNEG" value="4" /> | ||
| + | </qualifier> | ||
| + | |||
| + | <qualifier> | ||
| + | <name code="tnmMucosaSymbol" | ||
| + | codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | ||
| + | <value xsi:type="CD" code="m1" | ||
| + | codeSystem="2.16.840.1.113883.15.6"/> | ||
| + | </qualifier> | ||
| + | <qualifier> | ||
| + | <name code="tnmSubmucosaSymbol" | ||
| + | codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | ||
| + | <value xsi:type="CD" code="sm2" | ||
codeSystem="2.16.840.1.113883.15.6"/> | codeSystem="2.16.840.1.113883.15.6"/> | ||
</qualifier> | </qualifier> | ||
| Zeile 1.337: | Zeile 1.429: | ||
</observation> | </observation> | ||
</entryRelationship> | </entryRelationship> | ||
| − | <entryRelationship typeCode=" | + | |
| + | <!-- N value --> | ||
| + | <entryRelationship typeCode="SPRT"> | ||
<observation classCode="OBS" moodCode="EVN"> | <observation classCode="OBS" moodCode="EVN"> | ||
<code code="tnmN" | <code code="tnmN" | ||
| Zeile 1.350: | Zeile 1.444: | ||
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | ||
<value xsi:type="CD" code="p" | <value xsi:type="CD" code="p" | ||
| − | codeSystem="2. | + | codeSystem="1.2.276.0.76.5.341"/> |
| + | </qualifier> | ||
| + | <qualifier> | ||
| + | <name code="tnmCFactor" | ||
| + | codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | ||
| + | <value xsi:type="CD" code="C5" | ||
| + | codeSystem="1.2.276.0.76.5.341"/> | ||
</qualifier> | </qualifier> | ||
<qualifier> | <qualifier> | ||
| Zeile 1.359: | Zeile 1.459: | ||
</qualifier> | </qualifier> | ||
<qualifier> | <qualifier> | ||
| − | < | + | <code code="tnmI" |
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | ||
| − | <value xsi:type=" | + | <value xsi:type="BL" code="true"/> |
| − | |||
</qualifier> | </qualifier> | ||
<qualifier> | <qualifier> | ||
| − | < | + | <code code="tnmMol" |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | ||
| − | <value xsi:type=" | + | <value xsi:type="BL" value="true"/> |
| − | |||
</qualifier> | </qualifier> | ||
</value> | </value> | ||
| + | <entryRelationship typeCode="SPRT"> | ||
| + | <observation classCode="OBS" moodCode="EVN"> | ||
| + | <code code="21894-1" codeSystem="2.16.840.1.113883.6.1"/> | ||
| + | <value xsi:type="RTO_PQ_PQ"> | ||
| + | <numerator value="2" unit="1"/> | ||
| + | <denominator value="12" unit="1"/> | ||
| + | </value> | ||
| + | </observation> | ||
| + | </entryRelationship> | ||
</observation> | </observation> | ||
</entryRelationship> | </entryRelationship> | ||
| − | <entryRelationship typeCode=" | + | |
| + | <!-- M value --> | ||
| + | <entryRelationship typeCode="SPRT"> | ||
<observation classCode="OBS" moodCode="EVN"> | <observation classCode="OBS" moodCode="EVN"> | ||
<code code="tnmM" | <code code="tnmM" | ||
| Zeile 1.392: | Zeile 1.495: | ||
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | ||
<value xsi:type="CD" code="c" | <value xsi:type="CD" code="c" | ||
| − | codeSystem="2. | + | codeSystem="1.2.276.0.76.5.341"/> |
</qualifier> | </qualifier> | ||
<qualifier> | <qualifier> | ||
| − | <name code=" | + | <name code="tnmCFactor" |
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | ||
| − | <value xsi:type="CD" code=" | + | <value xsi:type="CD" code="C2" |
| − | codeSystem="2. | + | codeSystem="1.2.276.0.76.5.341"/> |
</qualifier> | </qualifier> | ||
<qualifier> | <qualifier> | ||
<name code="tnmI" | <name code="tnmI" | ||
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | ||
| − | <value xsi:type=" | + | <value xsi:type="BL" value="true" /> |
| − | |||
</qualifier> | </qualifier> | ||
<qualifier> | <qualifier> | ||
| − | < | + | <code code="tnmMol" |
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | ||
| − | <value xsi:type=" | + | <value xsi:type="BL" value="true" /> |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
</qualifier> | </qualifier> | ||
</value> | </value> | ||
</observation> | </observation> | ||
</entryRelationship> | </entryRelationship> | ||
| − | <entryRelationship typeCode=" | + | |
| + | <!-- further attributes --> | ||
| + | <entryRelationship typeCode="SPRT"> | ||
<observation classCode="OBS" moodCode="EVN"> | <observation classCode="OBS" moodCode="EVN"> | ||
<code code="tnmS" | <code code="tnmS" | ||
| Zeile 1.432: | Zeile 1.529: | ||
</observation> | </observation> | ||
</entryRelationship> | </entryRelationship> | ||
| − | <entryRelationship typeCode=" | + | <entryRelationship typeCode="SPRT"> |
<observation classCode="OBS" moodCode="EVN"> | <observation classCode="OBS" moodCode="EVN"> | ||
<code code="tnmL" | <code code="tnmL" | ||
| Zeile 1.443: | Zeile 1.540: | ||
</observation> | </observation> | ||
</entryRelationship> | </entryRelationship> | ||
| − | <entryRelationship typeCode=" | + | <entryRelationship typeCode="SPRT"> |
<observation classCode="OBS" moodCode="EVN"> | <observation classCode="OBS" moodCode="EVN"> | ||
<code code="tnmV" | <code code="tnmV" | ||
| Zeile 1.454: | Zeile 1.551: | ||
</observation> | </observation> | ||
</entryRelationship> | </entryRelationship> | ||
| − | <entryRelationship typeCode=" | + | <entryRelationship typeCode="SPRT"> |
<observation classCode="OBS" moodCode="EVN"> | <observation classCode="OBS" moodCode="EVN"> | ||
<code code="tnmPn" | <code code="tnmPn" | ||
| Zeile 1.465: | Zeile 1.562: | ||
</observation> | </observation> | ||
</entryRelationship> | </entryRelationship> | ||
| − | <entryRelationship typeCode=" | + | <entryRelationship typeCode="SPRT"> |
<observation classCode="OBS" moodCode="EVN"> | <observation classCode="OBS" moodCode="EVN"> | ||
| − | <code code=" | + | <code code="336" |
| − | codeSystem="2.16.840.1.113883. | + | codeSystem="2.16.840.1.113883.3.7.1.0"/> |
| − | + | <value xsi:type="CD" code="2" | |
| − | + | codeSystem="1.2.276.0.76.5.336"/> | |
| − | |||
| − | <value xsi:type=" | ||
</observation> | </observation> | ||
</entryRelationship> | </entryRelationship> | ||
| − | <entryRelationship typeCode=" | + | <entryRelationship typeCode="SPRT"> |
<observation classCode="OBS" moodCode="EVN"> | <observation classCode="OBS" moodCode="EVN"> | ||
| − | < | + | <templateId root="1.2.276.0.76.3.1.131.1.10.3.7"/> |
| − | codeSystem="2. | + | <id extension="1234041" |
| + | root="1.2.276.0.76.3.1.131.1.4.3.9999.9999.999919"/> | ||
| + | <code code="tnmR" codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | ||
<text> | <text> | ||
| − | + | gesamt R1, Fernmetastasen vorhanden | |
</text> | </text> | ||
| − | <value xsi:type=" | + | <effectiveTime value="20110326"/> |
| − | + | <value xsi:type="CD" code="R1" | |
| − | + | codeSystem="2.16.840.1.113883.15.6"> | |
| − | + | <qualifier> | |
| − | + | <name code="tnmRDomain" | |
| − | + | codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | |
| − | + | <value xsi:type="CD" code="G" codeSystem="1.2.276.0.76.5.414"/> | |
| − | + | </qualifier> | |
| − | + | <qualifier> | |
| − | </ | + | <name code="tnmRLocation" |
| − | <value xsi:type=" | + | codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> |
| − | + | <value xsi:type="CD" code="L" | |
| − | + | codeSystem="1.2.276.0.76.5.415"/> | |
| − | + | </qualifier> | |
| − | + | </value> | |
| − | + | <entryRelationship typeCode="SPRT"> | |
| − | + | <observation classCode="OBS" moodCode="EVN"> | |
| − | + | <code code="residualIs" | |
| − | + | codeSystem="1.2.276.0.76.3.1.131.1.5.1 "/> | |
| − | + | <value xsi:type="BL" value="true"/> | |
| − | + | </observation> | |
| − | + | </entryRelationship> | |
| + | <entryRelationship typeCode="SPRT"> | ||
| + | <observation classCode="OBS" moodCode="EVN"> | ||
| + | <code code="residualGt1mm" | ||
| + | codeSystem="1.2.276.0.76.3.1.131.1.5.1 "/> | ||
| + | <value xsi:type="BL" value="true"/> | ||
| + | </observation> | ||
| + | </entryRelationship> | ||
| + | <entryRelationship typeCode="SPRT"> | ||
| + | <observation classCode="OBS" moodCode="EVN"> | ||
| + | <code code="residualCy+" | ||
| + | codeSystem="1.2.276.0.76.3.1.131.1.5.1 "/> | ||
| + | <value xsi:type="BL" value="false"/> | ||
| + | </observation> | ||
</entryRelationship> | </entryRelationship> | ||
</observation> | </observation> | ||
| Zeile 1.516: | Zeile 1.626: | ||
}} | }} | ||
| − | ==== | + | ====Stadiengruppierung nach UICC==== |
| − | |||
{| class="hl7table" | {| class="hl7table" | ||
| − | |||
|- | |- | ||
| − | ! | + | ! !! !!UICC!! !! !! |
|- | |- | ||
| − | + | !Code !!Beschreibung !!Bedeutung !!5. !!6. !!7. | |
|- | |- | ||
| − | | | + | |okk || ||TX, N0, M0 ||X ||X ||X |
|- | |- | ||
| − | | | + | |0 ||Carcinoma in situ ||Tis, N0, M0 ||X ||X ||X |
|- | |- | ||
| − | | | + | |0a || || ||X ||X ||X |
|- | |- | ||
| − | | | + | |0is || || ||X ||X ||X |
|- | |- | ||
| − | | | + | |I || || || ||X ||X |
|- | |- | ||
| − | | | + | |IA || ||T1, N0, M0 ||X ||X ||X |
|- | |- | ||
| − | | | + | |IA1 || || ||X ||X ||X |
|- | |- | ||
| − | | | + | |IA2 || || ||X ||X ||X |
|- | |- | ||
| − | | | + | |IB || ||T2, N0, M0 ||X ||X ||X |
|- | |- | ||
| − | | | + | |IB1 || || ||X ||X ||X |
|- | |- | ||
| − | | | + | |IB2 || || ||X ||X ||X |
|- | |- | ||
| − | | | + | |IC || || ||X ||X ||X |
|- | |- | ||
| − | | | + | |II || || ||X ||X ||X |
|- | |- | ||
| − | | | + | |IIA || ||T1, N1, M0 ||X ||X ||X |
|- | |- | ||
| − | | | + | |IIA1 || || || || ||X |
|- | |- | ||
| − | | | + | |IIA2 || || || || ||X |
|- | |- | ||
| − | | | + | |IIB || ||T2, N1, M0 ||X ||X ||X |
|- | |- | ||
| − | | | + | |IIC || ||T3, N0, M0 ||X ||X ||X |
|- | |- | ||
| − | | | + | |III || || ||X ||X ||X |
|- | |- | ||
| − | | | + | |rowspan="3"|IIIA || ||T1, N2, M0 ||X ||X ||X |
|- | |- | ||
| − | | | + | | ||T2, N2, M0 || || || |
|- | |- | ||
| − | | | + | | ||T3, N1,2, M0 || || || |
|- | |- | ||
| − | | | + | |rowspan=22"|IIIB || ||T4, jedes N, M0 ||X ||X ||X |
|- | |- | ||
| − | | | + | | || ||jedes T, N3, M0 || || || |
|- | |- | ||
| − | | | + | |IIIC || || ||X ||X ||X |
|- | |- | ||
| − | | | + | |IS || || ||X ||X ||X |
|- | |- | ||
| − | | | + | |IV || ||jedes T, jedes N, M1 ||X ||X ||X |
|- | |- | ||
| − | | | + | |IVA || || ||X ||X ||X |
|- | |- | ||
| − | | | + | |IVB || || ||X ||X ||X |
| − | |||
| − | |||
| − | |||
| − | |||
|- | |- | ||
| + | |IVC || || ||X ||X ||X | ||
|} | |} | ||
| − | Tabelle: Codes für | + | Tabelle: Codes für Stadiengruppierung nach UICC<sup>16</sup> <br> |
| − | Value Set (OID 1.2.276.0.76.11. | + | Value Set (OID 1.2.276.0.76.11.5)<br> |
UICC 5. Auflage (OID 2.16.840.1.113883.15.8)<br> | UICC 5. Auflage (OID 2.16.840.1.113883.15.8)<br> | ||
| − | UICC 6. Auflage | + | UICC 6. Auflage (OID 2.16.840.1.113883.15.7)<br> |
UICC 7. Auflage (OID 2.16.840.1.113883.15.6) | UICC 7. Auflage (OID 2.16.840.1.113883.15.6) | ||
| − | ==== | + | |
| + | ====Ausdehnung des Primärtumors (T)==== | ||
| + | Bekannte T-Kategorien (ohne Darstellung möglicher Zusätze). Die Beschreibung variiert je nach Entität. Der Code soll nur in Zusammenhang mit der Art der Tumorerkrankung übermittelt werden, da die Kodes mit unterschiedlichen Bedeutungen belegt sind (z.B. T1 entspricht 2-3 cm oder 2-4 cm). | ||
{| class="hl7table" | {| class="hl7table" | ||
| − | ! !! !! !! !! | + | ! !! !! !!UICC!! !! !! |
| + | |- | ||
| + | !Code!!Description!!Bedeutung!!5.!!6.!!7.!! SCT | ||
| + | |- | ||
| + | |Ta||Noninvasive papillary urothelial carcinoma ||Nicht invasiv ||X||X||X ||X | ||
| + | |- | ||
| + | |Tis||Carcinoma in situ||Nicht invasiv||X||X||X||X | ||
| + | |- | ||
| + | |Tis(pu)||in situ urothelial carcinoma of prostatic urethra||Nicht invasiv || || ||X ||X | ||
| + | |- | ||
| + | |Tis(pd)||in situ urothelial carcinoma of prostatic gland ducts||Nicht invasiv || || ||X ||X | ||
| + | |- | ||
| + | |T0||No evidence of primary tumour||Kein Primärtumor nach¬weisbar|| ||X||X||X | ||
|- | |- | ||
| − | + | |T1|| || ||X||X||X|| X | |
|- | |- | ||
| − | | | + | |T1mic|| ||Microinvasion||X||X|| ||X |
|- | |- | ||
| − | | | + | |T1mi|| ||Microinvasion|| || ||X || |
|- | |- | ||
| − | | | + | |T1a|| || ||X||X||X||X |
|- | |- | ||
| − | | | + | |T1a1|| || ||X||X||X||X |
|- | |- | ||
| − | | | + | |T1a2|| || ||X||X||X||X |
|- | |- | ||
| − | | | + | |T1b|| || ||X||X||X||X |
|- | |- | ||
| − | | | + | |T1b1|| || ||X||X||X||X |
|- | |- | ||
| − | | | + | |T1b2|| || ||X||X||X||X |
|- | |- | ||
| − | | | + | |T1c||||||X||X||X||X |
|- | |- | ||
| − | | | + | |T1d||||||||||X||X |
|- | |- | ||
| − | | | + | |T2||||||X||X||X||X |
|- | |- | ||
| − | | | + | |T2a||||||X||X||X||X |
|- | |- | ||
| − | | | + | |T2a1||||||||||X||X |
|- | |- | ||
| − | | | + | |T2a2||||||||||X||X |
|- | |- | ||
| − | | | + | |T2b||||||X||X||X||X |
|- | |- | ||
| − | | | + | |T2c||||||X||X||X||X |
|- | |- | ||
| − | | | + | |T2d||||||||||X||X |
|- | |- | ||
| − | | | + | |T3||||||X||X||X||X |
|- | |- | ||
| − | | | + | |T3a||||||X||X||X||X |
|- | |- | ||
| − | | | + | |T3b||||||X||X||X||X |
|- | |- | ||
| − | | | + | |T3c||||||X||X||X||X |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
|- | |- | ||
| − | + | |T3d||||||||X||X||X | |
|- | |- | ||
| − | | | + | |T4||||||X||X||X||X |
|- | |- | ||
| − | | | + | |T4a||||||X||X||X||X |
|- | |- | ||
| − | | | + | |T4b||||||X||X||X||X |
|- | |- | ||
| − | | | + | |T4c|| || ||X||X||X||X |
|- | |- | ||
| − | | | + | |T4d|| || ||X||X||X||X |
|- | |- | ||
| − | | | + | |T4e|| || || || ||X||X |
|- | |- | ||
| − | | | + | |TX||Primary tumour cannot be assessed||Stadium des Primärtumors kann nicht nachgewiesen werden.|| || || || |
| − | |||
| − | |||
|- | |- | ||
|} | |} | ||
| − | Tabelle | + | Tabelle: Codes für Ausdehnung des Primärtumors (T)<br> |
| − | Value Set (OID 1.2.276.0.76.11. | + | Value Set (OID 1.2.276.0.76.11.1)<br> |
UICC 5. Auflage (OID 2.16.840.1.113883.15.8)<br> | UICC 5. Auflage (OID 2.16.840.1.113883.15.8)<br> | ||
| − | UICC 6. Auflage (OID 2.16.840.1.113883.15.7)<br> | + | UICC 6. Auflage (OID 2.16.840.1.113883.15.7)<br> |
UICC 7. Auflage (OID 2.16.840.1.113883.15.6) | UICC 7. Auflage (OID 2.16.840.1.113883.15.6) | ||
| − | ==== | + | ====Nodalstatus (regionäre Lymphknoten) (N)==== |
| − | |||
| − | |||
| − | |||
{| class="hl7table" | {| class="hl7table" | ||
| − | !Code!!Description!!Bedeutung | + | ! !! !! !! !!UICC!! !! !! |
| + | |- | ||
| + | !Code!!Description!!Bedeutung!!Entität!!5.!!6.!!7.!!SCT | ||
|- | |- | ||
| − | | | + | |N0||No regional lymph node metastasis||Kein Lymphknotenbefall||alle||X||X||X||X |
|- | |- | ||
| − | | | + | |N1||||||||X||X||X||X |
|- | |- | ||
| − | | | + | |N1mi||lymph node micrometastasis||||alle||||X||X??|| |
|- | |- | ||
| − | | | + | |N1mi||Bilateral regional lymph node metastasis||||Vulva||||X||X ????|| |
|- | |- | ||
| − | | | + | |N1a||||||alle||X||X||X|| X |
|- | |- | ||
| − | | | + | |N1b||||||||X||X||X||X |
|- | |- | ||
| − | | | + | |N1b1||||||||X|||| ???||X |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
|- | |- | ||
| − | + | |N1b2||||||||X|||| ???||X | |
|- | |- | ||
| − | + | |N1b3||||||||X|||| ???||X | |
|- | |- | ||
| − | | | + | |N1b4||||||||X|||| ???||X |
|- | |- | ||
| − | | | + | |N1c||||||||||X||X ???|| |
|- | |- | ||
| − | | | + | |N2||||||||X||X||X||X |
|- | |- | ||
| − | | | + | |N2a||||||||X||X||X||X |
|- | |- | ||
| − | | | + | |N2b||||||||X||X||X||X |
|- | |- | ||
| − | | | + | |N2c||||||||X||X||X||X |
|- | |- | ||
| − | | | + | |N3||||||||X||X||X||X |
|- | |- | ||
| − | | | + | |N3a||||||||X||X||X|| |
|- | |- | ||
| − | | | + | |N3b||||||||X||X||X|| |
|- | |- | ||
| − | | | + | |N3c||||||||||X||X|| |
|- | |- | ||
| − | | | + | |NX||Regional lymph nodes cannot be assessed||Lymphknotenbefall unbekannt||||X||X||X|| |
|- | |- | ||
| − | | | + | |} |
| − | | | + | Tabelle: Codes für Nodalstatus (N)<br> |
| − | + | Value Set (OID 1.2.276.0.76.11.2)<br> | |
| + | UICC 5. Auflage (OID 2.16.840.1.113883.15.8)<br> | ||
| + | UICC 6. Auflage OID 2.16.840.1.113883.15.7)<br> | ||
| + | UICC 7. Auflage (OID 2.16.840.1.113883.15.6) | ||
| + | |||
| + | ====Fernmetastasen (M)==== | ||
| + | Der Code zur Beurteilung von Fernmetastasen soll nur in Zusammenhang mit der Art der Tumorerkrankung übermittelt werden | ||
| + | |||
| + | {| class="hl7table" | ||
| + | ! !! !! !! !!UICC!! !! | ||
|- | |- | ||
| − | + | !Code!!Description!!Bedeutung!!Entität!!5.!!6.!!7. | |
|- | |- | ||
| − | | | + | |M0||No distant metastasis||Fernmetastasen nicht vorhanden (für p nicht anwendbar)||alle||X||X||X |
|- | |- | ||
| − | | | + | |M1||Distant metastasis||Fernmetastasen vorhanden||alle||X||X||X |
|- | |- | ||
| − | | | + | |M1a||||||nur Ösophagus und Prostata||X||X||X |
|- | |- | ||
| − | | | + | |M1b||||||nur Ösophagus und Prostata||X||X||X |
|- | |- | ||
| − | | | + | |M1c||||||||X||X||X |
|- | |- | ||
| − | | | + | |M1d||||||||||||X |
|- | |- | ||
| − | | | + | |M1e||||||||||||X |
|- | |- | ||
| − | | | + | |MX||Distant metastasis cannot be assessed (für p nicht anwendbar) ||||alle||X||X||X |
|- | |- | ||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
|} | |} | ||
| − | Tabelle: Codes für | + | Tabelle 22: Codes für Fernmetastasen (M)<br> |
| − | Value Set (OID 1.2.276.0.76.11. | + | Value Set (OID 1.2.276.0.76.11.3)<br> |
UICC 5. Auflage (OID 2.16.840.1.113883.15.8)<br> | UICC 5. Auflage (OID 2.16.840.1.113883.15.8)<br> | ||
UICC 6. Auflage (OID 2.16.840.1.113883.15.7)<br> | UICC 6. Auflage (OID 2.16.840.1.113883.15.7)<br> | ||
UICC 7. Auflage (OID 2.16.840.1.113883.15.6) | UICC 7. Auflage (OID 2.16.840.1.113883.15.6) | ||
| + | |||
====Veneninvasion==== | ====Veneninvasion==== | ||
| Zeile 1.816: | Zeile 1.903: | ||
|LX||lmphatic invasion cannot be assessed||Lymphsysteminvasion nicht fest¬stellbar | |LX||lmphatic invasion cannot be assessed||Lymphsysteminvasion nicht fest¬stellbar | ||
|- | |- | ||
| − | |} | + | |} |
| − | Tabelle: Codes für Lymphgefäßinvasion<br> | + | Tabelle: Codes für Lymphgefäßinvasion<br> |
| − | Value Set (OID 1.2.276.0.76.11.7)<br> | + | Value Set (OID 1.2.276.0.76.11.7)<br> |
| − | UICC 5. Auflage (OID 2.16.840.1.113883.15.8)<br> | + | UICC 5. Auflage (OID 2.16.840.1.113883.15.8)<br> |
| − | UICC 6. Auflage (OID 2.16.840.1.113883.15.7)<br> | + | UICC 6. Auflage (OID 2.16.840.1.113883.15.7)<br> |
| − | UICC 7. Auflage (OID 2.16.840.1.113883.15.6) | + | UICC 7. Auflage (OID 2.16.840.1.113883.15.6) |
| − | + | ||
| − | ==== | + | ====Perineuralscheideninvasion==== |
| − | + | ||
| − | {| class="hl7table" | + | {| class="hl7table" |
| − | !Code!!Description!!Bedeutung | + | !Code!!Description!!Bedeutung |
| − | |- | + | |- |
| − | |Pn0||no perineural invasion ||keine perineurale Invasion | + | |Pn0||no perineural invasion ||keine perineurale Invasion |
| + | |- | ||
| + | |Pn1||Perineural invasion||perineurale Invasion | ||
| + | |- | ||
| + | |PnX||unknown||Unbekannt | ||
| + | |- | ||
| + | |} | ||
| + | Tabelle: Codes für Perineuralinvasion<br> | ||
| + | Value Set (OID 1.2.276.0.76.11.8)<br> | ||
| + | UICC 5. Auflage (OID 2.16.840.1.113883.15.8)<br> | ||
| + | UICC 6. Auflage (OID 2.16.840.1.113883.15.7)<br> | ||
| + | UICC 7. Auflage (OID 2.16.840.1.113883.15.6) | ||
| + | |||
| + | ====Unterscheidung autoptisch/klinisch/pathologisch==== | ||
| + | |||
| + | {| class="hl7table" | ||
| + | !Code!!Description!!Bedeutung | ||
| + | |- | ||
| + | |a||autoptisch || | ||
| + | |- | ||
| + | |c||Assessment according to clinical criteria||Beurteilung nach klinischen Kriterien | ||
| + | |- | ||
| + | |u||Assessment according to clinical criteria based on ultraound evidence||Beurteilung nach klinischen Kriterien / Ultraschall | ||
|- | |- | ||
| − | | | + | |ct||Assessment according to clinical criteria based on CT evidence||Beurteilung nach klinischen Kriterien /CT |
|- | |- | ||
| − | | | + | |mr||Assessment according to clinical criteria based on magnetic resonance evidence||Beurteilung nach klinischen Kriterien / MRT |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
|- | |- | ||
|p||Assessment according to the pathological results||nach dem pathologischen Befund | |p||Assessment according to the pathological results||nach dem pathologischen Befund | ||
Aktuelle Version vom 28. September 2017, 13:19 Uhr
Dieses Material ist Teil des Leitfadens Arztbrief 2.x.
|
Inhaltsverzeichnis
- 1 Entry: TNM-Klassifikation
- 1.1 Darstellung der TNM-Klassifikation in HL7
- 1.2 Erläuterung
- 1.2.1 TNM-Klassifikation UICC-Stadien)
- 1.2.2 TNM y-Symbol (Präfix)
- 1.2.3 TNM r-Symbol (Präfix)
- 1.2.4 TNM a-Symbol (Präfix)
- 1.2.5 T: Tumor
- 1.2.6 Art der Sicherung (qualifier)
- 1.2.7 C-Faktor (Certainty) (qualifier)
- 1.2.8 m-Symbol: Multiplizität (multiple tumor) (qualifier)
- 1.2.9 Multiplizität (numerisch): Anzahl der Tumorherde (qualifier)
- 1.2.10 Mucosainfiltration (qualifier)
- 1.2.11 Submucosainfiltration (qualifier)
- 1.2.12 N: Nodalstatus
- 1.2.13 Art der Sicherung (qualifier)
- 1.2.14 C-Faktor (Certainty)
- 1.2.15 i (Suffix): ITC durch Immunhistochemie (qualifier)
- 1.2.16 mol (Suffix): ITC durch Molekularpathologie
- 1.2.17 Anzahl Lymphknoten
- 1.2.18 M: Metastasen
- 1.2.19 Art der Sicherung (qualifier)
- 1.2.20 C-Faktor (Certainty)
- 1.2.21 i (Suffix): ITC durch Immunhistochemie (qualifier)
- 1.2.22 mol (Suffix): RT-PCR (ITC durch Molekularpathologie) (qualifier)
- 1.2.23 S: Serumtumormarker
- 1.2.24 L: Lymphgefäßinvasion
- 1.2.25 V: Veneninvasion
- 1.2.26 Pn: Perineuralinvasion
- 1.2.27 G: histopathologisches Grading
- 1.2.28 R: Residualtumor
- 1.3 Beispiel
- 1.4 verwendete Terminologie
- 1.4.1 Stadiengruppierung nach UICC
- 1.4.2 Ausdehnung des Primärtumors (T)
- 1.4.3 Nodalstatus (regionäre Lymphknoten) (N)
- 1.4.4 Fernmetastasen (M)
- 1.4.5 Veneninvasion
- 1.4.6 Lymphgefäßinvasion
- 1.4.7 Perineuralscheideninvasion
- 1.4.8 Unterscheidung autoptisch/klinisch/pathologisch
- 1.4.9 Beschränkung auf Mukosa
- 1.4.10 Beschränkung auf Submukosa
- 1.4.11 C-Faktor (Certainty, Diagnosesicherheit)
- 1.4.12 Lokalisation von Fernmetastasen
Entry: TNM-Klassifikation
| Template-Metadaten | |
| Template-Typ | Entry |
| Template ID | 1.2.276.0.76.3.1.131.1.10.3.5 ????? |
| generischeres Template | <Verweis auf das Template> |
| genutztes Templates | R-Klassifikation |
| abgeleitete Templates | cdapath:TNM-Klassifikation-Entry (Template) |
| Generelle Beschreibung | Hiermit wird auf Entry-Level die Übermittlung von Tumordiagnosedaten (UICC TNM-Klassifikation) definiert. |
| allg. Erläuterung | |
| Verhältnis zu IHE | neu: Abstimmung mit QRPH und AP |
| Ballotierungsstatus | in Arbeit |
| Erweiterbarkeit | geschlossen |
Darstellung der TNM-Klassifikation in HL7
Die Darstellung der TNM-Klassifikation in HL7 V3 erfolgt nach folgendem Schema. Es gibt eine zentrale Observation-Klasse, über welche das UICC-Stadium (als value) und/oder die Tumorformel (als Unterlement originalText dieses values) angegeben werden. Ist das UICC-Stadium nicht bekannt (z.B. weil bei einer pathologischen Befundung keine Angaben zu Metastasen existieren), wird im value der Haupt-Observation ein nullFlavor="NA" übermittelt. Die Tumorformel kann dennoch im originalText übermittelt werden.
Einzelangaben des werden ebenfalls in Observation-Klassen eingebunden, die über eine ActRelationShip-Klasse mit dem code SPRT mit der zentralen Observation-Klasse verbunden werden.
Diese Angaben sind (in Fachterminologie):
| TNM-Symbol | Bedeutung |
|---|---|
| y | nach neoadjuvanter Therapie |
| r | Beurteilung eines Rezidivs |
| a | Beurteilung erstmals aus Anlass einer Autopsie |
| T | (Primär-)Tumor |
| N | Nodus = Lymphknoten |
| M | Metastasen |
| S | Serumtumormarker (für bestimmte Tumorentitäten) |
| L | Lymphgefäßinvasion |
| V | Veneninvasion |
| Pn | Perineuralinvasion |
| G | Histopathologisches Grading |
| R | Residualtumor |
Weitere Angaben werden als qualifier für die jeweiligen values angegeben:
| TNM-Symbol | Bedeutung | gültig für |
|---|---|---|
| c/u/ct/mr/p | klinisch (Ultraschall, CT, MRT)/pathologisch festgestellt | T, N, M |
| C | Certainty Factor | T, N, M |
| m | multipler Tumor (unquantifiziert) bzw. Angabe der Anzahl der Tumore |
T |
| sn | Sentinel Nodes | N |
| m1, m2, m3 | auf Mukosa beschränkt, 1., 2. 3. Drittel | T, gilt nur für gastrointestinale Karzinome |
| sm1, sm2, sm3 | auf Submukosa beschränkt, 1., 2. 3. Drittel | T, gilt nur für gastrointestinale Karzinome |
| (Lokalisationscode) | Lokalisation der Metastasen | M |
Weiterhin existieren Zusatzangaben zu bestimmten Observations, die wiederum über eine ActRelationship als Observation angegeben werden:
| TNM-Symbol | Bedeutung | gültig für |
|---|---|---|
| (kein) | Multiplizität (Anzahl der Tumorherde, numerisch) | T |
| i- | immunhistochemisch nachgewiesene isolierte Tumorzellen (ITC) | N, M |
| i+ | immunhistochemisch ausgeschlossene isolierte Tumorzellen (ITC) | N, M |
| mol+ | molekularpathologisch nachgewiesene isolierte Tumorzellen (ITC) | N, M |
| mol- | molekularpathologisch ausgeschlossene isolierte Tumorzellen (ITC) | N, M |
| (kein) | Anzahl entfernter und befallener Lymphknoten wird angegeben als RTO aus befallenen zu entfernten Lymphknoten |
N |
Isolierte Tumorzellen sind einzeln oder in Clustern von bis zu 200 Zellen oder bis zu 200 Mikrometern Clusterdurchmesser vorkommende Tumorzellen, die bis auf das Melanom mit N0 kodiert werden. Mikrometastasen in Lymphknoten sind größer als 200 Mikrometer und kleiner als 2 mm, in aller Regel nur durch mikroskopische Untersuchung sichtbar.
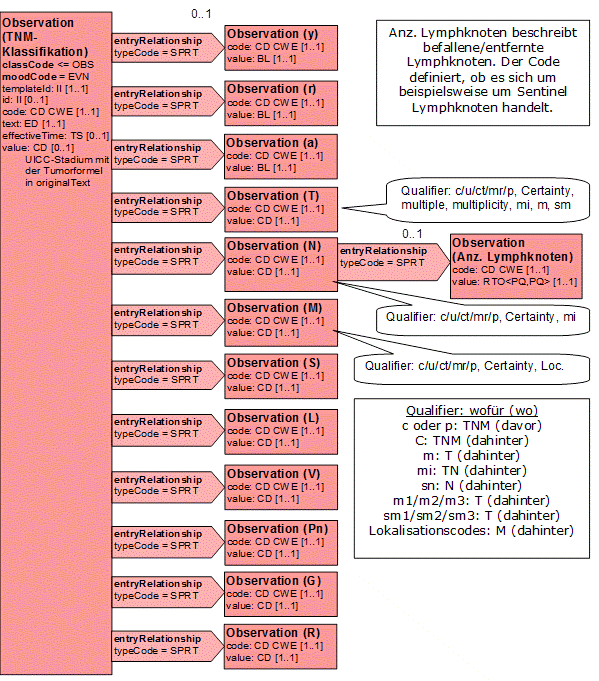
Abbildung: Darstellung der TNM-Klassifikation
|
|
TODO: Abgleich mit TNM-Template aus APSR 2.0 erforderlich! |
Anmerkung: Die Begriffe "davor" bzw. "dahinter" in obiger Grafik geben an, ob dieser Qualifier in der Darstellung als Tumorformel vor oder hinter der entsprechenden Kategorie (T, N oder M) aufgeführt wird. In der XML-Darstellung spielt dies jedoch keine Rolle.
Zum einfacheren Verständnis kann dies auch in einer hierarchischen Form dargestellt werden. Die jeweils anwendbaren Qualifier sind dabei in Klammern angegeben:
+- UICC-Stadium, Tumorformel +- y +- r +- a +- T-Kategorie (c/u/ct/mr/p, C, m, m1/m2/m3, sm1/sm2/sm3) +- N-Kategorie (c/u/ct/mr/p, C, sn) | +- (i+/i-: ITC durch Immunhistochemie nur für p) | +- (mol+/mol-: ITC durch Molekularpathologie nur für p) | +- Anzahl der untersuchten und positiv bewerteten Knoten +- M-Kategorie (c/u/ct/mr/p, C, Lokalisationscodes) | +- (i+/i-: ITC durch Immunhistochemie) | +- (mol+/mol-: ITC durch Molekularpathologie) +- S (Serumtumormarker) +- L (Lymphgefäßinvasion) +- V (Veneninvasion) +- Pn (Perineuralscheideninvasion) +- G (histopathologisches Grading) +- R (Residualtumor)
Abbildung: Baumdarstellung der TNM-Klassifikation
Erläuterung
| Lvl | RIM | Name | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|
TNM-Klassifikation UICC-Stadien) | ||||||
| 3 | act | observation | 1..1 | F | @classCode="OBS", @moodCode="EVN" | |
| 4 | act | templateId | II | 1..1 | F | @root="1.2.276.0.76.3.1.131.1.10.3.5" |
| 4 | act | id | II | 0..1 | optional | ID der diagnostischen Angabe |
| 5 | act | @root | 1..1 | optional | OID des sendenden Systems, um diagnostische Angaben eindeutig zu identifizieren | |
| 5 | act | @extension | 1..1 | optional | ID der diagnostischen Angabe im sendenden System | |
| 4 | act | code | CD CWE | 1..1 | F | @code="tnmStage", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 4 | act | text | ED | 1..1 | required | textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert |
| 4 | act | effectiveTime | TS | 0..1 | optional | Das Datum, an dem die Diagnose gestellt wurde |
| 4 | act | value | CD | 1..1 | R | Code für das UICC-Stadium Value Set: 1.2.276.0.76.11.5 |
| 4 | rel | entryRelationship | 0..1 | optional | fix: @typeCode="SPRT" | |
TNM y-Symbol (Präfix) | ||||||
| 5 | act | observation | 1..1 | F | Stadium nach neoadjuvanter Therapie @classCode="OBS", @moodCode="EVN" | |
| 6 | act | code | CD CWE | 1..1 | F | @code="tnmYSymbol", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 6 | act | text | ED | 1..1 | required | textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert |
| 6 | act | value | BL | 1..1 | required | |
| 4 | rel | entryRelationship | 0..1 | optional | fix: @typeCode="SPRT" | |
TNM r-Symbol (Präfix) | ||||||
| 5 | act | observation | 1..1 | F | Stadium eines Rezidive nach tumorfreiem Intervall classCode="OBS", @moodCode="EVN" | |
| 6 | act | code | CD CWE | 1..1 | required | fix: @code="tnmRSymbol", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 6 | act | text | ED | 1..1 | R | textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert |
| 6 | act | value | BL | 1..1 | R | |
| 4 | rel | entryRelationship | 0..1 | F | @typeCode="SPRT" | |
| 5 | TNM a-Symbol (Präfix) | |||||
| 5 | act | observation | 1..1 | F | Beurteilung erstmals aus Anlass einer Autopsie classCode="OBS", @moodCode="EVN" | |
| 6 | act | code | CD CWE | 1..1 | required | fix: @code="tnmASymbol", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 6 | act | text | ED | 1..1 | R | textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert |
| 6 | act | value | BL | 1..1 | R | |
| 4 | rel | entryRelationship | 0..1 | F | @typeCode="SPRT" | |
| 5 | T: Tumor | |||||
| 5 | act | observation | 1..1 | F | @classCode="OBS", @moodCode="EVN" | |
| 6 | act | code | CD CWE | 1..1 | F | @code="tnmT", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 6 | act | text | ED | 1..1 | required | textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert |
| 6 | act | value | CD | 1..1 | required | Value Set: 1.2.276.0.76.11.1 |
| 7 | act | qualifier | CR | 0..1 | optional | Art der Sicherung (qualifier)(klinisch (Ultraschall, CT, MRT), pathologisch) |
| 8 | act | name | CV | 1..1 | F | @code="tnmCp", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | CV | 1..1 | R | Value Set: 1.2.276.0.76.11.9 |
| 7 | act | qualifier | CR | 0..1 | O | C-Faktor (Certainty) (qualifier) |
| 8 | act | name | CV | 1..1 | F | @code="tnmCFactor", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | CV | 1..1 | required | TODO: Value Set OID; fix: @codeSystem="1.2.276.0.76.5.341" |
| 7 | act | qualifier | CR | 0..1 | O | m-Symbol: Multiplizität (multiple tumor) (qualifier) |
| 8 | act | name | CV | 1..1 | F | @code="tnmMSymbol", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | BL | 1..1 | R | |
| 7 | act | qualifier | CR | 0..1 | required | Multiplizität (numerisch): Anzahl der Tumorherde (qualifier) |
| 8 | act | code | CV | 1..1 | required | fix: @code="tnmMultiplicity", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | INT.NONNEG | 1..1 | required | |
| 7 | act | qualifier | CR | 0..1 | O | Mucosainfiltration (qualifier) |
| 8 | act | name | CV | 1..1 | F | @code="tnmMucosaSymbol", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | CV | 1..1 | required | TODO: Value Set OID (Code "m1", "m2" oder "m3"); fix: @codeSystem="2.16.840.1.113883.15.6" |
| 7 | act | qualifier | CR | 0..1 | O | Submucosainfiltration (qualifier) |
| 8 | act | name | CV | 1..1 | F | @code="tnmSubmucosaSymbol", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | CV | 1..1 | required | TODO: Value Set OID (Code "sm1", "sm2" oder "sm3"); fix: @codeSystem="2.16.840.1.113883.15.6" |
| 4 | rel | entryRelationship | 0..1 | optional | fix: @typeCode="SPRT" | |
N: Nodalstatus | ||||||
| 5 | act | observation | 1..1 | F | @classCode="OBS", @moodCode="EVN" | |
| 6 | act | code | CD CWE | 1..1 | required | fix: @code="tnmN", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 6 | act | text | ED | 1..1 | required | textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert |
| 6 | act | value | CD | 1..1 | required | Value Set: 1.2.276.0.76.11.2 |
| 7 | act | qualifier | CR | 0..1 | optional | Art der Sicherung (qualifier)Art der Sicherung (klinisch (Ultraschall, CT, MRT), pathologisch) |
| 8 | act | name | CV | 1..1 | required | fix: @code="tnmCp", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | CV | 1..1 | required | Value Set: 1.2.276.0.76.11.9 |
| 7 | act | qualifier | CR | 0..1 | optional | C-Faktor (Certainty) |
| 8 | act | name | CV | 1..1 | required | fix: @code="tnmCFactor", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | CV | 1..1 | required | TODO: Value Set OID; fix: @codeSystem="1.2.276.0.76.5.341" |
| 7 | act | qualifier | CR | 0..1 | optional | sn-Symbol (Sentinel Nodes) |
| 8 | act | name | CV | 1..1 | required | fix: @code="tnmSn", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | CV | 1..1 | required | TODO: Value Set OID; fix: @codeSystem="2.16.840.1.113883.15.6" |
| 7 | act | qualifier | 1..1 | required | i (Suffix): ITC durch Immunhistochemie (qualifier) | |
| 8 | act | name | CD CWE | 1..1 | F | @code="tnmI", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | CD | 1..1 | required | TODO: Value Set OID (Code "i+" oder "i-"); fix: @codeSystem="2.16.840.1.113883.15.6" |
| 7 | act | qualifier | 1..1 | required | mol (Suffix): ITC durch Molekularpathologiefix: @classCode="OBS", @moodCode="EVN" | |
| 8 | act | name | CD CWE | 1..1 | required | fix: @code="tnmMol", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | CD | 1..1 | required | TODO: Value Set OID (Code "mol+" oder "mol-"); fix: @codeSystem="2.16.840.1.113883.15.6" |
| 6 | rel | entryRelationship | 0..1 | optional | fix: @typeCode="SPRT" | |
| 7 | act | observation | 1..1 | required | Anzahl LymphknotenDiese Observation kann auch standalone bzw. in anderem Kontext verwendet werden. | |
| 8 | act | code | CD CWE | 1..1 | required | für die Angabe von Lymphknoten allgemein fix: @code="21894-1", @codeSystem="2.16.840.1.113883.6.1" für die Angabe von Sentinel Nodes fix: @code="SentinelNodes", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | RTO_PQ_PQ | 1..1 | required | |
| 9 | act | numerator | PQ | 1..1 | required | @value: Anzahl der befallenen Lymphknoten
fix: @unit="1" |
| 9 | act | denominator | PQ | 1..1 | required | @value: Anzahl der entfernten (untersuchten) Lymphknoten
fix: @unit="1" |
| 4 | rel | entryRelationship | 0..1 | optional | fix: @typeCode="SPRT" | |
M: Metastasen | ||||||
| 5 | act | observation | 1..1 | F | @classCode="OBS", @moodCode="EVN" | |
| 6 | act | code | CD CWE | 1..1 | required | fix: @code="tnmM", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 6 | act | text | ED | 1..1 | required | textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert |
| 6 | act | value | CD | 1..1 | required | Value Set: 1.2.276.0.76.11.3 |
| 7 | act | qualifier | CR | 0..1 | optional | Art der Sicherung (qualifier)Art der Sicherung (klinisch (Ultraschall, CT, MRT), pathologisch) |
| 8 | act | name | CV | 1..1 | required | fix: @code="tnmCp", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | CV | 1..1 | required | Value Set: 1.2.276.0.76.11.9 |
| 7 | act | qualifier | CR | 0..1 | optional | C-Faktor (Certainty) |
| 8 | act | name | CV | 1..1 | required | fix: @code="tnmCFactor", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | CV | 1..1 | required | TODO: Value Set OID; fix: @codeSystem="1.2.276.0.76.5.341" |
| 7 | act | qualifier | CR | 0..* | optional | Lokalisationscodes |
| 8 | act | name | CV | 1..1 | required | fix: @code="21920-4", @codeSystem="2.16.840.1.113883.6.1" |
| 8 | act | value | CV | 1..1 | required | TODO: Value Set OID; fix: @codeSystem="1.2.276.0.76.5.401" |
| 7 | act | qualifier | CR | 1..1 | required | i (Suffix): ITC durch Immunhistochemie (qualifier)fix: @classCode="OBS", @moodCode="EVN" |
| 8 | act | name | CD CWE | 1..1 | required | fix: @code="tnmI", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | CD | 1..1 | required | TODO: Value Set OID (Code "i+" oder "i-"); fix: @codeSystem="2.16.840.1.113883.15.6" |
| 7 | act | qualifier | CR | 1..1 | required | mol (Suffix): RT-PCR (ITC durch Molekularpathologie) (qualifier)fix: @classCode="OBS", @moodCode="EVN" |
| 8 | act | name | CD CWE | 1..1 | required | fix: @code="tnmMol", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 8 | act | value | CD | 1..1 | required | TODO: Value Set OID (Code "mol+" oder "mol-"); fix: @codeSystem="2.16.840.1.113883.15.6" |
| 4 | rel | entryRelationship | 0..1 | optional | fix: @typeCode="SPRT" | |
S: Serumtumormarker | ||||||
| 5 | act | observation | 1..1 | F | @classCode="OBS", @moodCode="EVN" | |
| 6 | act | code | CD CWE | 1..1 | required | fix: @code="tnmS", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 6 | act | text | ED | 1..1 | required | textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert |
| 6 | act | value | CD | 1..1 | required | TODO Value Set OID |
| 4 | rel | entryRelationship | 0..1 | optional | fix: @typeCode="SPRT" | |
L: Lymphgefäßinvasion | ||||||
| 5 | act | observation | 1..1 | F | @classCode="OBS", @moodCode="EVN" | |
| 6 | act | code | CD CWE | 1..1 | required | fix: @code="tnmL", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 6 | act | text | ED | 1..1 | required | textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert |
| 6 | act | value | CD | 1..1 | required | Value Set: 1.2.276.0.76.11.7 |
| 4 | rel | entryRelationship | 0..1 | optional | fix: @typeCode="SPRT" | |
V: Veneninvasion | ||||||
| 5 | act | observation | 1..1 | F | @classCode="OBS", @moodCode="EVN" | |
| 6 | act | code | CD CWE | 1..1 | required | fix: @code="tnmV", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 6 | act | text | ED | 1..1 | required | textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert |
| 6 | act | value | CD | 1..1 | required | Value Set: 1.2.276.0.76.11.6 |
| 4 | rel | entryRelationship | 0..1 | optional | fix: @typeCode="SPRT" | |
Pn: Perineuralinvasion | ||||||
| 5 | act | observation | 1..1 | F | @classCode="OBS", @moodCode="EVN" | |
| 6 | act | code | CD CWE | 1..1 | required | fix: @code="tnmPn", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| 6 | act | text | ED | 1..1 | required | textliche Beschreibung, i.d.R. vom sendenden System automatisch generiert |
| 6 | act | value | CD | 1..1 | required | Value Set: 1.2.276.0.76.11.8 |
| 4 | rel | entryRelationship | 0..1 | optional | fix: @typeCode="SPRT" | |
G: histopathologisches Grading | ||||||
| 5 | act | observation | 1..1 | required | siehe Template Grading | |
| 4 | rel | entryRelationship | 0..1 | optional | fix: @typeCode="SPRT" | |
R: Residualtumor | ||||||
| 5 | act | observation | 1..1 | required | siehe Template |
Beispiel
Das folgende Beispiel stellt das Maximum der möglichen Angaben dar, wohl wissend, dass dies aus medizinisch-fachlicher Sicht nicht korrekt ist.
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.276.0.76.3.1.131.1.10.3.5"/>
<id extension="tnm12345"
root="1.2.276.0.76.3.1.131.1.4.3.9999.9999.999909"/>
<code code="tnmStage"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
Tumorklassifikation vom 26.03.2011: ayr UICC IV
</text>
<effectiveTime value="20110326"/>
<value xsi:type="CD" code="IV"
codeSystem="2.16.840.1.113883.15.6" />
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="tnmYSymbol"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
y
</text>
<value xsi:type="BL" value="true" />
</observation>
</entryRelationship>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="tnmRSymbol"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
r
</text>
<value xsi:type="BL" value="true" />
</observation>
</entryRelationship>
<!-- T value -->
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="tnmT"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
pT3am(4)(m1)(sm2)C4
</text>
<value xsi:type="CD" code="T3a"
codeSystem="2.16.840.1.113883.15.6">
<qualifier>
<name code="tnmCp"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="p"
codeSystem="2.16.840.1.113883.15.6"/>
</qualifier>
<qualifier>
<name code="tnmCFactor"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="C4"
codeSystem="1.2.276.0.76.5.341"/>
</qualifier>
<qualifier>
<name code="tnmMSymbol"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="m"
codeSystem="2.16.840.1.113883.15.6"/>
</qualifier>
<qualifier>
<code code="tnmMultiplicity"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="INT.NONNEG" value="4" />
</qualifier>
<qualifier>
<name code="tnmMucosaSymbol"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="m1"
codeSystem="2.16.840.1.113883.15.6"/>
</qualifier>
<qualifier>
<name code="tnmSubmucosaSymbol"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="sm2"
codeSystem="2.16.840.1.113883.15.6"/>
</qualifier>
</value>
</observation>
</entryRelationship>
<!-- N value -->
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="tnmN"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
pN3(sn)(i+)(mol+)C5
</text>
<value xsi:type="CD" code="N3"
codeSystem="2.16.840.1.113883.15.6">
<qualifier>
<name code="tnmCp"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="p"
codeSystem="1.2.276.0.76.5.341"/>
</qualifier>
<qualifier>
<name code="tnmCFactor"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="C5"
codeSystem="1.2.276.0.76.5.341"/>
</qualifier>
<qualifier>
<name code="tnmSn"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="sn"
codeSystem="2.16.840.1.113883.15.6"/>
</qualifier>
<qualifier>
<code code="tnmI"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="BL" code="true"/>
</qualifier>
<qualifier>
<code code="tnmMol"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="BL" value="true"/>
</qualifier>
</value>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="21894-1" codeSystem="2.16.840.1.113883.6.1"/>
<value xsi:type="RTO_PQ_PQ">
<numerator value="2" unit="1"/>
<denominator value="12" unit="1"/>
</value>
</observation>
</entryRelationship>
</observation>
</entryRelationship>
<!-- M value -->
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="tnmM"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
cM1(sn)(i+)(mol+)C2
</text>
<value xsi:type="CD" code="M1"
codeSystem="2.16.840.1.113883.15.6">
<qualifier>
<name code="tnmCp"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="c"
codeSystem="1.2.276.0.76.5.341"/>
</qualifier>
<qualifier>
<name code="tnmCFactor"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="C2"
codeSystem="1.2.276.0.76.5.341"/>
</qualifier>
<qualifier>
<name code="tnmI"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="BL" value="true" />
</qualifier>
<qualifier>
<code code="tnmMol"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="BL" value="true" />
</qualifier>
</value>
</observation>
</entryRelationship>
<!-- further attributes -->
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="tnmS"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
S2
</text>
<value xsi:type="CD" code="S2"
codeSystem="2.16.840.1.113883.15.6"/>
</observation>
</entryRelationship>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="tnmL"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
L1
</text>
<value xsi:type="CD" code="L1"
codeSystem="2.16.840.1.113883.15.6"/>
</observation>
</entryRelationship>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="tnmV"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
V1
</text>
<value xsi:type="CD" code="V1"
codeSystem="2.16.840.1.113883.15.6"/>
</observation>
</entryRelationship>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="tnmPn"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
PnX
</text>
<value xsi:type="CD" code="PnX"
codeSystem="2.16.840.1.113883.15.6"/>
</observation>
</entryRelationship>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="336"
codeSystem="2.16.840.1.113883.3.7.1.0"/>
<value xsi:type="CD" code="2"
codeSystem="1.2.276.0.76.5.336"/>
</observation>
</entryRelationship>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.276.0.76.3.1.131.1.10.3.7"/>
<id extension="1234041"
root="1.2.276.0.76.3.1.131.1.4.3.9999.9999.999919"/>
<code code="tnmR" codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
gesamt R1, Fernmetastasen vorhanden
</text>
<effectiveTime value="20110326"/>
<value xsi:type="CD" code="R1"
codeSystem="2.16.840.1.113883.15.6">
<qualifier>
<name code="tnmRDomain"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="G" codeSystem="1.2.276.0.76.5.414"/>
</qualifier>
<qualifier>
<name code="tnmRLocation"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="L"
codeSystem="1.2.276.0.76.5.415"/>
</qualifier>
</value>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="residualIs"
codeSystem="1.2.276.0.76.3.1.131.1.5.1 "/>
<value xsi:type="BL" value="true"/>
</observation>
</entryRelationship>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="residualGt1mm"
codeSystem="1.2.276.0.76.3.1.131.1.5.1 "/>
<value xsi:type="BL" value="true"/>
</observation>
</entryRelationship>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="residualCy+"
codeSystem="1.2.276.0.76.3.1.131.1.5.1 "/>
<value xsi:type="BL" value="false"/>
</observation>
</entryRelationship>
</observation>
verwendete Terminologie
Stadiengruppierung nach UICC
| UICC | |||||
|---|---|---|---|---|---|
| Code | Beschreibung | Bedeutung | 5. | 6. | 7. |
| okk | TX, N0, M0 | X | X | X | |
| 0 | Carcinoma in situ | Tis, N0, M0 | X | X | X |
| 0a | X | X | X | ||
| 0is | X | X | X | ||
| I | X | X | |||
| IA | T1, N0, M0 | X | X | X | |
| IA1 | X | X | X | ||
| IA2 | X | X | X | ||
| IB | T2, N0, M0 | X | X | X | |
| IB1 | X | X | X | ||
| IB2 | X | X | X | ||
| IC | X | X | X | ||
| II | X | X | X | ||
| IIA | T1, N1, M0 | X | X | X | |
| IIA1 | X | ||||
| IIA2 | X | ||||
| IIB | T2, N1, M0 | X | X | X | |
| IIC | T3, N0, M0 | X | X | X | |
| III | X | X | X | ||
| IIIA | T1, N2, M0 | X | X | X | |
| T2, N2, M0 | |||||
| T3, N1,2, M0 | |||||
| IIIB | T4, jedes N, M0 | X | X | X | |
| jedes T, N3, M0 | |||||
| IIIC | X | X | X | ||
| IS | X | X | X | ||
| IV | jedes T, jedes N, M1 | X | X | X | |
| IVA | X | X | X | ||
| IVB | X | X | X | ||
| IVC | X | X | X |
Tabelle: Codes für Stadiengruppierung nach UICC16
Value Set (OID 1.2.276.0.76.11.5)
UICC 5. Auflage (OID 2.16.840.1.113883.15.8)
UICC 6. Auflage (OID 2.16.840.1.113883.15.7)
UICC 7. Auflage (OID 2.16.840.1.113883.15.6)
Ausdehnung des Primärtumors (T)
Bekannte T-Kategorien (ohne Darstellung möglicher Zusätze). Die Beschreibung variiert je nach Entität. Der Code soll nur in Zusammenhang mit der Art der Tumorerkrankung übermittelt werden, da die Kodes mit unterschiedlichen Bedeutungen belegt sind (z.B. T1 entspricht 2-3 cm oder 2-4 cm).
| UICC | ||||||
|---|---|---|---|---|---|---|
| Code | Description | Bedeutung | 5. | 6. | 7. | SCT |
| Ta | Noninvasive papillary urothelial carcinoma | Nicht invasiv | X | X | X | X |
| Tis | Carcinoma in situ | Nicht invasiv | X | X | X | X |
| Tis(pu) | in situ urothelial carcinoma of prostatic urethra | Nicht invasiv | X | X | ||
| Tis(pd) | in situ urothelial carcinoma of prostatic gland ducts | Nicht invasiv | X | X | ||
| T0 | No evidence of primary tumour | Kein Primärtumor nach¬weisbar | X | X | X | |
| T1 | X | X | X | X | ||
| T1mic | Microinvasion | X | X | X | ||
| T1mi | Microinvasion | X | ||||
| T1a | X | X | X | X | ||
| T1a1 | X | X | X | X | ||
| T1a2 | X | X | X | X | ||
| T1b | X | X | X | X | ||
| T1b1 | X | X | X | X | ||
| T1b2 | X | X | X | X | ||
| T1c | X | X | X | X | ||
| T1d | X | X | ||||
| T2 | X | X | X | X | ||
| T2a | X | X | X | X | ||
| T2a1 | X | X | ||||
| T2a2 | X | X | ||||
| T2b | X | X | X | X | ||
| T2c | X | X | X | X | ||
| T2d | X | X | ||||
| T3 | X | X | X | X | ||
| T3a | X | X | X | X | ||
| T3b | X | X | X | X | ||
| T3c | X | X | X | X | ||
| T3d | X | X | X | |||
| T4 | X | X | X | X | ||
| T4a | X | X | X | X | ||
| T4b | X | X | X | X | ||
| T4c | X | X | X | X | ||
| T4d | X | X | X | X | ||
| T4e | X | X | ||||
| TX | Primary tumour cannot be assessed | Stadium des Primärtumors kann nicht nachgewiesen werden. |
Tabelle: Codes für Ausdehnung des Primärtumors (T)
Value Set (OID 1.2.276.0.76.11.1)
UICC 5. Auflage (OID 2.16.840.1.113883.15.8)
UICC 6. Auflage (OID 2.16.840.1.113883.15.7)
UICC 7. Auflage (OID 2.16.840.1.113883.15.6)
Nodalstatus (regionäre Lymphknoten) (N)
| UICC | |||||||
|---|---|---|---|---|---|---|---|
| Code | Description | Bedeutung | Entität | 5. | 6. | 7. | SCT |
| N0 | No regional lymph node metastasis | Kein Lymphknotenbefall | alle | X | X | X | X |
| N1 | X | X | X | X | |||
| N1mi | lymph node micrometastasis | alle | X | X?? | |||
| N1mi | Bilateral regional lymph node metastasis | Vulva | X | X ???? | |||
| N1a | alle | X | X | X | X | ||
| N1b | X | X | X | X | |||
| N1b1 | X | ??? | X | ||||
| N1b2 | X | ??? | X | ||||
| N1b3 | X | ??? | X | ||||
| N1b4 | X | ??? | X | ||||
| N1c | X | X ??? | |||||
| N2 | X | X | X | X | |||
| N2a | X | X | X | X | |||
| N2b | X | X | X | X | |||
| N2c | X | X | X | X | |||
| N3 | X | X | X | X | |||
| N3a | X | X | X | ||||
| N3b | X | X | X | ||||
| N3c | X | X | |||||
| NX | Regional lymph nodes cannot be assessed | Lymphknotenbefall unbekannt | X | X | X |
Tabelle: Codes für Nodalstatus (N)
Value Set (OID 1.2.276.0.76.11.2)
UICC 5. Auflage (OID 2.16.840.1.113883.15.8)
UICC 6. Auflage OID 2.16.840.1.113883.15.7)
UICC 7. Auflage (OID 2.16.840.1.113883.15.6)
Fernmetastasen (M)
Der Code zur Beurteilung von Fernmetastasen soll nur in Zusammenhang mit der Art der Tumorerkrankung übermittelt werden
| UICC | ||||||
|---|---|---|---|---|---|---|
| Code | Description | Bedeutung | Entität | 5. | 6. | 7. |
| M0 | No distant metastasis | Fernmetastasen nicht vorhanden (für p nicht anwendbar) | alle | X | X | X |
| M1 | Distant metastasis | Fernmetastasen vorhanden | alle | X | X | X |
| M1a | nur Ösophagus und Prostata | X | X | X | ||
| M1b | nur Ösophagus und Prostata | X | X | X | ||
| M1c | X | X | X | |||
| M1d | X | |||||
| M1e | X | |||||
| MX | Distant metastasis cannot be assessed (für p nicht anwendbar) | alle | X | X | X |
Tabelle 22: Codes für Fernmetastasen (M)
Value Set (OID 1.2.276.0.76.11.3)
UICC 5. Auflage (OID 2.16.840.1.113883.15.8)
UICC 6. Auflage (OID 2.16.840.1.113883.15.7)
UICC 7. Auflage (OID 2.16.840.1.113883.15.6)
Veneninvasion
| Code | Description | Bedeutung |
|---|---|---|
| V0 | no venous invasion | keine Veneninvasion |
| V1 | microscopic venous invasion | mikroskopische Veneninvasion |
| V2 | macroscopic venous invasion | makroskopische Veneninvasion |
| VX | venous invasion cannot be assessed | Veneninvasion nicht feststellbar |
Tabelle: Codes für Veneninvasion
Value Set (OID 1.2.276.0.76.11.6)
UICC 5. Auflage (OID 2.16.840.1.113883.15.8)
UICC 6. Auflage (OID 2.16.840.1.113883.15.7)
UICC 7. Auflage (OID 2.16.840.1.113883.15.6)
Lymphgefäßinvasion
| Code | Description | Bedeutung |
|---|---|---|
| L0 | no lymphatic invasion | keine Lymphsysteminvasion |
| L1 | lymphatic invasion | Lymphsysteminvasion |
| LX | lmphatic invasion cannot be assessed | Lymphsysteminvasion nicht fest¬stellbar |
Tabelle: Codes für Lymphgefäßinvasion
Value Set (OID 1.2.276.0.76.11.7)
UICC 5. Auflage (OID 2.16.840.1.113883.15.8)
UICC 6. Auflage (OID 2.16.840.1.113883.15.7)
UICC 7. Auflage (OID 2.16.840.1.113883.15.6)
Perineuralscheideninvasion
| Code | Description | Bedeutung |
|---|---|---|
| Pn0 | no perineural invasion | keine perineurale Invasion |
| Pn1 | Perineural invasion | perineurale Invasion |
| PnX | unknown | Unbekannt |
Tabelle: Codes für Perineuralinvasion
Value Set (OID 1.2.276.0.76.11.8)
UICC 5. Auflage (OID 2.16.840.1.113883.15.8)
UICC 6. Auflage (OID 2.16.840.1.113883.15.7)
UICC 7. Auflage (OID 2.16.840.1.113883.15.6)
Unterscheidung autoptisch/klinisch/pathologisch
| Code | Description | Bedeutung |
|---|---|---|
| a | autoptisch | |
| c | Assessment according to clinical criteria | Beurteilung nach klinischen Kriterien |
| u | Assessment according to clinical criteria based on ultraound evidence | Beurteilung nach klinischen Kriterien / Ultraschall |
| ct | Assessment according to clinical criteria based on CT evidence | Beurteilung nach klinischen Kriterien /CT |
| mr | Assessment according to clinical criteria based on magnetic resonance evidence | Beurteilung nach klinischen Kriterien / MRT |
| p | Assessment according to the pathological results | nach dem pathologischen Befund |
Tabelle: Codes für Unterscheidung klinisch/pathologisch
Value Set (OID 1.2.276.0.76.11.9)
UICC 5. Auflage (OID 2.16.840.1.113883.15.8)
UICC 6. Auflage (OID 2.16.840.1.113883.15.7)
UICC 7. Auflage (OID 2.16.840.1.113883.15.6)
Beschränkung auf Mukosa
Gilt nur für gastrointestinale Karzinome.
| Code | Description | Bedeutung |
|---|---|---|
| m1 | restricted to mucosa | auf Mukosa beschränkt, 1. Drittel |
| m2 | restricted to mucosa | auf Mukosa beschränkt, 2. Drittel |
| m3 | restricted to mucosa | auf Mukosa beschränkt, 3. Drittel |
Tabelle: Codes für Beschränkung auf Mukosa
Value Set (OID 1.2.276.0.76.11.TODO)
UICC 5. Auflage (OID 2.16.840.1.113883.15.8)
UICC 6. Auflage (OID 2.16.840.1.113883.15.7)
UICC 7. Auflage (OID 2.16.840.1.113883.15.6)
Beschränkung auf Submukosa
Gilt nur für gastrointestinale Karzinome.
| Code | Description | Bedeutung |
|---|---|---|
| sm1 | restricted to submucosa | auf Submukosa beschränkt, 1. Drittel |
| sm2 | restricted to submucosa | auf Submukosa beschränkt, 2. Drittel |
| sm3 | restricted to submucosa | auf Submukosa beschränkt, 3. Drittel |
Tabelle: Codes für Beschränkung auf Submukosa
Value Set (OID 1.2.276.0.76.11.TODO)
UICC 5. Auflage (OID 2.16.840.1.113883.15.8)
UICC 6. Auflage (OID 2.16.840.1.113883.15.7)
UICC 7. Auflage (OID 2.16.840.1.113883.15.6)
C-Faktor (Certainty, Diagnosesicherheit)
| Code | Description | Nachweis |
|---|---|---|
| C1 | Evidence from standard diagnostic means (e.g., inspection, palpation, and standard radiography, intra¬luminal endoscopy for tumours of certain organs) | Nachweis durch diagnostische Standardmethoden (Inspektion, Palpation, einfache Röntgenaufnahmen) |
| C2 | Evidence obtained by special diag¬nostic means (e.g., radiographic imaging in special projections, tomography, computerized tomo¬graphy \[CT\], ultrasonography, lympho¬graphy, angiography; scinti¬graphy; magnetic resonance imaging \[MRI\]; endoscopy, biopsy, and cytology) | Nachweis durch spezielle klinische diagnostische Methoden einschließlich Computertomogramm, Magnet-Resonanz-Tomographie |
| C3 | Evidence from surgical exploration, including biopsy and cytology | Nachweis durch Operation, einschließlich Biopsie und Zytologie |
| C4 | Evidence of the extent of disease following definitive surgery and pathological examination of the resected specimen | Nachweis durch operative Behandlung mit pathologischer Untersuchung der entnommenen Gewebeteile |
| C5 | Evidence from autopsy | Nachweis durch Autopsie |
Tabelle: Codes für C-Faktor (OID 1.2.276.0.76.5.341)
Lokalisation von Fernmetastasen
| Code | Description | Bedeutung | |
|---|---|---|---|
| PUL | Pulmonary | Pulmonal | Lungenmetastase |
| OSS | Osseous | Ossär | Knochenmetastase |
| HEP | Hepatic | Hepatisch | Lebermetastase |
| BRA | Brain | Cerebral | Hirnmetastase |
| LYM | Lymph Nodes | Lymphonodulär | Lymphknotenmetastase |
| OTH | Others | Andere | Andere Metastase |
| MAR | Bone Marrow | Medullär | Knochenmarkmetastase |
| PLE | Pleura | Pleural | RippenfellMetastase |
| ADR | Adrenals | Adrenal | Nebennierenmetastase |
| SKI | Skin | dermal | Hautmetastase |
Tabelle: Codes für Lokalisation von Fernmetastasen (OID 1.2.276.0.76.5.401)