Grading-Entry (Template)
K (→Spezifikation) |
(→Modell) |
||
| Zeile 35: | Zeile 35: | ||
[[Datei:Cdaab2_Grading_Modell_130726.GIF]] | [[Datei:Cdaab2_Grading_Modell_130726.GIF]] | ||
| + | |||
| + | Wenn keine speziellen Scoresysteme zum Grading verwendet werden, muss über einen Qualifier die Art des Gradings (zweistufig, dreistufig, vierstufig) angegeben werden. | ||
| + | |||
| + | Gradingangaben aus speziellen Score-Systemen werden ebenfalls in Observation-Klassen eingebunden, die über eine ActRelationShip-Klasse mit dem code SPRT mit der zentralen Observation-Klasse verbunden werden. | ||
=== Spezifikation=== | === Spezifikation=== | ||
Version vom 28. Juli 2013, 14:50 Uhr
Dieses Material ist Teil des Leitfadens Arztbrief 2.x.
|
Inhaltsverzeichnis
Entry: Grading
| Template-Metadaten | |
| Template-Typ | Entry |
| Template ID | 1.2.276.0.76.5.336 |
| genutztes Template | ICD-O-Diagnose, TNM Klassifikation |
| abgeleitete Templates | |
| Generelle Beschreibung | Malignitätsgrad / Differenzierungsgrad eines Tumors |
| allg. Erläuterung | ICD-O-Diagnose |
| Ballotierungsstatus | in Arbeit |
| Erweiterbarkeit | geschlossen |
Definition und Zweck
Das Grading ist eine Methode der Histopathologie, den Differenzierungsgrad / Malignitätsgrad eines malignen Tumors semiquantitativ zu beschreiben bzw. zu klassifizieren. Die Rationale des Grading ist die Tatsache, dass mit zunehmender "Unähnlichkeit" des Tumorgewebes zum Ausgangsgewebe, bezeichnet als Entdifferenzierung, die Aggressivität der meisten Tumoren zunimmt und die Prognose sich verschlechtert. Große Abweichungen von der Histoarchtektur, starke Pleomorphie der Tumorzellen und Zeichen einer hohen Proliferationsaktivität entsprechen einem schlecht differenzierten Tumor mit einem hohen Malignitätsgrad. Tumoren, die nur schwach proliferieren, geringgradige Strukturanomalien aufweisen und Zellkerne haben, die sich nur geringfügig vom Muttergewebe unterscheiden, sind gut differenziert und haben einen niedrigen Malignitätsgrad. Bei Anwendung dieser Regeln können meist jeweils 10-25% einer Tumorentität als gut, bzw. schlecht differenzierte Tumoren bezeichnet werden, die Mehrzahl hat jedoch morphologische Charakteristika, die diese eindeutige Zuordnung nicht zulassen und dann als mäßig differenziert bezeichnet werden müssen. Die recht subjektive Einschätzung des Differenzierungsgrades kann durch die Zuhilfenahme von teilweise sehr komplizierten Scoring-Systemen (s.u.) sowie durch die Messung von morphologischen oder molekularen Eigenschaften objektiviert werden, so dass der Anteil der "intermediären" Differenzierungsgrade verkleinert wird. Für eine zunehmende Reihe von Tumorentitäten wird heutzutage eine Unterteilung in nur zwei Malignitätsgrade vorgenommen: low grade (geringe Malignität, gut differenziert) und high grade (hohe Malignität, schlecht differenziert), wobei der ursprüngliche intermediäre Differenzierungsgrad je nach Entität dem low oder high grade zugeschlagen wird.
Das Entry Template "Grading" kann in allen diagnoseassoziierten Sections und Entries eingebunden werden. Es ist analog zum Qualifier "Grading" in den Temples für ICD-O und für TNM zu verwenden. In dieses Entry können weiter Entries zu speziellen Gradingscore-Systemen (siehe Assessment Scales) verschachtelt werden, falls diese zusätzliche Informationen zum Differenzierungsgrad liefern oder Einzelwerte von Scores mitgeteilt werden sollen.
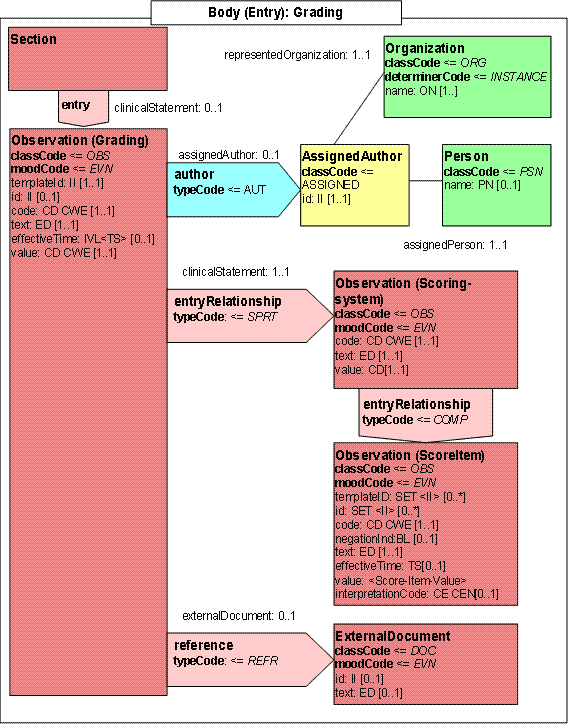
Modell
Wenn keine speziellen Scoresysteme zum Grading verwendet werden, muss über einen Qualifier die Art des Gradings (zweistufig, dreistufig, vierstufig) angegeben werden.
Gradingangaben aus speziellen Score-Systemen werden ebenfalls in Observation-Klassen eingebunden, die über eine ActRelationShip-Klasse mit dem code SPRT mit der zentralen Observation-Klasse verbunden werden.
Spezifikation
Beispiel
muss noch angepasst werden!! GH
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.276.0.76.3.1.131.1.10.3.9"/>
<id extension="1234009"
root="1.2.276.0.76.3.1.131.1.4.3.9999.9999.999919"/>
<code code="33732-9"
codeSystem="2.16.840.1.113883.6.1"/>
<text>
Grad 2
</text>
<effectiveTime value="20110326"/>
<value xsi:type="CD" code="L" codeSystem="1.2.276.0.76.5.336">
</value>
<!-- Einzelwerte zum Score: 1. Wert -->
<entryRelationship typeCode='COMP' inversionInd='false'>
<observation moodCode='EVN' classCode='OBS'>
<code code='44641-9' displayName=''
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC' />
<statusCode code='completed'/>
<value xsi:type='CD' code='4' codeSystem='1.2.276.0.76.5.402'/>
</value>
</observation>
</entryRelationship>
<!-- Einzelwerte zum Score: 2. Wert -->
<entryRelationship typeCode='COMP' inversionInd='false'>
<observation moodCode='EVN' classCode='OBS'>
<code code='44642-7' displayName=''
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC' />
<statusCode code='completed'/>
<value xsi:type='CD' code='5' codeSystem='1.2.276.0.76.5.403'/>
</value>
</observation>
</entryRelationship>
</observation>
Value set
Value Set (OID TODO - Code System 1.2.276.0.76.5.336):
| Code | Beschreibung |
|---|---|
| 1 | Gut differenziert (G1) |
| 2 | Mäßig differenziert (G2) |
| 3 | Schlecht differenziert (G3) |
| 4 | Undifferenziert (G4) |
| L | Niedriggradig maligne (G1-2) |
| M | Mittelgradig maligne (G2-3) |
| H | Hochgradig maligne (G3-4) |
| B | Grenzfall (Borderline) |
| X | Differenzierungsgrad nicht bestimmbar |