Body
Dieses Material ist Teil des Leitfadens Pathologiebefund.
|
Inhaltsverzeichnis
CDA Body
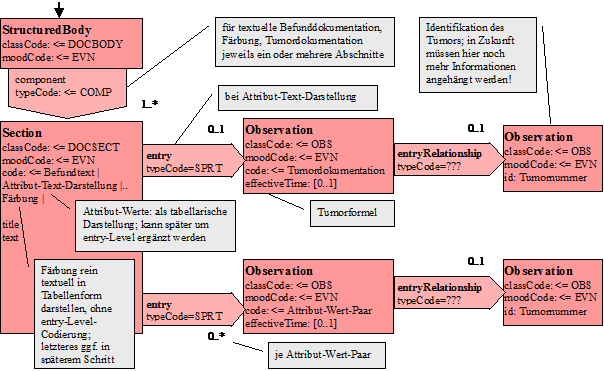
Die eigentliche klinische Dokumentation wird im so genannten CDA Body festgehalten. Im Vordergrund steht hier „lesbarer" (narrativer) Text, der verpflichtender Bestandteil von CDA R2 Dokumenten ist und die Interoperabilität zwischen den menschlichen Kommunikationspartnern garantiert. Hier sind Möglichkeiten gegeben, diesen Text grob zu strukturieren, wie man dies von den Möglichkeiten der Textverarbeitung her kennt. Zur Strukturierung stellt die Standardspezifikation eine Reihe von XML-Elementen zur Verfügung, die als Body Structures zusammengefasst werden können. Der Body enthält ein oder mehrere Abschnitte (sections). Diese können auch ineinander geschachtelt sein, so wie Kapitel und Unterkapitel in einem Buch. Zudem sind Strukturierungen im textuellen Bereich im Sinne von Tabellen oder Listen möglich.
- Abschnitte <section>
- Paragrafen <paragraph>
- Kennzeichnung von bestimmten Inhalten <content>
- Überschriften <caption>
- Tabellen <table>
- Listen <list>
Sections enthalten immer einen narrativen Block und erfüllen damit eine der oben genannten Maximen von CDA: die Mensch-zu-Mensch-Interoperabilität, die Lesbarkeit der Informationen für den Menschen. Im narrativen Block, durch das Textattribut in der section-Klasse repräsentiert, wird eingebetteter Text innerhalb eines Abschnittes angegeben. Dabei kann mit oben genanntem <content> Element bestimmter Inhalt gesondert gekennzeichnet werden.
Zusammengefasst werden im Textblock (teils so auch schon in CDA Release 1 realisiert) u.a. folgende Möglichkeiten der Struktur- und Formgebung des fließenden Textes gegeben:
- Zeilenumbrüche <br>
- Stilistische Angaben (unterstreichen, fett, kursiv etc.)
- Hoch- und Tiefstellung von Text
- Fußnoten
- Symbole
- Revisionsmarken im Text wie <delete>, <insert>
Mit den beschriebenen Body Strukturen können CDA Entries verbunden sein. Diese repräsentieren den „computerlesbaren Teil" innerhalb eines Dokumentenabschnitts. Body Entries sind im Prinzip eine Auswahl aus Klassen mitsamt Attributen aus dem HL7 Referenz-Informationsmodell (RIM).
Dieses Material ist Teil des Leitfadens Pathologiebefund.
|
Modell
Nachfolgend ist das CDA-Modell angegeben, so wie es für den Pathologie-Bericht instanziiert wird:
Abbildung 5: Level-3-Modell
Dieses Material ist Teil des Leitfadens Pathologiebefund.
|
Beispiele für Befunde
Zu Beginn ein relativ einfaches und kurzes Beispiel:
Material:
Zystenbalg Regio 38
Makroskopische Beurteilung:
Zusammen pampelmusenkerngroße (Durchmesser 12 mm) membranöse festelastische grauweißliche Gewebsstücke.
Mikroskopische Beurteilung:
Partiell durch ein sehr schmales, nicht verhorntes Plattenepithel ausgekleideter Balganteil einer odontogenen Zyste mit einzelnen Malassezschen Epithelnestern und herdbetonter sehr schütterer rundzelliger entzündlicher subepithelialer Infiltration. Eingesprengt wenig Hartmaterial.
Diagnose:
Follikuläre Zyste. Kein Anhalt für Malignität oder Spezifität am vorliegenden Material.
Unterschrift
Material:
Sonographisch gestützte Stanzbiopsie Mamma re. 10 Uhr
Makroskopische Beurteilung:
Mehrere (gemäß klinischer Angabe fünf) zusammen 51 mm lange weiche bis mittelfeste teils grauweißliche, teils graugelbliche Punktionszylinder von max. Bleistiftminenstärke.
Mikroskopische Beurteilung: (HE, CK 5/14, CK 7):
Alle gewonnenen Punktionszylinder wurden vollständig gebettet und mit 13 Schnittstufen untersucht. Sie bestehen aus lipomatös und fibrolipomatös transformiertem Brustdrüsengewebe und lassen in mindestens sechs Punktionszylinderteilstücken neben atrophischen Drüsenlobuli und ektatischen Milchgangsanschnitten sowie kleineren Adenoseherden (mikrozystische und blunt-duct-Adenosen) in wechselnder Dichte in ein hyalinelastotisch transformiertes Stroma eingebettete unterschiedlich großkalibrige Tubuli erkennen, diese vielfach mit Abknickungen, die ausgekleidet werden von einem einreihigen, überwiegend gering, örtlich mäßig atypischen flach kubischen Epithel ohne nennenswerte mitotische Aktivität und fehlende basale Myoepithellage in der CK 5/14-Reaktion, vereinbar mit Tumorinfiltraten eines tubulären Mammakarzinoms (Malignitätsgrad 1 nach Elston und Ellis; 1+2+1); vgl. auch EH-Nr. 7082/12.
Diagnose: Tubuläres Karzinom der Mamma.
Klassifikation nach NHSBSP: B 5b
Zum Ergebnis der noch ausstehenden Hormonrezeptorbestimmung und des HER-2/neu- Status erfolgt ein Nachbericht. Telefonische Vorabinformation am 31.05.2012.
Unterschrift
1. Nachbericht:
Ergebnis der immunhistochemischen Hormonrezeptorbestimmung:
Östrogenrezeptoren: über 80 % positiver Zellkerne, vorherrschende Färbeintensität: stark, IRS 12 nach Remmele und Stegner.
Progesteronrezeptoren: über 80 % positiver Zellkerne, vorherrschende Färbeintensität: stark, IRS 12 nach Remmele und Stegner.
Der Tumor ist endokrin-responsiv.
Ergebnis der in-situ-Hybridisierung (BDISH) des HER2-neu-Gens:
Nach einer in-situ-Hybridisierung des Genlocus c-erb-B2 und des Zentromer von Chromosom 17 wurden die ISH-Signale von 30 Tumorzellen ausgezählt. Die untersuchten invasiven Tumorzellen zeigen ein mittleres Verhältnis 1,19 (Gen/Zentromer). Es liegt also keine Amplifikation des HER2-neu Lokus vor (ZytoDot 2C SPEC Her- 2/CEN17 Probe Kit).
Unterschrift
Material:
1. Sentinel-LK re. 2. Segmentresektat Mamma re.
Makroskopische Beurteilung:
1. Backpflaumengroßes (40 x 20 x 15 mm messendes) Fettgewebsstück, darin
eingeschlossen drei, max. gewürzkorngroße (größter Durchmesser 6 mm) mittelfeste
grauweißliche Gewebsknoten.
2. Auf Styroporplatte nadelfixiertes, zweifach fadenmarkiertes fettgewebsreiches
Mammaresektat aus dem zentralen Drüsenkörper von 119 g in einer Ausdehnung
von 6,5 x 7 x 4 cm mit zentral aufsitzender, 4 cm durchmessender Mamille/Areole.
Mamille und unmittelbar retromamilläres Drüsenkörpergewebe in einer Ausdehnung
von 12 mm derb knotig grauweiß induriert. Auf der Mamille selbst aufgelagert
eine kleine Kruste.
Das Resektat wird von lateral beginnend in neun Scheiben lamelliert. Weitere
verdächtige Knotenbildungen kommen nicht zur Darstellung.
Sicherheitszonen der beschriebenen Knotenbildungen zu allen Resektionsrändern
mindestens 10 mm bzw. deutlich darüber.
Schnellschnittdiagnose:
1. Alle drei Sentinellymphknoten im Schnellschnitt metastasenfrei.
2. Karzinom unmittelbar retromamillär, Sicherheitszonen nach allen Seiten
mindestens 10 mm bzw. deutlich darüber.
Telefonische Befundübermittlung am 15.06.2012, 11.59 Uhr an Herrn CA Dr. XY.
Mikroskopische Beurteilung:
1. Alle drei Sentinellymphknoten, die vollständig gebettet und in Stufenserienschnitten
mit jeweils 12 Schnittstufen aufgearbeitet wurden, sind metastasenfrei.
Man erkennt kleinherdige lipomatöse Transformationen und narbige Fibrosierungen.
Umgebendes Fettgewebe unauffällig.
2. Das Mammaresektat wurde unter Berücksichtigung der Topografie und Markierungen mit 13 Paraffinblöcken mit jeweils Schnittstufen untersucht. In der Haut der Mamillenregion bzw. unmittelbar retromamillär unter Einbeziehung der lokalisationstypisch größeren Ductus und Sinus lacteripheri Tumorgewebe des in der Stanzbiopsie vordiagnostizierten invasiven Mammakarzinoms (vgl. EH-Nr. 7226/12), jetzt als gemischt invasiv duktales und lobuläres Mammakarzinom zu klassifizieren (Malignitätsgrad 2 nach Elston und Ellis; 3+2+1) mit vordergründig dissolut einzelzelligem Wachstumsmuster mit Ausbildung sog. indian files und Targetstrukturen um unbeeinträchtigte Milchgänge, aber auch tubulären Differenzierungen und kleiner intraduktaler Komponente, die an umschriebener Stelle den invasiven Karzinomanteil nach dorsal um 1 mm überschreitet. Es besteht eine wechselnd stark ausgeprägte Stromasklerose mit örtlich schütter rundzellig entzündlicher Stromareaktion und man sieht in einem der Schnittpräparate eine umschriebene, herdförmig eingeblutete narbige Fibrose mit reaktiven Veränderungen der bedeckenden Epidermis bei Zustand nach vorausgegangener Punchbiopsie. Karzinomentfernung im Gesunden, Sicherheitszonen nach allen Seiten jeweils deutlich über 10 mm. Breit anhängender Saum tumorfreien Brustdrüsengewebes betont lipomatös transformiert ohne sonstigen nennenswerten pathologischen Befund.
Zusammenfassung:
Ungewöhnlich oberflächlich retromamillär bzw. in der Haut der Mamillenregion rechts gelegenes gemischt invasiv duktales und lobuläres Mammakarzinom. Karzinomentfernung im Gesunden (Sicherheitszonen nach allen Seiten über 10 mm). Drei metastasenfreie Sentinellymphknoten.
Unterbeauftragte Untersuchung:
Unfixiertes Tumorgewebe wurde zur uPA/PAI-1-Bestimmung an das Institut für Pathologie des UKD gesandt. Über das Ergebnis wird nachberichtet.
Tumorklassifikation:
TNM (UICC, 7. Auflage):
pT1c pN0 (0/3sn), R0, L0, V0
Grading:
G 2
ICD-O-3:
C 50.0, M 8522/3
Unterschrift
1. Nachbericht:
Ergebnis der uPA/PAI-1-Analyse:
Methodik: Am unfixierten Gewebe des invasiven Mammakarzinoms wurde nach Kryokonservierung mit dem FEMTELLE® ELISA-Test die Aktivität des Plasminogenaktivators vom Urokinasetyp (uPA) und des Plasminogen-Inhibitors (PAI-1) bestimmt (Institut für Pathologie des UKD, Prof. Dr. XYZ).
Untersuchungsergebnis:
uPA: 6,27 ng/mg Protein
PAI-1: 12,4 ng/mg Protein
Bewertung:
Bei diesem Ergebnis kann bei nodal-negativen Mammakarzinom, Malignitätsgrad 2 nach Elston und Ellis entsprechend der Interdisziplinären S3-Leitlinie für die Diagnostik, Therapie und Nachsorge des Mammakarzinoms von einem hohen Rezidivrisiko ausgegangen werden.
Unterschrift
PathoBerichtText
Wiederholung der Ki-67-Färbung von der 2. Fraktion (rechts).
Beurteilung
1. Isolierte Tumorzellen in einem funktionsgesteigerten Lymphknoten (linke Axilla Sentinellymphknoten 544cps).
2. Teils glanduläres, teils solides, invasives ductales Adenocarcinom der Brustdrüse, geringe nukleäre Atypien sowie ductales Carcinoma in situ mit geringen Atypien, DCIS I und Mikrokalk bis 0,2mm (rechte Mamma oben zwischen den Quadranten, Resektat); immunhistologisch fokal starke Expression des Östrogenrezeptors in etwa 60% der Tumorzellen.
Score nach Elston und Ellis: 4.
Immunreaktiver Score: Östrogenrezeptor 9, Progesteronrezeptor 0.
Onkoprotein C-erbB-2 Index: 0
Größter Durchmesser des invasiven Carcinoms etwa 9mm, das invasive Carcinom reicht zumindest an die craniale Abtragungsebene im peripheren (cranialen) Pol. Durchmesser der in situ Komponente etwa 35mm, Abstand von der nächstgelegenen (posterioren) Abtragungsebene etwa 0,4mm, die in situ Komponente breitet sich allerdings ebenfalls in den peripheren (cranialen) Pol aus und erreicht damit dort mindestens die Abtragungsebene.
3. Weitgehend solides, invasives ductales Adenocarcinom der Brustdrüse, mittelgradige, herdförmig schwere nukleäre Atypien, etwa in gleicher Größe ductales Carcinoma in situ mit schweren Atypien, DCIS III sowie Nekrosen und Verkalkungen bis 4mm (Resektat linke Mamma oben außen); immunhistologisch starke Expression des Östrogenrezeptors und des Progesteronrezeptors jeweils in etwa 85% der Tumorzellen. Immunreaktiver Score: Östrogenrezeptor 12, Progesteronrezeptor 12. Kleine Wachstumsfraktion (Ki-67 um 10 %). Onkoprotein C-erbB-2 (Her-2-Neu-Index: 0). Größter Durchmesser der invasiven Komponente und der in-situ-Komponente jeweils etwa 23mm. Abstand der invasiven Komponente von der nächstgelegenen Abtragungsebene (posterior) 3,2mm, Abstand von anterior 12mm, von cranial 20mm, von caudal 23mm, von medial 30mm, von lateral 13mm. Die in-situ-Komponente breitet sich zumindest bis an die posteriore Abtragungsebene aus.
4. Neun tumorfreie Lymphknoten (linke Axilla).
5. Tumorfreies Fettgewebe (linke Mamma, Nachresektat lateral).
6. Überwiegend tumorfreies Fettgewebe mit kleinen Anteilen von tumorfreiem Brustdrüsengewebe (linke Mamma, Nachresektat mamillenwärts).
7. Überwiegend tumorfreies Fettgewebe mit kleinen Anteilen von tumorfreiem Brustdrüsengewebe (linke Mamma, Nachresektat cranial).
Auch nach Wiederholung lässt sich die Wachstumsfraktion in dem kleinen Tumor auf der rechten Seite nicht darstellen, vermutlich wurde das Antigen durch die schwere thermische Schädigung zerstört.
Pathologiebefundbericht
| Template ID | 1.3.6.1.4.1.19376.1.8.1.1.1 (AP APSR 6.2.3.1) | |
| General Description | In diesem Template werden die Daten zum Pathologiebefundbericht übermittelt. | |
| Status | identisch zu IHE | |
| LOINC Code | Opt. | Description |
| 11526-1 | R | Pathology Study |
Definition und Zweck
Dieses Template definiert die Grundlage für die Einschränkungen jedes strukturierten Pathologiebefundberichts unabhängig von Organ oder Diagnose. Es ist damit das generische template für jeden strukturierten Pathologiebefund.
Beispiel
<ClinicalDocument xmlns='urn:hl7-org:v3'>
<typeId extension="POCD_HD000040" root="2.16.840.1.113883.1.3"/>
<templateId root='1.3.6.1.4.1.19376.1.8.1.1.1'/>
<id root='1.3.6.1.4.1.19376.1.8.9' extension='123'/>
<code code='11526-1' displayName='Pathology study'
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title>Anatomic pathology structured report</title>
<effectiveTime value='20100114153000-0700'/>
<confidentialityCode code='N' displayName='Normal' codeSystem='2.16.840.1.113883.5.25'/>
<languageCode code='en-US'/>
<!-- one patient -->
<recordTarget><patientRole> .. </patientRole></recordTarget>
<!-- one or more author -->
<author> .. </author>
<!-- one or more transcriptionists -->
<dataEnterer> .. </dataEnterer>
<!-- one or more person who provided useful information as input to this document -->
<informant> .. </informant>
<!-- the organization (laboratory) issuing this report and in charge with its lifecycle -->
<custodian> .. </custodian>
<!-- zero or more intended recipient other than the ordering physician (« copy to ») -->
<informationRecipient> .. </informationRecipient>
<!-- the person (lab director) legally responsible for this report, who may have signed it -->
<legalAuthenticator> .. </legalAuthenticator>
<!-- one or more pathologists who validated and/or corrected the content -->
<authenticator> .. </authenticator>
<!-- the ordering physician, and the date-time the order was issued -->
<participant typeCode='REF'> .. </participant>
<!-- zero or more distinct specimen collector, and the date-time the specimen was collected -->
<participant typeCode='DIST'> .. </participant>2
<inFulfillmentOf> .. </inFulfillmentOf>
<!-- The service documented and the primary laboratory having performed it -->
<documentationOf> .. </documentationOf>
<!-- zero or one encompassing encounter -->
<component><encompassingEncounter> .. </encompassingEncounter></component>
<!-- The body of the report -->
<component>
<structuredBody>
<component>
<section>
<templateId root='1.3.6.1.4.1.19376.1.8.1.2.1'/>
<code code='22636-5' displayName=’Pathology report relevant history'
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title>CLINICAL INFORMATION SECTION</title>
<text>
Tissue submitted: left breast biopsy and apical axillary tissue
</text>
<component>
<section>
<templateId root= '1.3.6.1.4.1.19376.1.5.3.1.3.1'/>
<code code='42349-1' displayName= ‘Reason for referral’
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title>Reason for anatomic pathology procedure</title>
<text>Breast mass - left breast</text>
</section>
</component>
<component>
<section>
<templateId root='1.3.6.1.4.1.19376.1.5.3.1.3.4'/>
<code code=’10164-2’ displayName= ‘History of present illness’
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title> History of present illness </title>
<text>Carcinoma of breast. Post operative diagnosis: same.
left UOQ breast mass.
</text>
</section>
</component>
</section>
</component>
<component>
<section>
<templateId root='1.3.6.1.4.1.19376.1.8.1.2.3'/>
<code code='22634-0' displayName='Pathology report gross observation'
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title>MACROSCOPIC OBSERVATION SECTION</title>
<text> </text>
<entry> <entry/>
:
</section>
</component>
<component>
<section>
<templateId root='1.3.6.1.4.1.19376.1.8.1.2.4'/>
<code code='22635-7' displayName='Pathology report microscopic observation'
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title>MICROSCOPIC OBSERVATION SECTION</title>
<text> </text>
<entry> <entry/>
:
</section>
</component>
<component>
<section>
<templateId root='1.3.6.1.4.1.19376.1.8.1.2.5'/>
<code code='22637-3' displayName='Pathology report diagnosis'
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title>DIAGNOSIS SECTION</title>
<text> </text>
<entry> <entry/>
:
</section>
</component>
<component>
<section>
<templateId root='1.3.6.1.4.1.19376.1.8.1.2.6'/>
<code code='46059-2' displayName=' Special treatments and procedures section '
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title>PROCEDURE STEPS SECTION</title>
<text> </text>
<entry> <entry/>
:
</section>
</component>
</structuredBody>
</component>
</ClinicalDocument>
Spezifikation
Die Spezifikation des Header-Abschnitts ist in jenem Abschnitt beschrieben.
Inwieweit die sehr umfangreiche Header-Spezifikation von IHE damit weiter eingeschränkt werden soll, muss entschieden werden. GH
In folgender Tabelle ist die Spezifikation des Body ausgeführt.
| Datenelement | Verw. | Kard. | Path and Constraints (Xpath + indentation) | Vokabular / Quelle | Bemerk. | DT | HL7 V2.5.1 |
| Report body | R | [1..1] | component/structuredBody | ||||
| Anrede Section | O | [0..1] | component/section
templateId[@root="2.999.889.777.22.77.10.10"] |
11 | |||
| Klinische Information Section | R2 | [0..1] | component/section
[/templateId/@root="1.3.6.1.4.1.19376.1.8.1.2.1"] |
PAT TF-3: 6.2.4.1 | 12 | OBR-13 | |
| Intraoperative Untersuchung
Section |
R2 | [0..1] | component/section
[/templateId/@root="1.3.6.1.4.1.19376.1.8.1.2.2"] |
PAT TF-3: 6.2.4.2 | 13 | ||
| Makroskopische Untersuchung
Section |
R | [1..1] | component/section
[/templateId/@root="1.3.6.1.4.1.19376.1.8.1.2.3"] |
PAT TF-3: 6.2.4.3 | 14 | ||
| Mikroskopische Untersuchung
Section |
R | [1..1] | component/section
[/templateId/@root="1.3.6.1.4.1.19376.1.8.1.2.4"] |
PAT TF-3: 6.2.4.4 | 15 | ||
| Diagnose Section | R | [1..1] | component/section
[/templateId/@root="1.3.6.1.4.1.19376.1.8.1.2.5"] |
PAT TF-3: 6.2.4.5 | 16 | ||
| Materialaufbereitung Section | O | [0..1] | component/section
[/templateId/@root="1.3.6.1.4.1.19376.1.8.1.2.6"] |
PAT TF-3: 6.2.4.6 | 17 | ||
| Schlusstext Section | O | [0..1] | component/section
templateId[@root=" ????? "] |
18 |
Bemerkungen:
(11): Die Anrede ist in DE im Pathologiebefund optional. Wenn vorhanden, wird sie wie im Arztbrief behandelt.
(12): Die Klinische Information Section enthält die Angaben des einsendenden Arztes: Anamnese, prä- und postoperative Diagnosen, Laborwerte, Probenbenennung, Probengewinnung, Fragestellung.
(13): Die Inraoperative Untersuchung Section enthält die Intraoperative (Gefrierschnitt-)Diagnose für jedes untersuchte Material, die Probenbeschreibung und die Benennung der intraoperativen Untersuchungsverfahren sowie für Spezialuntersuchungen abgezweigte Probenteile (z.B. Durchflusszytometrie, Zytogenetik, molekularpathologische Untersuchungen, Elektronenmikroskopie).
(14): Die Makroskopische Beschreibung Section enthält die Beschreibung aller erhaltenen Proben (Probentyp und -zustand), Links zu Makrofotos, die prozessurale Beschreibung und die Gewebsverwendung für zusätzliche Untersuchungsverfahren sowie die versendung in Biobanken.
(15): Die Mikroskopische Beschreibung Section enthält die histopathologischen Befunde zusammen mit den ergebnissen der zusätzlichen Untersuchungsverfahren (z.B. Histochemie, Immunhistochemie, Molekularpathologie).
(16): Die Diagnose Section enthält die Diagnosen aller eingesandten Proben eines Falles. Sie werden für jede Probe oder jede gruppe von Proben separat dargestellt. Die Section schließt zusätzliche Untersuchungsbefunde, Diagramme, statische bilder und Virtual Slides ein. Sie sammelt den minimalen Satz für die Diagnose relevanter Informationen.
(17): Die Materialaufbereitung Section enthält die Beschreibung der Gewebsaufarbeitung. Sie ist in deutschen Pathologiebefunden optional: repräsentative Proben und davon abgeleitete Proben für zusätzliche Untersuchungsmethoden oder Biobanken.
(18): Der Schlusstext ist in DE im Pathologiebefund optional. Wenn vorhanden, wird er wie im Arztbrief behandelt.
Organspezifischer Pathologiebefundbericht
| Template ID | 1.3.6.1.4.1.19376.1.8.1.1.2.1 - 20 (AP APSR 6.2.3.2) | |
| General Description | In diesem Template werden die Daten zum organspezifischen Pathologiebefundbericht übermittelt. | |
| Status | identisch zu IHE | |
| LOINC Code | Opt. | Description |
| 11526-1 | R2 | Pathology Study |
Definition und Zweck
Dieses Template definiert die Grundlage für die organspezifischen Einschränkungen von strukturierten Pathologiebefundberichten mit Tumordiagnosen. Das Parent Template ist 1.3.6.1.4.1.19376.1.8.1.1.1 (Pathologiebefundbericht), von dem es alle Eigenschaften erbt.
Von IHE wurden bisher 20 organspezifische Tumortemplates von Mammakarzinomen bis Larynxkarzinome spezifiziert (IHE_PAT_APSR_Rev1-1_TI_2011_03_31, Table 4.1.2.2-1).
Beispiel
<ClinicalDocument xmlns='urn:hl7-org:v3'>
<typeId extension="POCD_HD000040" root="2.16.840.1.113883.1.3"/>
<!-- conformance to a generic APSR content module -->
<templateId root='1.3.6.1.4.1.19376.1.8.1.1.1'/>
<!-- conformance to a breast content module -->
<templateId root='1.3.6.1.4.1.19376.1.8.1.1.2.1'/>
<!-- remainder of the header not shown, similar to the header content of a generic APSR -->
<component>
<structuredBody>
<!—Same hierarchy of sections and entries as in generic APSR -->
</structuredBody>
</component>
</ClinicalDocument>
Spezifikation
Die organspezifischen Document Content Module berücksichtigen die Hierarchie von <section> und <entry> Elementen des generischen Document Content Moduls sowie die Beschränkungen ihrer Parent-Templates.
Jedes organspezifische Document Content Modul besteht aus einer Vokabularsatzbeschränkung für die Content Module "AP Observation" und "Specimen Collection Procedure", welche in jedem der <entry> Elemente dieses Document Content Moduls verschachtelt sein können.