Pathologiebefund auf der Basis von CDA R2
|
|
Dieses Dokument gibt wieder:
Implementierungsleitfaden Pathologiebefund auf der Basis von CDA R2 (08). Die Teilmaterialien gehören der Kategorie cdapath an. |
HL7 Clinical Document Architecture Release 2
für das deutsche Gesundheitswesen
| Abstimmungsdokument | |||
|---|---|---|---|
| Version | Datum | Status | Realm |
| 08 | 2012 | ||
| noch kein download verfügbar | |||
| Kontributoren | ||
|---|---|---|
| Agfa HealthCare GmbH | Bonn | |

|
Vivantes Netzwerk für Gesundheit | Berlin |
| Prof.Dr. Gunter Haroske | Dresden | |
HL7 Deutschland e.V.
Geschäftsstelle Köln
An der Schanz 1
50735 Köln
Implementierungsleitfaden
Pathologie-Befunde auf Basisvon HL7 CDA Rel.2
zur Abstimmung durch die Mitglieder von HL7 Deutschland e.V.
Ansprechpartner:
Dr. Frank Oemig, Agfa HealthCare GmbH (Bonn)
Dokumentinformation
Inhaltsverzeichnis
- 1 Dokumentenhistorie
- 2 Editor
- 3 Autoren
- 4 Mit Beiträgen von
- 5 Autoren und Copyright-Hinweis, Nutzungshinweise
- 6 Einleitung
- 7 Dynamisches Modell
- 8 Statisches Modell
- 8.1 Übersicht
- 8.2 Gesamtstruktur
- 8.3 CDA-Header
- 8.4 CDA Body
- 8.4.1 Modell
- 8.4.2 Abschnitte ("Sections")
- 8.4.2.1 Anrede
- 8.4.2.2 Vorbefunde
- 8.4.2.3 Klinische Information / Fragestellung
- 8.4.2.4 Grundleiden/Todesursache (klin.)
- 8.4.2.5 Grundleiden/Todesursache (autoptisch)
- 8.4.2.6 Äußere Leichenschau
- 8.4.2.7 Innere leichenschau
- 8.4.2.8 Material
- 8.4.2.9 Materialaufbereitung
- 8.4.2.10 Makroskopische Beschreibung
- 8.4.2.11 Mikroskopische Beschreibung
- 8.4.2.12 Unterbeauftragung
- 8.4.2.13 Diagnosen
- 8.4.2.14 Zusammenfassung: ausführl. kritische gutachterliche Stellungnahme/Epikrise/Kommentar
- 8.4.2.15 Turmorformel
- 8.4.2.16 Diagnose(n) konsiliarischer Untersuchungen
- 8.4.2.17 Weitergabemodus
- 8.4.2.18 Gruß
- 8.4.2.19 Anlagen
- 8.4.2.20 Attribut-Wert-Paare
- 8.5 Beispiele für Befunde
- 9 Vokabeldomänen
- 10 Anhang A: Diverses
Dokumentenhistorie
| Version | Stand | Bearbeiter | Beschreibung | Dok.-OID |
|---|---|---|---|---|
| 06 | 13.04.12 | FO et.al. | Wikifizierung + Überarbeitung: Neustrukturierung | n.a. |
| 05 | 23.02.10 | FO | Überarbeitung: Neustrukturierung | n.a. |
| 04 | 16.11.09 | FO | Überarbeitung: Neustrukturierung | n.a. |
| 03 | 15.09.09 | FO | Überarbeitung | n.a. |
| 02 | 08.07.09 | FO | Überarbeitung | n.a. |
| 01 | 23.06.09 | IR | Dokument erstellt | n.a. |
Editor
Dr. Frank Oemig, AGFA HealthCare GmbH, Bonn
Autoren
- Ivonne Riedel, Agfa HealthCare GmbH, Bonn, (IR)
- Dr. Frank Oemig, Agfa HealthCare GmbH, Bonn (FO)
- Prof.Dr. Gunter Haroske, Dresden (GH)
Mit Beiträgen von
- Dr. Jochen Thümmler, Agfa HealthCare GmbH, früher bei Vivantes Netzwerk für Gesundheit, Berlin, (JT)
- Dr. Stefan Sabutsch, ELGA, Wien, (SS)
Autoren und Copyright-Hinweis, Nutzungshinweise
Nachnutzungs- bzw. Veröffentlichungsansprüche
Das vorliegende Dokument wurde von Agfa HealthCare GbmH, Bonn, und in Kooperation mit der HL7-Benutzergruppe e.V. entwickelt. Die Nachnutzungs- bzw. Veröffentlichungsansprüche sind nicht beschränkt.
Der Inhalt dieser Spezifikation ist öffentlich.
Zu beachten ist, dass Teile dieses Dokuments auf dem Abstimmungspaket 2 vom 17.Mai 2009 und der Normative Edition 2008 von HL7-Version 3 beruhen, für die © Health Level Seven, Inc. gilt. Näheres unter http://www.hl7.de/ und http://www.hl7.org/.
Die Erweiterung oder Ablehnung der Spezifikation, ganz oder in Teilen, ist dem Vorstand der Benutzergruppe und den Editoren/Autoren schriftlich anzuzeigen.
Alle auf nationale Verhältnisse angepassten und veröffentlichten HL7-Spezifkationen können ohne Lizenz- und Nutzungsgebühren in jeder Art von Anwendungssoftware verwendet werden.
Disclaimer
Obwohl diese Publikation mit größter Sorgfalt erstellt wurde, kann weder HL7 Deutschland e.V. noch die an der Erstellung beteiligten Firmen keinerlei Haftung für direkten oder indirekten Schaden übernehmen, die durch den Inhalt dieser Spezifikation entstehen könnten.
Einleitung
Einleitung
Dieses Dokument enthält einen ersten Entwurf für die Umsetzung von Pathologie-Berichten mit Hilfe von HL7 CDA R2. Exemplarisch soll diese Entwicklung für die Pathologieintegration innerhalb des Vivantes Netzwerks für Gesundheit, Berlin, prototypisch genutzt werden.
Orientiert wird dabei auf eine möglichst vollständige Berücksichtigung des "Leitfadens Pathologie/Neuropathologie (ehem. TM-30)" des Sektorkomitees Pathologie für die Anwendung der DIN EN ISO/IEC 17020 in der Pathologie/Neuropathologie.
Weiterhin wird angestrebt, die durch den Bundesverband Deutscher Pathologen und die Deutsche Gesellschaft für Pathologie veröffentlichten "Empfehlungen zur pathologisch-anatomischen Diagnostik von Kolorektalen Karzinomen, Mammakarzinomen und Prostatakarzinomen" in HL7 CDA R2 kompatible Templates zur Integration als Checklisten in Pathologie-Management-Systeme umzusetzen.
Auf dieser Basis soll der Import von HL7 CDA R2 Dokumenten von der Pathologie in KIS-Systeme sowie in Tumormeldungen und Qualitätssicherungs- und Tumordokumentationssysteme (z.B. AQUA, MaSC, ix.mid etc.) umgesetzt werden.
Grundlage
Grundlage dieses Konzeptes ist der Implementierungsleitfaden der VHitG für den Arztbrief des deutschen Gesundheitswesens sowie der Diagnose- und Datentypleitfaden.
- VHitG Arztbrief, v1.5, [CDAr2Arztbrief], 2006
- Diagnoseleitfaden v0.99b, 13.12.09
- Datentypleitfaden
|
|
Bei IHE Anatomic Pathology sind zwei Trial Implementation seit 31.3.11 im Netz: "Anatomic Pathology reporting to Public Health (ARPH)" und "Anatomic Pathology Structured Reports (APSR)"(http://www.ihe.net/), das letztere auch mit einem Appendix für das Value Set.
Hierbei handelt es sich jedoch um HL7 v2.5 ORU Nachrichten, so dass die Inhalte nicht direkt weiter genutzt werden können! Ist das noch korrekt? G.H. |
Disclaimer
Dieses Dokument enthält keine komplette Spezifikation eines HL7 CDA R2 Arztbriefes bzw. Dokumentes. Es werden Teile eines Arztbriefes spezifiziert, wie er im Rahmen der Pathologieintegration innerhalb der Vivantes Gruppe benötigt werden. Ziel dieser Integration soll es sein, alle für die onkologische Tumordokumentation relevanten Daten in ORBIS zu importieren. Eine Vollständigkeit des Arztbriefes kann daher nicht gewährleistet werden.
Weiterhin wird nur eine unidirektionale Kommunikation des HL7 CDA Arztbriefes spezifiziert – Import nach ORBIS.
Dynamisches Modell
Übersicht
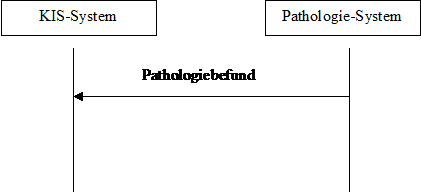
Das dynamische Modell sieht das relativ einfach aus:
Abbildung 1: dynamisches Modell
Im Prinzip agiert das Pathologiesystem als Content Creator und das KIS-System als Content Consumer. Entsprechend können auch andere Systeme diese beiden Rollen übernehmen.
Statisches Modell
Übersicht
In diesem Abschnitt wird grob der Aufbau und die Struktur von HL7 CDA R2 Dokumenten erläutert (entnommen aus dem Implementierungsleitfaden des VHitG-Arztbriefes, Kapitel 3).
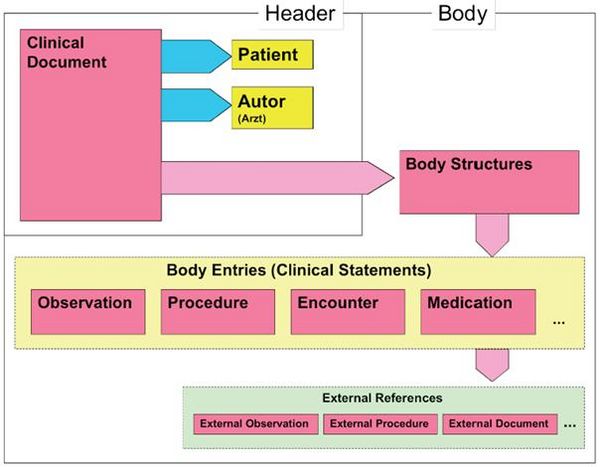
Wie alle Spezifikationen von Nachrichten in HL7 basiert auch die Clinical Document Architecture auf dem RIM und ist als HL7 V3 Modell repräsentiert. Grob gesprochen besteht ein CDA Dokument aus einem Header und einem Body, der wiederum Body Structures und Body Entries aufweist. An die Entries können externe Referenzen (External References) geknüpft sein. Der folgende Überblick zeigt die Hauptkomponenten des CDA R2 Modells auf und in der folgenden Abbildung ist das Ganze in XML-artiger Darstellung gezeigt.
Abbildung 2: CDA-RMIM (vereinfachte Darstellung)
Die nachfolgende vereinfachte Graphik zeigt die Darstellung in XML:
Abbildung 3: CDA Level 3 Entries (vereinfachter Ausschnitt)
Die Informationen zum Patienten, zum Dokument selbst, zu den weiteren beteiligten Personen und Organisationen sowie der dokumentierten Episode (Zeitereignisse) sind zum CDA Header zusammengefasst, hochstrukturiert und von der Semantik her festgelegt.
Die Informationen im Header unterstützen einen Austausch klinischer Dokumente über Institutionsgrenzen hinweg. Er trägt Informationen über das Dokument selbst (eine eineindeutige Identifikation, eine Andeutung des Typs des Dokuments), über „Teilnehmer" am Dokument (an der Dokumentation beteiligte Heilberufler, Autoren, und natürlich den Patienten selbst), sowie über Beziehungen zu Dokumenten (zu Anforderungen und anderen Dokumenten). Mit den Informationen des Headers werden Dokumentenmanagementsysteme unterstützt, der Header stellt dafür entsprechende Mechanismen zur Verfügung. Schließlich hat man mit den im CDA Header verfügbaren Informationen die Zusammenführung einer individuellen (lebenslangen) Patientenakte vor Augen.
Gesamtstruktur
|
|
Anm.: Die folgende Abbildung muss noch an die im weiteren ausgeführten Gliederungen angepasst werden. |
Abbildung 4: Gesamtstruktur
Dokumenttypen
|
|
Die im VHitG-Arztbrief vorgeschlagenen Dokumententypisierung (Surgical pathology report und Autopsy report) könnte bereits ausreichend sein. Zweit-/Nachberichte, Konsiliarberichte und Zytologische Befunde weisen die gleiche Grundstruktur auf und sollten daher keine separaten Dokumententypen sein.
Für Zweit-, besser Nachberichte gilt, dass sie neue Informationen zu einem bereits vorhandenen Befund hinzufügen (nachträgliche Spezialuntersuchungen, Zweitmeinungen, Konsiliarbefunde, Antworten auf klinische Fragestellungen, etc.). Sie beziehen sich immer auf einen vorhandenen Befund, in dem sie u.U. auch schon angekündigt werden (siehe ELGA-Beispiel). Ein Konsiliarbefund stellt einen eigenständigen Erstbefund dar, der vom konsilsuchenden Auftraggeber (Pathologe) in dessen Erstbefund aufgenommen oder als Nachbericht an einen Erstbefund angefügt wird. Gemäß des HL7-Moduls Dokumenttypen lauten die Spezifikationen für Pathologisch-anatomische Begutachtung
Obduktionsgutachten
Die Taxonomie dieses Moduls, betreffend Pathologiebefunde, erscheint nicht durchgängig logisch und sollte nochmals diskutiert werden.
Die Grundstruktur eines Befundes ist: Material - Makroskopische Beurteilung - Mikroskopische Beurteilung - Diagnose - Zusammenfassung. Die Klinische Fragestellung wird von einigen Kollegen in den Befund übernommen, sinnvoll wäre ihre Berücksichtigung bei einer bidirektionalen Verbindung zum KIS. Immunhistologischer, elektronenmikroskopischer und molekularpathologischer Befund sind Ergebnis der pathologischen Stufendiagnostik und in der Regel im Befund eingearbeitet. Tabellarische strukturierte Darstellungen (auch Checklisten) sollten als Entry vorgesehen werden. |
| Lvl | Dokumenttyp Abschnitt |
Pathologisch- anatomische Begutachtung/ Erstbericht |
Obduktions-/ Sektions- gutachten |
LOINC | Beschreibung | CDA- Level |
|---|---|---|---|---|---|---|
| 1 | Anrede | 0..1 | 0..1 | 1 | ||
| 1 | Vorbefunde | 0..1 | 0..1 | 1 | ||
| 1 | Klinische Informationen | 0..1 | 0..1 | 1 | ||
| 2 | Fragestellung | 0..1 | 0..1 | 1 | ||
| 1 | Grundleiden/Todesursache (klinisch) | 1..1 | 2 | |||
| 1 | Grundleiden/Todesursache (autoptisch) | 1..1 | 2 | |||
| 2 | Äußere Leichenschau | 1..1 | 1 | |||
| 2 | Innere Leichenschau | 1..1 | 1 | |||
| 1 | Material | 1..* | 1 | |||
| 2 | Materialaufbereitung | 1..* | 1..* | 46059-2 | 1 | |
| 1 | Makroskopische Beschreibung | 1..* | 1..* | 22634-0 | 1 | |
| 2 | Intraoperativer Schnellschnitt | 0..* | 2 | |||
| 1 | Mikroskopische Beschreibung | 1..* | 1..1 | 22635-7 | 1 | |
| 2 | Immunhistologie | 0..* | 0..* | 3 | ||
| 2 | Molekularpathologie | 0..* | 0..* | 1 | ||
| 2 | Elektronenmikroskopie | 0..* | 0..* | 1 | ||
| 2 | Präparatradiographie | 0..* | 1 | |||
| 1 | Unterbeauftragung | 0..* | 0..* | 1 | ||
| 1 | Diagnose(n) konsiliarischer Untersuchungen | 0..* | 0..* | 1 | ||
| 1 | Diagnose | 1..* | 22637-3 | 3 | ||
| 1 | ausführliche kritische gutachterliche Stellungnahme/Epikrise/Kommentar | 0..1 | 0..1 | 35660-0 | 1 | |
| 1 | Verschlüsselung/ Stadium/spezielle Schlüssel | 0..* | 0..* | 3 | ||
| 1 | Weitergabemodus | 0..1 | 0..1 | 2 | ||
| 1 | Gruß | 0..1 | 0..1 | 1 | ||
| 1 | Anlagen | 0..* | 0..* | |||
| 1 | immunhistologische Tabelle | 0..1 | 0..1 | 3 | ||
| 1 | molekularpathologische Tabelle | 0..1 | 0..1 | 2 | ||
| 1 | Checklisten | 0..* | 3 | |||
| 1 | weitere Attribut-Wert-Paare | 0..* | 0..* | 3 |
Tabelle 1: Dokumenttypen und deren Inhalt und zugehörige LOINC-Codes
CDA-Header
Die Regelungen zum Header können aus dem VHitG-Arztbrief, Kapitel 7, vollständig übernommen werden. Einzig die Liste der teilnehmenden Personen (participants) ist um den Einsender zu ergänzen, der in der Regel auch der beabsichtigte Empfänger des Dokuments (informationRecipient) ist. Er ist nicht identisch mit einem Ein- oder Überweiser.
IHE_APSR_TF_Supplement-2011-03-31 schlägt dafür noch zwei weitere participant-Rollen vor: - Ordering physician (participant[@typeCode="REF"]/templateId[@root="1.3.6.1.4.1.19376.1.3.3.1.6"], HL-7 ORC-12, OBR-16, ORC-9 - Specimen collector (participant[@typeCode="DIST"]/templateId[@root="1.3.6.1.4.1.19376.1.8.1.4.1"], HL-7 OBR-10, SPM-17.
Beide in der usage-Kategorie R2 (required, if known)
Die weiteren Ausführungen erübrigen sich damit bis auf die Dokumententypisierung!
Alle XML Arztbriefe beginnen mit dem Wurzelelement ClinicalDocument und der vorgeschriebene Zeichensatz ist UTF-8.
Daraus ergibt sich folgende Struktur, die wie aufgeführt umzusetzen ist. Dabei sind fett gedruckte Bereiche unverändert einzubauen.
<?xml version="1.0"? encoding="UTF-8">
<ClinicalDocument
xmlns="urn:hl7-org:v3"
xmlns:voc="urn:hl7-org:v3/voc"
xmlns:xsi="http://www.w3.org/2001/XMLSchema-instance">
<typeId root="2.16.840.1.113883.1.3" extension="POCD_HD000040"/>
<!-- CDA Header -->
... siehe Beschreibung CDA R2 Header
<!-- CDA Body -->
<component>
<structuredBody>
... siehe Beschreibung CDA R2 Body
</structuredBody>
</component>
</ClinicalDocument>
In diesem Abschnitt werden die Elemente des CDA Headers erläutert, die zwingend in den CDA HL7 R2 Arztbrief einzubinden sind.
| < | Element | DT | Card | Conf | Beschreibung | |
|---|---|---|---|---|---|---|
| ClinicalDocument | 1..1 | M | Dokument |
Dokumenten-ID
| @ | Attribut | DT | Card | Conf | Beschreibung | |
|---|---|---|---|---|---|---|
| id | II | 1..1 | Dokumenten-ID |
Jeder Arztbrief muss genau eine eindeutige DokumentenID aufweisen. Diese DokumentenID identifiziert ein Dokument weltweit und für alle eindeutig.
Diese muss folgendermaßen aussehen:
<id extension="13234453645" root="2.16.840.1.113883.2.6.15.3.427.1"/>
Das @extension Attribut enthält eine eindeutige Dokumentennummer, die von der in @root genannten Authority vergeben wird. Im @root Attribut wird das Dokument-erzeugende Anwendungssystem über eine OID identifiziert: Für die Kommunikation nach außen muss eine OID gewählt werden, die eindeutig für die Instanz des Anwendersystems ist. In der Regel werden diese OIDs vom Hersteller des jeweiligen Anwendersystems kommen, der seine tatsächlichen Installationen (Applikations-Instanzen) mit entsprechenden eindeutigen OIDs zu versehen hat. Das heißt, dass jede Installation eines Anbieters eine eindeutige OID besitzt und verwendet.
Typisierung des Dokuments
| @ | Attribut | DT | Card | Conf | Beschreibung | |
|---|---|---|---|---|---|---|
| code | CE CWE | 1..1 | Typ des Dokuments |
Über das @code Attribut wird eine Typisierung des Dokuments vorgenommen.
[Im Falle der Integration des Pathologiesystems von Vivantes ist folgender Eintrag zu verwenden.]
| Code | Dokumenten-Typ | Deutsche Bezeichnung | Berufsgruppe | Umgebung |
|---|---|---|---|---|
| 11529-5 | Surgical pathology report | Pathologischer Befundbericht/Pathologisch-anatomische Begutachtung | ||
| 11526-1 | Pathology study | Pathologischer Befundbericht/Pathologisch-anatomische Begutachtung | ||
| 18743-5 | Autopsie report | Autopsiebericht/Obduktionsbericht |
Tabelle 3: LOINC-Codes für Dokumenttypen (OID 2.16.840.1.113883.6.1)
Die LOINC-Codes 11526-1 und 11529-5 beschreiben offenbar vollständig identische Sachverhalte. Da IHE-APSR ausschließlich 11526-1 verwendet, sollte dieser auch in HL-7 verwendet werden. Dies steht im Widerspruch zum VHitG-Arztbrief, der den Code 11529-5 vorsieht!
[Weitere Typen sind bei Interesse dem Implementierungsleitfaden der VHitG S. 46 Tabelle 3 zu entnehmen.]
Für das @code Attribut wird das LOINC Codesystem verwendet. Es muss das @codeSystem Attribut daher mit dem OID des LOINC gefüllt werden.
<code code="11526-1" codeSystem="2.16.840.1.113883.6.1"/>
Titel
| @ | Attribut | DT | Card | Conf | Beschreibung | |
|---|---|---|---|---|---|---|
| title | ST | 1..1 | Title des Dokuments |
<title>Pathologisch anatomische Begutachtung [mit kritischer Stellungnahme]</title>
Erstellungsdatum
| @ | Attribut | DT | Card | Conf | Beschreibung | |
|---|---|---|---|---|---|---|
| effectiveTime | TS | 1..1 | Erstellungsdatum |
Das @effectiveTime Attribut enthält das Erstellungdatum des Dokumentes. Es muss mindestens eine Jahres-, Montats- und Tagesangabe enthalten. Eine Stunden- und Minutenangabe ist optional.
<effectiveTime value="200509241634"/>
Patient
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.7 ???? | |
| General Description | In diesem Abschnitt im CDA Header wird der Patient beschrieben/erfasst. Dieser setzt sich zusammen aus einer Patientenrolle sowie dem Patienten selbst. Diese werden im recordTarget zusammengeführt. | |
| LOINC Code | Opt. | Description |
| ???? | O | |
Im CDA-Header muss mindestens eine Patientenrolle beschrieben sein, die genau von einer Person gespielt wird.
| Lvl | RIM | AE | Name | Desc | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|---|---|
| 1 | part | elm | recordTarget | Patient | 1..1 | required | ||
| 2 | role | elm | patientRole | Patient | 1..1 | required | Die Rolle des Patienten wird durch eine Person gespielt.
| |
| 3 | role | elm | id | ID des Patienten | 1..1 | required | Verpflichtend muss in diesem Bereich die Patientenidentifikationsnummer angegeben werden. Diese setzt sich zusammen aus dem @extension Attribut, das die ID des Patienten enthält sowie dem @root Attribut, das die OID des Systems enthält, das die ID vergeben hat.
| |
| 4 | role | att | extension | ID des Patienten | 1..1 | required | ||
| 4 | role | att | root | ID des Patienten | 1..1 | required |
| |
| 3 | ent | elm | Person | 0..1 | optional | Patient | ||
| 4 | ent | elm | Person.name | Name des Patienten | 0..1 | optional | In dem Attribut @name ist der Name des Patienten untergebracht. Der Name wird wiederrum unterteilt in die @given und @family Attribute, die den Vornamen und den Familiennamen des Patienten enthalten. |
Ein kompletter recordTarget ist im Folgenden angegeben.
<recordTarget>
<!--- Patienten-Daten -->
<patientRole>
<id extension="6245" root="2.16.840.1.113883.3.933"/>
<patient>
<name>
<prefix>Dr.</prefix>
<given>Paul</given>
<family>Pappel</family>
</name>
</patient>
</patientRole>
</recordTarget>
Autor
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.7 ???? | |
| General Description | Autor des Berichts | |
| LOINC Code | Opt. | Description |
| ???? | O | |
| Lvl | RIM | AE | Name | Desc | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|---|---|
| 1 | part | elm | author | Autor | 1..1 | required | Autor: Neben dem Patienten muss ein Autor (author) angegeben werden, welcher das Dokument verfasst hat. | |
| 2 | part | elm | time | Zeitpunkt | TS | 1..1 | required | Im verpflichtend anzugebenden @time Attribut wird der Zeitpunkt der Dokumentation angegeben. |
| 2 | role | att | auhtor | Autor | 1..1 | required | Informationen über den Autor werden in der assignedAuthor Klasse angegeben. | |
| 4 | ent | elm | Author.name | Name des Autors | PN | 0..1 | optional | In dem Attribut @name ist der Name des Autors untergebracht. Der Name wird wiederum unterteilt in die @given und @family Attribute, die den Vornamen und den Familiennamen des Autors enthalten. |
Ein kompletter author ist im Folgenden angegeben.
<author>
<time value="20050829"/>
<assignedAuthor>
<id extension="190388km89" root="2.16.840.1.113883.3.24535"/>
<assignedPerson>
<name>
<prefix>Dr.med.</prefix>
<given>Theo</given>
<family>Phyllin</family>
</name>
</assignedPerson>
</assignedAuthor>
</author>
Verwaltende Organisation
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.7 ???? | |
| General Description | verwaltende Organisation. | |
| LOINC Code | Opt. | Description |
| ???? | O | |
| Lvl | RIM | AE | Name | Desc | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|---|---|
| 1 | ent | elm | custodian | verwaltende Organisation | 1..1 | required | Patient | |
| 2 | ent | elm | id | Identifkation |
Die Organisation (custodian), die für die Verwaltung des Dokuments verantwortlich ist, muss verpflichtend in der entsprechenden Klasse wiedergegeben werden.
Die Organisation muss mindestens mit einer ID gekennzeichnet werden.
Ein kompletter custodian ist im Folgenden angegeben.
<custodian>
<assignedCustodian>
<representedCustodianOrganization>
<id extension="175648374" root="1.2.276.0.76.4.5">
<name>
...
</name>
</representedCustodianOrganization>
</assignedCustodian>
</custodian>
CDA Body
Die eigentliche klinische Dokumentation wird im so genannten CDA Body festgehalten. Im Vordergrund steht hier „lesbarer" (narrativer) Text, der verpflichtender Bestandteil von CDA R2 Dokumenten ist und die Interoperabilität zwischen den menschlichen Kommunikationspartnern garantiert. Hier sind Möglichkeiten gegeben, diesen Text grob zu strukturieren, wie man dies von den Möglichkeiten der Textverarbeitung her kennt. Zur Strukturierung stellt die Standardspezifikation eine Reihe von XML-Elementen zur Verfügung, die als Body Structures zusammengefasst werden können. Der Body enthält ein oder mehrere Abschnitte (sections). Diese können auch ineinander geschachtelt sein, so wie Kapitel und Unterkapitel in einem Buch. Zudem sind Strukturierungen im textuellen Bereich im Sinne von Tabellen oder Listen möglich.
- Abschnitte <section>
- Paragrafen <paragraph>
- Kennzeichnung von bestimmten Inhalten <content>
- Überschriften <caption>
- Tabellen <table>
- Listen <list>
Sections enthalten immer einen narrativen Block und erfüllen damit eine der oben genannten Maximen von CDA: die Mensch-zu-Mensch-Interoperabilität, die Lesbarkeit der Informationen für den Menschen. Im narrativen Block, durch das Textattribut in der section-Klasse repräsentiert, wird eingebetteter Text innerhalb eines Abschnittes angegeben. Dabei kann mit oben genanntem <content> Element bestimmter Inhalt gesondert gekennzeichnet werden.
Zusammengefasst werden im Textblock (teils so auch schon in CDA Release 1 realisiert) u.a. folgende Möglichkeiten der Struktur- und Formgebung des fließenden Textes gegeben:
- Zeilenumbrüche <br>
- Stilistische Angaben (unterstreichen, fett, kursiv etc.)
- Hoch- und Tiefstellung von Text
- Fußnoten
- Symbole
- Revisionsmarken im Text wie <delete>, <insert>
Mit den beschriebenen Body Strukturen können CDA Entries verbunden sein. Diese repräsentieren den „computerlesbaren Teil" innerhalb eines Dokumentenabschnitts. Body Entries sind im Prinzip eine Auswahl aus Klassen mitsamt Attributen aus dem HL7 Referenz-Informationsmodell (RIM).
Modell
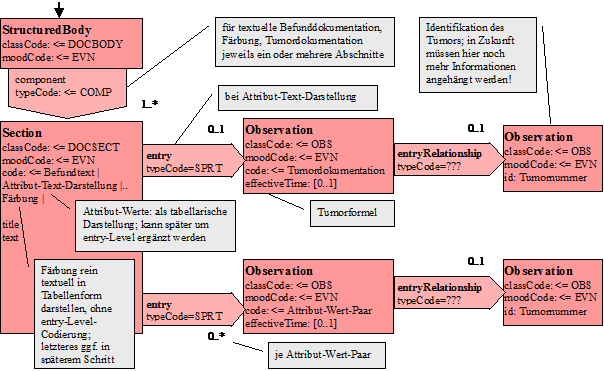
Nachfolgend ist das CDA-Modell angegeben, so wie es für den Pathologie-Bericht instanziiert wird:
Abbildung 5: Level-3-Modell
Abschnitte ("Sections")
Im folgenden sollen die einzelnen Abschnitte näher spezifiziert werden.
Anrede
| Template ID | <OID für das Template> | |
| General Description | In diesem Abschnitt wird die Anrede formuliert. Daten sind keine Enthalten. Dieser Abschnitt ist bereits Bestandteil des VHitG-Arztbriefes. | |
| LOINC Code | Opt. | Description |
| ???? | O | |
<section>
<templateId root='1.3.6.1.4.1.19376.1.8.?????'/>
<code code='?????' displayName=’??????'
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title>Anrede</title>
<text>Sehr geehrter Herr Kollege, ... </text>
</section>
Vorbefunde
| Template ID | <OID für das Template> | |
| General Description | In diesem Abschnitt werden die Informationen zu Vorbefunden übermittelt. | |
| LOINC Code | Opt. | Description |
| ???? | O | |
Vorbefunde werden i.d.R. vom Pathologie-Management-System (PMS) bereit gestellt. Im Strukturierten Befund sollten sie allenfalls mit der jeweiligen Fall-Nr. aufgeführt werden, sofern sie Relevanz zum aktuellen Befund besitzen.
Klinische Information / Fragestellung
| Template ID | <OID für das Template> | |
| General Description | In diesem Abschnitt werden die klinischen Informationen übermittelt. | |
| LOINC Code | Opt. | Description |
| ???? | O | |
Vorgeschichte, Laborbefunde etc. werden als "Clinical Information Section", mit weiteren Subsections "Reason for referral" und " History of Present Illness" zusammengefasst.
IHE schlägt dazu folgendes Beispiel vor:
<section>
<templateId root='1.3.6.1.4.1.19376.1.8.1.2.1'/>
<code code='22636-5' displayName=’Pathology report relevant history'
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title>CLINICAL INFORMATION SECTION</title>
<text>Tissue submitted: left breast biopsy and apical axillary tissue </text>
<component>
<section>
<templateId root= '1.3.6.1.4.1.19376.1.5.3.1.3.1'/>
<code code='42349-1' displayName= ‘Reason for referral’
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title>Reason for anatomic pathology procedure</title>
<text>Breast mass - left breast</text>
</section>
</component>
<component>
<section>
<templateId root='1.3.6.1.4.1.19376.1.5.3.1.3.4'/>
<code code=’10164-2’ displayName= ‘History of present illness’
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title> History of present illness </title>
<text>Carcinoma of breast. Post operative diagnosis:
same.left UOQ breast mass.</text>
</section>
</component></section>
Das IHE-Beispiel ist nicht ganz konsistent, da LOINC-Code display name und Title nicht vollständig übereinstimmen. Außerdem ist hier die Materialangabe unkodiert vorgenommen worden
Grundleiden/Todesursache (klin.)
| Template ID | <OID für das Template> | |
| General Description | In diesem Abschnitt werden die Informationen zur klin. Todesursache übermittelt. | |
| LOINC Code | Opt. | Description |
| ???? | O | |
Dieser Abschnitt ist nur Bestandteil des Obduktionsbefundes / Sektionsberichtes. Im Einzelnen werden hier tabellarisch folgende Items aufgelistet:
| Bedeutung | Diagnosetext | Zeitdauer zwischen Krankheit und Tod |
ICD-10 Code |
|---|---|---|---|
| Unmittelbar zum Tode führende Krankheit (Todesursache) | Ia) 1..1 | 0..1 | 1..1 |
| Vorausgegangene Ursache | Ib) 0..1 | 0..1 | 0..1 |
| Vorausgegangene Ursache (Grundleiden) | Ic) 0..1 | 0..1 | 0..1 |
| Begleitkrankheit | II 0..1 | 0..1 | 0..1 |
| Begleitkrankheit | II 0..1 | 0..1 | 0..1 |
Was ist hier mit den Codes gemeint?
Für diese Tabellen wurden noch keine Codes vorgeschlagen
Grundleiden/Todesursache (autoptisch)
| Template ID | <OID für das Template> | |
| General Description | In diesem Abschnitt werden die Informationen zur autoptischen Todesursache übermittelt. | |
| LOINC Code | Opt. | Description |
| ???? | O | |
Dieser Abschnitt ist nur Bestandteil des Obduktionsbefundes / Sektionsberichtes. Im Einzelnen werden hier tabellarisch folgende Items aufgelistet:
| Bedeutung | Diagnosetext | ICD-10 Code |
|---|---|---|
| Unmittelbar zum Tode führende Krankheit (Todesursache) | Ia) 1..1 | 1..1 |
| Vorausgegangene Ursache | Ib) 0..1 | 0..1 |
| Vorausgegangene Ursache (Grundleiden) | Ic) 0..1 | 0..1 |
| Begleitkrankheit | II 0..1 | 0..1 |
| Begleitkrankheit | II 0..1 | 0..1 |
Was ist hier mit den Codes gemeint?
Für diese Tabellen wurden noch keine Codes vorgeschlagen
Äußere Leichenschau
| Template ID | <OID für das Template> | |
| General Description | In diesem Abschnitt werden die Informationen zur äußeren Leichenschau übermittelt. | |
| LOINC Code | Opt. | Description |
| ???? | O | |
tbd
Innere leichenschau
| Template ID | <OID für das Template> | |
| General Description | In diesem Abschnitt werden die Informationen zur inneren Leichenschau übermittelt. | |
| LOINC Code | Opt. | Description |
| ???? | O | |
tbd
Material
| Template ID | <OID für das Template> | |
| General Description | In diesem Abschnitt werden die Informationen zum Material übermittelt. | |
| LOINC Code | Opt. | Description |
| ???? | O | |
Dieser Abschnitt spielt eine zentrale Rolle bei der Organisation des gesamten Befundberichtes.
Das Konzept lautet: "Untersuchungsmaterial, d. h. bioptisch, operativ oder durch Punktion oder durch Sammeln gewonnenes Gewebs- oder Zellmaterial sowie Körperflüssigkeiten, das zur pathologisch-anatomischen Begutachtung eingesandt wurde. Jedes vom Einsender in separatem Gefäß ("container") übersandtes oder auf dem Untersuchungsantrag separat bezeichnetes Untersuchungsmaterial ist innerhalb einer Begutachtung auch separat zu behandeln. Die Materialbezeichnung richtet sich nach der des Einsenders".
Jedes Material ist makroskopisch und mikroskopisch zu beschreiben. Die diagnostische Bewertung kann mehrere Materialien synoptisch behandeln.
Synonyme: Untersuchungsmaterial, Probe, Probenmaterial
Die verschiedenen Funktionen und Ausprägungen finden sich auch in IHE_PAT_Suppl_APSR_Rev1-1_TI_2011 unter dem Stichwort "Specimen", siehe auch die use cases in IHE_PAT_TF-1.
Der Befund wird in jeder Section organisiert nach dem Material (specimen or group of specimens)
Materialaufbereitung
| Template ID | <OID für das Template> | |
| General Description | In diesem Abschnitt werden die Informationen zur Materialaufbereitung übermittelt. | |
| LOINC Code | Opt. | Description |
| ???? | O | |
Für diese Section ist durch IHE APSR vorgeschlagen, "Procedure Steps" zu verwenden (OID 1.3.6.1.4.1.19376.1.8.1.2.6).
Als ein Template soll es in einem entry-Element eingesetzt werden. Für den Prozedurencode und den target site code gibt es vorgeschlagene value sets.
Diese Section beschreibt die Materialaufbereitung: Repräsentative Proben und davon abgeleitete Gewebsproben für weitere Untersuchungstechniken (Flowzytometrie, zytogenetische und molekularpathologische Untersuchungen, Elektronenmikroskopie etc.) oder Biobanken.
Diese Section muss ein Code Element enthalten, das mit folgenden Attributen gefüllt ist:
| Lvl | RIM | AE | Name | Desc | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|---|---|
| 1 | act | elm | title | 1..1 | required | |||
| 1 | act | elm | text | 1..1 | required | Diese Section soll ein Text Element enthalten, welches die Information lesbar enthält. | ||
| 1 | act | elm | code | CD | 1..1 | required | ||
| 2 | act | att | @code="46059-2" | 1..1 | required | |||
| 2 | act | att | @codeSystem="2.16.840.1.113883.6.1" | 1..1 | required | |||
| 2 | act | att | @displayName="Special treatments and procedures section" | 1..1 | required | |||
| 2 | part | elm | 1..1 | required | ||||
| 3 | role | elm | author | 1..1 | required | Diese Section sollte ein Autor Element enthalten, wenn der Autor dieser Section von dem in höheren Ebenen des Dokuments verschieden ist.
| ||
| 4 | ent | elm | Person | 0..1 | optional | Arzt |
Diese Section enthält keine Subsections oder Entries.
Beispiel:
<component>
<section>
<templateId root='1.3.6.1.4.1.19376.1.8.1.2.6'/>
<code code='46059-2' displayName='Special treatments and procedures section'
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title>PROCEDURE STEPS</title>
<text>
Probe 1 (bezeichnet mit “Mammabiopsie links”) wird für Gefrierschnitt aufgearbeitet.
Restgewebe für Paraffinhistologie.
Probe 2 (bezeichnet als “ax.Fettgewebe apikal”), in zwei Teilen übersandt, wird vollständig gebettet und mit zahlreichen Schnittstufen untersucht.
</text>
</section>
</component>
Codes für immunhistochemische Färbungen
Antikörper
| Code | Antikörper | Bedeutung |
|---|---|---|
| 40563-9 | Aktin(glattmuskulär)) | reagiert mit Aktin in glatter Muskulatur |
| 10463-8 | Amyloid A | reagiert mit Amyloid vom Typ AA |
| ???? | CD45 | reagiert mit LCA |
| u.s.w. |
Tabelle 6: Antikörper (OID: 2.16.840.1.113883.6.1)
Antikörperklone
| Klon (Code) | Bedeutung |
|---|---|
| 1A4 | monoklonaler Maus-AK gegen glattmuskuläres Aktin |
| mc1 | monoklonaler Maus-AK gegen Amyloid vom Typ AA |
| 2B11 | monoklonaler Maus-AK gegen LCA |
| PD7/26 | monoklonaler Maus-AK gegen LCA |
| u.s.w. |
Tabelle 7: Klone monoklonaler Antikörper (OID: 1.2.276.0.76.5.????)
Antikörperhersteller
| OID | Hersteller |
|---|---|
| 1.3.6.1.4.1.11987 | DAKO |
| 2.16.840.1.113995 | Roche |
| ???? | Zytomed |
| 1.3.6.1.4.1.22868 | Ventana |
| 1.3.6.1.4.1.492 | DCS |
| u.s.w. |
Tabelle 8: Hersteller von Antikörpern (OID: 1.2.276.0.76.5.????)
Die Hersteller werden über die Klasse Entity mit @id identifiziert. Insofern wird diese Tabelle so nicht benötigt.
Antikörperklasse
| Code | Antikörperklasse | Bedeutung |
|---|---|---|
| 1 | Klasse I | AK für diagnostische Immunhisto-/-zytochemie,
die im Kontext mit Morphologie und klinischen Daten interpretiert werden. Sie dienen der Zelldifferenzierung. |
| 2 | Klasse II | AK für diagnostische Immunhisto-/-zytochemie mit
direkter Therapierelevanz, die als Einzelergebnis semiquantitativ bewertet werden. |
| 3 | CE | AK mit CE-Kennzeichnung.
Die Validierungsdaten wurden vom Hersteller erhoben. |
Tabelle 9: Antikörperklasse (OID: ????)
Protokoll der Färbung
| Protokoll-ID | |
|---|---|
| xyz | |
| abc | |
| u.s.w. |
Tabelle 10: ID des verwendeten Protokolls im Immunfärbeautomaten (OID: 1.2.276.0.76.5.????)
Woraus ergibt sich diese Information? Wird die generiert? Dann ist es eine Instanz-ID!
Färbeintensität
| Code | Färbeintensität | Bedeutung |
|---|---|---|
| 0 | keine | keine Reaktion |
| 1 | schwach | schwache Reaktion (im Vergleich zur Positivkontrolle) |
| 2 | mittel | mäßig starke Reaktion (im Vergleich zur Positivkontrolle) |
| 3 | stark | starke Reaktion (im Vergleich zur Positivkontrolle) |
Tabelle 11: Färbeintensität (OID: 1.2.276.0.76.5.????)
Färbemuster
| Code | Färbemuster | Bedeutung |
|---|---|---|
| 0 | keine Färbung | keine Reaktion |
| 1 | nukleär | Kerne gefärbt |
| 2 | zytoplasmatisch | Zytoplasma gefärbt |
| 3 | membranständig_komplett | Zellmembran vollständig gefärbt |
| 4 | membranständig_partiell | Zellmembran teilweise gefärbt |
| 5 | Kombination aus 1-3 oder 4 | mehrere Zellstrukturen gefärbt |
Tabelle 12: Färbemuster(OID: 1.2.276.0.76.5.????)
Verteilungsmuster der gefärbten Objekte
| Code | Verteilungsmuster | Bedeutung |
|---|---|---|
| 0 | keine Färbung | keine Objekte gefärbt |
| 1 | diffus | homogene Verteilung der gefärbten Objekte |
| 2 | fokal | Objekte nur herdförmig gefärbt |
| 3 | basal | Basalzellschicht gefärbt |
| 4 | luminal | lumenseitige Zellschicht gefärbt |
| 5 | mosaik | Mosaikmuster gefärbter Objekte im Verband |
Tabelle 13: Verteilungsmuster der gefärbten Objekte (OID: 1.2.276.0.76.5.????)
Anteil gefärbter Objekte
| Anteil positiver Objekte | Bedeutung |
|---|---|
| Anteil gefärbter Objekte an allen Objekten gleicher Art in % |
Tabelle 14: Anteil positiver Zellen (OID: 1.2.276.0.76.5.????)
Sollte als physical quantity übermittelt werden.
Gewebetyp
| Code | Gewebetyp | Bedeutung |
|---|---|---|
| 1 | Normalgewebe | nichtpathologisch verändertes Gewebe (in der Umgebung der Läsion) |
| 2 | Tumorgewebe | Tumorgewebe (Läsion) |
| 3 | Zellinie | Zelllinie mit definiertem Reaktionsausfall |
| 4 | Positive Gewebskontrolle (extern) | Als geeignet bekanntes Läsionsgewebe, nichtdiagnostisch, mit definiertem positiven Reaktionsausfall |
| 5 | Positive Gewebskontrolle (intern) | Läsionsgewebe, diagnostisch, mit erwartetem definierten positiven Reaktionsausfall |
| 6 | Negative Gewebskontrolle (intern) | Läsionsgewebe, diagnostisch, mit erwartetem definierten negativen Reaktionsausfall |
| 7 | Negative Färbekontrolle | Läsionsgewebe, diagnostisch, mit definiertem positiven Reaktionsausfall, ohne Primärantikörper |
| 8 | Externe Gewebskontrolle (separat) | Als geeignet bekanntes Läsionsgewebe, nichtdiagnostisch, auf separatem Objektträger, mit definiertem positiven und/oder negativen Reaktionsausfall |
| 9 | Externe Gewebskontrolle (on-slide) | Als geeignet bekanntes Läsionsgewebe, nichtdiagnostisch, auf diagnostischem Objektträger, mit definiertem positiven und/oder negativen Reaktionsausfall |
Tabelle 15: Gewebetypen (OID: 1.2.276.0.76.5.????)
Färbeergebnis
| Code | Färbeergebnis | Bedeutung |
|---|---|---|
| 0 | negativ | keine diagnostische Färbung |
| 1 | fraglich positiv | unsichere diagnostische Färbung |
| 2 | positiv | diagnostische Färbung |
| 9 | nicht auswertbar | diagnostisch nicht verwertbar |
Tabelle 16: Färbeergebnis (OID: 1.2.276.0.76.5.????)
Fixierung und Einbettung
| Code | Fixierung | Bedeutung |
|---|---|---|
| 0 | unfixiert | frisches oder gefrorenes Gewebe |
| 1 | FFPE | formalifixiert, paraffineingebettet |
| 2 | andere | andere Fixations- und/oder Einbettungsverfahren |
Tabelle 17: Fixierung (OID: 1.2.276.0.76.5.????)
Bildanalyseprogramm
| Programmname | Hersteller | Verwendungszweck |
|---|---|---|
| ImmunoRation | http://imtmicroscope.uta.fi/immunoratio | Quantifizierung von Hormonrezeptorexpression und Ki-67-Expression |
| ImmunoMembrane | http://imtmicroscope.uta.fi/immunomembrane | Quantifizierung der Her-2-neu-Rezeptorexpression |
| ACIS III | DAKO | Antigenquantifizierung für pharmDX Kits |
| VIAS | Ventana/Roche | Quantifizierung des Ventana/Roche Breast Panel |
| u.s.w. |
Tabelle 18: Bildanalyseprogramme (OID: 1.2.276.0.76.5.????)
Score-Typ
| Code | Name | Bedeutung |
|---|---|---|
| 1 | Remmele-Stegner | Hormonrezeptorexpression |
| 2 | Allred | Hormonrezeptorexpression |
| 3 | Her-2-neu-Brust | Her-2-neu-Expression bei Mammakarzinom |
| 4 | Her-2-neu-Magen | Her-2-neu-Expression bei Magenkarzinom |
| u.s.w. |
Tabelle 19: Score-Typ (OID: 1.2.276.0.76.7.2.????)
FO: Scores und die Ergebnisse sollten gemäß Scores und Assessment DSTU übermittelt werden!
Score-Ergebnis
| Score-Code | Ergebnis | Bedeutung |
|---|---|---|
| 1 | 0 | endokrin nicht responsiv |
| 1 | 1 | endokrin fraglich responsiv |
| 1 | 2-12 | endokrin responsiv |
| 3 | 0 und 1+ | keine Überexpression |
| 3 | 2+ | fragliche Überexpression |
| 3 | 3+ | Überexpression |
| u.s.w. |
Tabelle 20: Score-Ergebnis (OID: 1.2.276.0.76.5.????)
Kommentar: Die mit ???? endenden OID müssen alle noch endgültig definiert werden. Können wir das tun?
Makroskopische Beschreibung
Intraoperativer Schnellschnitt
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.7 ???? | |
| General Description | intraoperativer Schnellschnitt | |
| LOINC Code | Opt. | Description |
| ???? | O | |
Für diese Section ist durch IHE APSR vorgeschlagen, "Intraoperative Observation" zu verwenden (OID 1.3.6.1.4.1.19376.1.8.1.2.2), die die intraoperative Diagnose für jede beurteilte Probe einschl. Proben-und Prozedurbeschreibung beschreibt. Ein maschinenlesbares Entry-Modul "Specimen Intraoperative Observation Entry", Template ID 1.3.6.1.4.1.19376.1.8.1.3.2 sowie ein Modul "Author of the section", Template ID 1.3.6.1.4.1.19376.1.8.1.4.2 innerhalb dieser Section ist vorgesehen. Für die Kodierung von 1.3.6.1.4.1.19376.1.8.1.3.2 ist LOINC vorgesehen, ein Ergebnis einer Anfrage am Regenstrief-Institut steht aber noch aus.
Dieser Vorschlag sollte in deutscher Übersetzung angepasst werden.
Außerdem sollte ein weiteres Entry-Modul für Prozessdaten generiert werden, das Probeneingang (Specimen->SpecimenProcessStep), Ende der Probenuntersuchung (documentationOfServiceEvent->Specimen), Ende der Befundung (Specimen->ObservationEventauthor.time) oder Übermittlung Befund, jeweils lt Anhang A, Punkt 76, erfasst und zusätzlich die Diagnosequalität nach Abschluss der Untersuchungen in eine der vier Kategorien einteilt: Richtig Positiv, Richtig Negativ, Falsch Positiv, Falsch Negativ hinsichtlich der fast ausschließlich vorliegenden Fragestellung "Malignität?", oder noch allgemeiner in "übereinstimmend mit Referenzdiagnose: ja, nein, nicht angebbar" als BL kodiert. Für diese Kategorien gibt es offensichtlich noch keine Kodierungen (außer UMLS und SNOMED CT, hier "Modifier mainly for procedure (qualifier value), Concept ID 106239005)??
Mikroskopische Beschreibung
Die Abschnitte zur werden einzeln gemäß nachfolgender Spezifikation dargestellt, d.h. die entsprechenden Abschnitte werden nicht ineinander verschachtelt.
Immunhistologie
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.7 ???? | |
| General Description | In diesem Abschnitt werden die Daten zur Immunhistologie übermittelt. | |
| LOINC Code | Opt. | Description |
| ???? | O | |
Sollte als ein möglicher Procedure step geführt werden.
I.d.R. werden diagnoserelevante immunhistologische Befunde in der mikroskopischen Beschreibung erwähnt. In den Anlagen ist ein Vorschlag für eine sowohl diagnostisch als auch methodisch wichtige detaillierte Einzelbeschreibung zahlreicher Aspekte der durchgeführten Untersuchungen aufgeführt. Diese sollten zum großen Teil aus Prozessdaten der Laborautomaten / des pathologiesystems beritgestellt werden.
Elektronenmikroskopie
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.7 ???? | |
| General Description | In diesem Abschnitt werden die Daten zur Elektronenmikroskopie übermittelt. | |
| LOINC Code | Opt. | Description |
| ???? | O | |
Sollte als ein möglicher Procedure step geführt werden.
I.d.R. werden diagnoserelevante elektronenmikroskopische Befunde in der mikroskopischen Beschreibung erwähnt.
Molekularpathologie
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.7 ???? | |
| General Description | In diesem Abschnitt werden die Daten zur Molekularpathologie übermittelt. | |
| LOINC Code | Opt. | Description |
| ???? | O | |
Sollte als ein möglicher Procedure step geführt werden.
I.d.R. werden diagnoserelevante molekularpathologische Befunde in der mikroskopischen Beschreibung erwähnt.
Bisher keine Vorarbeiten für entry bekannt, einzelne Ergebnisse (Befunde) in LOINC und IHE APSR zu kodieren.
Präparatradiographie
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.7 ???? | |
| General Description | In diesem Abschnitt werden die Daten zur Präparatradiographie übermittelt. | |
| LOINC Code | Opt. | Description |
| ???? | O | |
Sollte als ein möglicher Procedure step geführt werden.
Diagnoserelevante Befunde werden in der mikroskopischen Beschreibung erwähnt. Vorschläge für kodierte Form liegen in den Checklisten für Mammakarzinome vor.
Unterbeauftragung
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.7 ???? | |
| General Description | In diesem Abschnitt werden die Informationen über Unterbeauftragungen übermittelt. | |
| LOINC Code | Opt. | Description |
| ???? | O | |
Untersuchungen, die als Unterauftrag weitergegeben werden, müssen hier gekennzeichnet werden:
- welches Material
- welche Untersuchung
- an wen gesandt
Die Ergebnisse derartiger Untersuchungen werden in der Regel als Nachbericht mitgeteilt.
Diagnosen
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.7 ???? | |
| General Description | In diesem Abschnitt werden die Diagnosen übermittelt. | |
| LOINC Code | Opt. | Description |
| ???? | O | |
Die Diagnosen sind gemäß Diagnoseleitfaden zu übermitteln! Die Darstellung wird aus den codierten Informationen (sofern z.B. aus Cancer Check List vorhanden) abgeleitet.
Im Falle einer Tumordiagnose enthält die Diagnose die Cancer Check List, wenn vorhanden organspezifisch, als entry (s. Anlagen und IHE_PAT_Suppl._APSR_Rev.1.1)
Trotzdem sollte hier noch ein vollständiges Beispiel angeführt werden!
Zusammenfassung: ausführl. kritische gutachterliche Stellungnahme/Epikrise/Kommentar
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.7 ???? | |
| General Description | In diesem Abschnitt werden eine ausführliche kritische Gutachterliche Stellungnahme/Epikrise/Kommentar übermittelt. | |
| LOINC Code | Opt. | Description |
| ???? | O | |
genauer klären, wie dieser Abschnitt heißen soll bzw. was darin enthalten ist!
Die Epikrise ist bereits Bestandteil des VHitG-Arztbriefes.
Nach IHE_APSR ist sie Subsection der Diagnose.
Turmorformel
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.7 ???? | |
| General Description | In diesem Abschnitt werden der Tumorformel übermittelt. | |
| LOINC Code | Opt. | Description |
| ???? | O | |
Verschlüsselung / Stadium / spezielle Schlüssel
In der Regel ist es für die Mehrzahl der meldepflichtigen Tumordiagnosen notwendig, die sog. Tumorformel nach dem Diagnoseleitfaden zu verschlüsseln.
Diagnose(n) konsiliarischer Untersuchungen
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.7 ???? | |
| General Description | In diesem Abschnitt werden die Diagnosen des Konsiliarpartners übermittelt. | |
| LOINC Code | Opt. | Description |
| ???? | O | |
Hier erfolgt die Angabe des Konsilpartners und dessen Diagnose / Antwort auf die konsiliarische Fragestellung.
Weitergabemodus
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.7 ???? | |
| General Description | In diesem Abschnitt werden die Daten zum Weitergabemodus übermittelt. | |
| LOINC Code | Opt. | Description |
| ???? | O | |
tbd
Gruß
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.7 ???? | |
| General Description | In diesem Abschnitt wird der Gruß übermittelt. | |
| LOINC Code | Opt. | Description |
| ???? | O | |
Dies ist bereits Bestandteil des VHitG-Arztbriefes.
Anlagen
Immunhistochemische Färbungen
Die Informationen zu immunhistochemischen Färbungen bestehen aus folgenden Informationen:
| Bedeutung | Datentyp | OID |
|---|---|---|
| Antikörper (Kurzbezeichnung) | Code oder String?? | 2.16.840.1.113883.6.1 |
| Klon | String | 1.2.276.0.76.5.???? |
| Hersteller | Code oder String?? | 1.2.276.0.76.5.???? |
| Antikörperklasse | CD | ???? |
| Protokoll-ID | II | 1.2.276.0.76.5.???? |
| Färbeintensität | CD | 1.2.276.0.76.5.???? |
| Färbemuster | CD | 1.2.276.0.76.5.???? |
| Verteilungsmuster | CD | 1.2.276.0.76.5.???? |
| Anteil positiver Zellen | PQ | ?? |
| Gewebetyp | CD | 1.2.276.0.76.5.???? |
| Färbeergebnis | CD | 1.2.276.0.76.5.???? |
| Fixierung | CD | 1.2.840.10008.???? |
| Bildanalyseprogramm | String | 1.2.276.0.76.5.???? |
| Score-Typ | vgl. Scores & Assessment DSTU | |
| Score-Ergebnis | vgl. Scores & Assessment DSTU |
Tabelle 4: Färbungen
Text-Beispiel
Nachfolgend ein Beispiel in der Text-Darstellung:
| Anti- körper | Klon | Her- steller | AK- Klasse | Proto- koll- ID | Reak- tions-stärke | Färbe- muster | Vertei- lungs- muster | %pos. Zellen | Gewebe- typ | Färbe- er- gebnis | Fixie- rung | Bild- analyse | Score- Typ | Score-Ergebnis |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Ki67 | 30-9 | Ventana | 2 | xyz | stark | nukleär | diffus | 9 | Tumor | positiv | FFPE | Immuno- Ratio | ||
| Ki67 | 30-9 | Ventana | 2 | xyz | keine | keine | keine | 0 | neg.Färbe- kontrolle | negativ | FFPE | |||
| CK5/6 | D5/16B4 | DAKO | 1 | uvw | mittel | membran- st. komplett | basal | Tumor | positiv | FFPE | ||||
| ER/PR pharmDX | 1D5 / ER 2-123 | DAKO | 3 | abc | stark | nukleär | diffus | 87 | Tumor | positiv | FFPE | Immuno- Ratio | Rem- mele | endokrin responsiv |
| ER/PR pharmDX | 1D5 / ER 2-123 | DAKO | 3 | abc | stark | nukleär | diffus | 95 | ext. Positiv on-slide-kontrolle |
positiv | FFPE | Immuno- Ratio | ||
| ER/PR pharmDX | 1D5 / ER 2-123 | DAKO | 3 | abc | mittel | nukleär | fokal | 30 | int. Positiv kontrolle |
positiv | FFPE | Immuno- Ratio | ||
| ER/PR pharmDX | 1D5 / ER 2-123 | DAKO | 3 | abc | keine | keine | keine | 0 | neg.Färbe kontrolle |
negativ | FFPE | |||
| ER/PR pharmDX | PgR 1294 | DAKO | 3 | def | mittel | nukleär | diffus | 38 | Tumor | positiv | FFPE | Immuno- Ratio | Rem- mele | endokrin responsiv |
| ER/PR pharmDX | PgR 1294 | DAKO | 3 | def | keine | keine | keine | 0 | neg.Färbe kontrolle |
negativ | FFPE |
Abbildung in CDA
Kommentar: muss noch an Textbeispiel angepasst werden.
<section>
<!-- Darstellung als Tabelle -->
<text>
<tbody>
<tr>
<th>Antikörper</th>
<th>Klon</th>
<th>Hersteller</th>
<th>AK-Klasse</th>
<th>Protokoll-ID</th>
<th>Reaktionsstärke</th>
<th>Färbemuster</th>
<th>Verteilungsmuster</th>
<th>% pos. Zellen</th>
<th>Gewebetyp</th>
<th>Färbeergebnis</th>
<th>Fixierung</th>
</tr>
<tr>
<td><content ID="d1">IF Ep MNF116</content></td>
<td><content ID="d2">positiv</content></td>
<td><content ID="d3">stark</content></td>
<td><content ID="d4"> </content></td>
<td><content ID="d5">diffus</content></td>
<td><content ID="d6">Formalin</content></td>
<td><content ID="d7">Tumor isolierte Tumorzelle</content></td>
</tr>
...
</tbody>
</text>
<!-— erste Information -->
<entry typeCode="DRIV">
<observation classCode="OBS" moodCode="EVN">
<code code="????"
codeSystem="??????"
displayName="Antikörperfärbung Art (kurz)" />
<value xsi:type="CD" code="?????" codeSystem="????">
<originalText><reference value="#d1"/></originalText>
</value>
</observation>
</entry>
<!-— zweite Information -->
<entry typeCode="DRIV">
<observation>
<code code="????"
codeSystem="??????"
displayName="Antikörperfärbung Reaktion" />
<value xsi:type="CD" code="2" codeSystem="????">
<originalText><reference value="#d2"/></originalText>
</value>
</observation>
</entry>
<!-— dritte Information -->
<entry typeCode="DRIV">
<observation classCode="OBS" moodCode="EVN">
<code code="xxxx"
codeSystem="a.b.c.dx.y.z"
displayName="Antikörperfärbung Reaktionsstärke" />
<value xsi:type="CD" code="3" codeSystem="????">
<originalText><reference value="#d3"/></originalText>
</value>
</observation>
</entry>
<!-— vierte Information -->
<entry typeCode="DRIV">
<observation classCode="OBS" moodCode="EVN">
<code code="xxxx"
codeSystem="a.b.c.dx.y.z"
displayName="Antikörperfärbung Prozent" />
<value xsi:type="CD" code="3" codeSystem="????">
<originalText><reference value="#d4"/></originalText>
</value>
</observation>
</entry>
<!-— fünfte Information -->
<entry typeCode="DRIV">
<observation classCode="OBS" moodCode="EVN">
<code code="xxxx"
codeSystem="a.b.c.dx.y.z"
displayName="Verteilung" />
<value xsi:type="CD" code="3" codeSystem="????">
<originalText><reference value="#d5"/></originalText>
</value>
</observation>
</entry>
<!-— sechste Information -->
<entry typeCode="DRIV">
<observation classCode="OBS" moodCode="EVN">
<code code="xxxx"
codeSystem="a.b.c.dx.y.z"
displayName="Fixierung" />
<value xsi:type="CD" code="3" codeSystem="????">
<originalText><reference value="#d6"/></originalText>
</value>
</observation>
</entry>
<!-— siebte Information -->
<entry typeCode="DRIV">
<observation classCode="OBS" moodCode="EVN">
<code code="xxxx"
codeSystem="a.b.c.dx.y.z"
displayName="Gewebe" />
<value xsi:type="CD" code="3" codeSystem="????">
<originalText><reference value="#d7"/></originalText>
</value>
</observation>
</entry>
...
</section>
Digitale Bilder
Makroskopische sowie mikroskopische Bilder und Virtual Slides müssen eingebunden werden können. Lösungen über DICOM Supplement 122: Specimen Module and Revised Pathology SOP Classes
Attribut-Wert-Paare
Die Attribut-Wert-Paare werden textuell aus den codierten Informationen abgeleitet (derived).
Hierzu gehören auch die Cancer Checklists.
Die zu verwendenden Vokabularien sind im Anhang detailliert aufgelistet.
Die Informationen werden als Attribut-Wert-Paare in Form einer Tabelle dargestellt, die wie folgt aussieht.
| Entnahme | Resektat |
|---|---|
| Kalk histologisch | Ja |
| Kalk (mm) | 0,2 |
Oder in XML:
<section>
<!-- Darstellung als Tabelle -->
<text>
<tbody>
<tr>
<td><content ID="d1">Entnahme</content></td>
<td>Resektat</td>
</tr>
<tr>
<td><content ID="d2">Kalk Histologisch</content></td>
<td>Ja</td>
</tr>
<tr>
<td><content ID="d3">Kalk (mm)</content></td>
<td>0,2</td>
</tr>
...
</tbody>
</text>
<!-— erste Information -->
<entry typeCode="DRIV">
<observation classCode="OBS" moodCode="EVN">
<code code"Mamma.Entnahme" codeSystem="??????" />
<value xsi:type="CD" code="Resektat" codeSystem="????">
<originalText><reference value="#d1"/></originalText>
</value>
</observation>
</entry>
<!-— zweite Information -->
<entry typeCode="DRIV">
<observation>
<code code="Mamma.Kalk Histologisch" codeSystem="??????" />
<value xsi:type="BL" code="true">
<originalText><reference value="#d2"/></originalText>
</value>
</observation>
</entry>
<!-— dritte Information -->
<entry typeCode="DRIV">
<observation classCode="OBS" moodCode="EVN">
<code code="Mamma.Kalk"
codeSystem="a.b.c.dx.y.z"
displayName="Kalk" />
<value xsi:type="PQ" value="0,2" unit="mm" >
<originalText><reference value="#d3"/></originalText>
</value>
</observation>
</entry>
<!—weitere Information -->
...
</section>
Beispiele für Befunde
Zu Beginn ein relativ einfaches und kurzes Beispiel:
Makroskopische Beurteilung:
Zusammen pampelmusenkerngroße (Durchmesser 12 mm) membranöse festelastische grauweißliche Gewebsstücke.
Mikroskopische Beurteilung:
Partiell durch ein sehr schmales, nicht verhorntes Plattenepithel ausgekleideter Balganteil einer odontogenen Zyste mit einzelnen Malassezschen Epithelnestern und herdbetonter sehr schütterer rundzelliger entzündlicher subepithelialer Infiltration. Eingesprengt wenig Hartmaterial.
Diagnose:
Follikuläre Zyste. Kein Anhalt für Malignität oder Spezifität am vorliegenden Material.
Unterschrift
Makroskopische Beurteilung:
Mehrere (gemäß klinischer Angabe fünf) zusammen 51 mm lange weiche bis mittelfeste teils grauweißliche, teils graugelbliche Punktionszylinder von max. Bleistiftminenstärke.
Mikroskopische Beurteilung: (HE, CK 5/14, CK 7):
Alle gewonnenen Punktionszylinder wurden vollständig gebettet und mit 13 Schnittstufen untersucht. Sie bestehen aus lipomatös und fibrolipomatös transformiertem Brustdrüsengewebe und lassen in mindestens sechs Punktionszylinderteilstücken neben atrophischen Drüsenlobuli und ektatischen Milchgangsanschnitten sowie kleineren Adenoseherden (mikrozystische und blunt-duct-Adenosen) in wechselnder Dichte in ein hyalinelastotisch transformiertes Stroma eingebettete unterschiedlich großkalibrige Tubuli erkennen, diese vielfach mit Abknickungen, die ausgekleidet werden von einem einreihigen, überwiegend gering, örtlich mäßig atypischen flach kubischen Epithel ohne nennenswerte mitotische Aktivität und fehlende basale Myoepithellage in der CK 5/14-Reaktion, vereinbar mit Tumorinfiltraten eines tubulären Mammakarzinoms (Malignitätsgrad 1 nach Elston und Ellis; 1+2+1); vgl. auch EH-Nr. 7082/12.
Diagnose: Tubuläres Karzinom der Mamma.
Klassifikation nach NHSBSP: B 5b
Zum Ergebnis der noch ausstehenden Hormonrezeptorbestimmung und des HER-2/neu- Status erfolgt ein Nachbericht. Telefonische Vorabinformation am 31.05.2012.
Unterschrift
1. Nachbericht:
Ergebnis der immunhistochemischen Hormonrezeptorbestimmung:
Östrogenrezeptoren: über 80 % positiver Zellkerne, vorherrschende Färbeintensität: stark, IRS 12 nach Remmele und Stegner.
Progesteronrezeptoren: über 80 % positiver Zellkerne, vorherrschende Färbeintensität: stark, IRS 12 nach Remmele und Stegner.
Der Tumor ist endokrin-responsiv.
Ergebnis der in-situ-Hybridisierung (BDISH) des HER2-neu-Gens:
Nach einer in-situ-Hybridisierung des Genlocus c-erb-B2 und des Zentromer von Chromosom 17 wurden die ISH-Signale von 30 Tumorzellen ausgezählt. Die untersuchten invasiven Tumorzellen zeigen ein mittleres Verhältnis 1,19 (Gen/Zentromer). Es liegt also keine Amplifikation des HER2-neu Lokus vor (ZytoDot 2C SPEC Her- 2/CEN17 Probe Kit).
Unterschrift
Makroskopische Beurteilung:
1. Backpflaumengroßes (40 x 20 x 15 mm messendes) Fettgewebsstück, darin
eingeschlossen drei, max. gewürzkorngroße (größter Durchmesser 6 mm) mittelfeste
grauweißliche Gewebsknoten.
2. Auf Styroporplatte nadelfixiertes, zweifach fadenmarkiertes fettgewebsreiches
Mammaresektat aus dem zentralen Drüsenkörper von 119 g in einer Ausdehnung
von 6,5 x 7 x 4 cm mit zentral aufsitzender, 4 cm durchmessender Mamille/Areole.
Mamille und unmittelbar retromamilläres Drüsenkörpergewebe in einer Ausdehnung
von 12 mm derb knotig grauweiß induriert. Auf der Mamille selbst aufgelagert
eine kleine Kruste.
Das Resektat wird von lateral beginnend in neun Scheiben lamelliert. Weitere
verdächtige Knotenbildungen kommen nicht zur Darstellung.
Sicherheitszonen der beschriebenen Knotenbildungen zu allen Resektionsrändern
mindestens 10 mm bzw. deutlich darüber.
Schnellschnittdiagnose:
1. Alle drei Sentinellymphknoten im Schnellschnitt metastasenfrei.
2. Karzinom unmittelbar retromamillär, Sicherheitszonen nach allen Seiten
mindestens 10 mm bzw. deutlich darüber.
Telefonische Befundübermittlung am 15.06.2012, 11.59 Uhr an Herrn CA Dr. XY.
Mikroskopische Beurteilung:
1. Alle drei Sentinellymphknoten, die vollständig gebettet und in Stufenserienschnitten
mit jeweils 12 Schnittstufen aufgearbeitet wurden, sind metastasenfrei.
Man erkennt kleinherdige lipomatöse Transformationen und narbige Fibrosierungen.
Umgebendes Fettgewebe unauffällig.
2. Das Mammaresektat wurde unter Berücksichtigung der Topografie und Markierungen mit 13 Paraffinblöcken mit jeweils Schnittstufen untersucht. In der Haut der Mamillenregion bzw. unmittelbar retromamillär unter Einbeziehung der lokalisationstypisch größeren Ductus und Sinus lacteripheri Tumorgewebe des in der Stanzbiopsie vordiagnostizierten invasiven Mammakarzinoms (vgl. EH-Nr. 7226/12), jetzt als gemischt invasiv duktales und lobuläres Mammakarzinom zu klassifizieren (Malignitätsgrad 2 nach Elston und Ellis; 3+2+1) mit vordergründig dissolut einzelzelligem Wachstumsmuster mit Ausbildung sog. indian files und Targetstrukturen um unbeeinträchtigte Milchgänge, aber auch tubulären Differenzierungen und kleiner intraduktaler Komponente, die an umschriebener Stelle den invasiven Karzinomanteil nach dorsal um 1 mm überschreitet. Es besteht eine wechselnd stark ausgeprägte Stromasklerose mit örtlich schütter rundzellig entzündlicher Stromareaktion und man sieht in einem der Schnittpräparate eine umschriebene, herdförmig eingeblutete narbige Fibrose mit reaktiven Veränderungen der bedeckenden Epidermis bei Zustand nach vorausgegangener Punchbiopsie. Karzinomentfernung im Gesunden, Sicherheitszonen nach allen Seiten jeweils deutlich über 10 mm. Breit anhängender Saum tumorfreien Brustdrüsengewebes betont lipomatös transformiert ohne sonstigen nennenswerten pathologischen Befund.
Zusammenfassung:
Ungewöhnlich oberflächlich retromamillär bzw. in der Haut der Mamillenregion rechts gelegenes gemischt invasiv duktales und lobuläres Mammakarzinom. Karzinomentfernung im Gesunden (Sicherheitszonen nach allen Seiten über 10 mm). Drei metastasenfreie Sentinellymphknoten.
Unterbeauftragte Untersuchung:
Unfixiertes Tumorgewebe wurde zur uPA/PAI-1-Bestimmung an das Institut für Pathologie des UKD gesandt. Über das Ergebnis wird nachberichtet.
Tumorklassifikation:
TNM (UICC, 7. Auflage):
pT1c pN0 (0/3sn), R0, L0, V0
Grading:
G 2
ICD-O-3:
C 50.0, M 8522/3
Unterschrift
1. Nachbericht:
Ergebnis der uPA/PAI-1-Analyse:
Methodik: Am unfixierten Gewebe des invasiven Mammakarzinoms wurde nach Kryokonservierung mit dem FEMTELLE® ELISA-Test die Aktivität des Plasminogenaktivators vom Urokinasetyp (uPA) und des Plasminogen-Inhibitors (PAI-1) bestimmt (Institut für Pathologie des UKD, Prof. Dr. XYZ).
Untersuchungsergebnis:
uPA: 6,27 ng/mg Protein
PAI-1: 12,4 ng/mg Protein
Bewertung:
Bei diesem Ergebnis kann bei nodal-negativen Mammakarzinom, Malignitätsgrad 2 nach Elston und Ellis entsprechend der Interdisziplinären S3-Leitlinie für die Diagnostik, Therapie und Nachsorge des Mammakarzinoms von einem hohen Rezidivrisiko ausgegangen werden.
Unterschrift
PathoBerichtText
Wiederholung der Ki-67-Färbung von der 2. Fraktion (rechts).
Beurteilung
1. Isolierte Tumorzellen in einem funktionsgesteigerten Lymphknoten (linke Axilla Sentinellymphknoten 544cps).
2. Teils glanduläres, teils solides, invasives ductales Adenocarcinom der Brustdrüse, geringe nukleäre Atypien sowie ductales Carcinoma in situ mit geringen Atypien, DCIS I und Mikrokalk bis 0,2mm (rechte Mamma oben zwischen den Quadranten, Resektat); immunhistologisch fokal starke Expression des Östrogenrezeptors in etwa 60% der Tumorzellen.
Score nach Elston und Ellis: 4.
Immunreaktiver Score: Östrogenrezeptor 9, Progesteronrezeptor 0.
Onkoprotein C-erbB-2 Index: 0
Größter Durchmesser des invasiven Carcinoms etwa 9mm, das invasive Carcinom reicht zumindest an die craniale Abtragungsebene im peripheren (cranialen) Pol. Durchmesser der in situ Komponente etwa 35mm, Abstand von der nächstgelegenen (posterioren) Abtragungsebene etwa 0,4mm, die in situ Komponente breitet sich allerdings ebenfalls in den peripheren (cranialen) Pol aus und erreicht damit dort mindestens die Abtragungsebene.
3. Weitgehend solides, invasives ductales Adenocarcinom der Brustdrüse, mittelgradige, herdförmig schwere nukleäre Atypien, etwa in gleicher Größe ductales Carcinoma in situ mit schweren Atypien, DCIS III sowie Nekrosen und Verkalkungen bis 4mm (Resektat linke Mamma oben außen); immunhistologisch starke Expression des Östrogenrezeptors und des Progesteronrezeptors jeweils in etwa 85% der Tumorzellen. Immunreaktiver Score: Östrogenrezeptor 12, Progesteronrezeptor 12. Kleine Wachstumsfraktion (Ki-67 um 10 %). Onkoprotein C-erbB-2 (Her-2-Neu-Index: 0). Größter Durchmesser der invasiven Komponente und der in-situ-Komponente jeweils etwa 23mm. Abstand der invasiven Komponente von der nächstgelegenen Abtragungsebene (posterior) 3,2mm, Abstand von anterior 12mm, von cranial 20mm, von caudal 23mm, von medial 30mm, von lateral 13mm. Die in-situ-Komponente breitet sich zumindest bis an die posteriore Abtragungsebene aus.
4. Neun tumorfreie Lymphknoten (linke Axilla).
5. Tumorfreies Fettgewebe (linke Mamma, Nachresektat lateral).
6. Überwiegend tumorfreies Fettgewebe mit kleinen Anteilen von tumorfreiem Brustdrüsengewebe (linke Mamma, Nachresektat mamillenwärts).
7. Überwiegend tumorfreies Fettgewebe mit kleinen Anteilen von tumorfreiem Brustdrüsengewebe (linke Mamma, Nachresektat cranial).
Auch nach Wiederholung lässt sich die Wachstumsfraktion in dem kleinen Tumor auf der rechten Seite nicht darstellen, vermutlich wurde das Antigen durch die schwere thermische Schädigung zerstört.
<StructuredBody>
<component>
<section>
<code code="PathoBerichtText" codeSystem="1.2.276.0.76.5.??????"/>
<title></title>
<text>
Wiederholung der Ki-67-Färbung von der 2. Fraktion (rechts).
</text>
</section>
</component>
<component>
<section>
<code code="PathoBerichtText" codeSystem="1.2.276.0.76.5.??????"/>
<title>Beurteilung</title>
<text>
1. Isolierte Tumorzellen in einem funktionsgesteigerten Lymphknoten
(linke Axilla Sentinellymphknoten 544cps).<br>
<br>
2. Teils glanduläres, teils solides, invasives ductales Adenocarcinom der Brustdrüse,
geringe nukleäre Atypien sowie ductales Carcinoma in situ mit geringen Atypien, DCIS I
und Mikrokalk bis 0,2mm (rechte Mamma oben zwischen den Quadranten, Resektat);
immunhistologisch fokal starke Expression des Östrogenrezeptors in etwa 60% der
Tumorzellen. <br>
Score nach Elston und Ellis: 4.<br>
Immunreaktiver Score: Östrogenrezeptor 9, Progesteronrezeptor 0.<br>
Onkoprotein C-erbB-2 Index: 0<br>Größter Durchmesser des invasiven Carcinoms etwa 9mm,
das invasive Carcinom reicht zumindest an die craniale Abtragungsebene im peripheren
(cranialen) Pol. Durchmesser der in situ Komponente etwa 35mm, Abstand von der
nächstgelegenen (posterioren) Abtragungsebene etwa 0,4mm, die in situ Komponente breitet
sich allerdings ebenfalls in den peripheren (cranialen) Pol aus und erreicht damit dort
mindestens die Abtragungsebene.<br>
<br>
3. Weitgehend solides, invasives ductales Adenocarcinom der Brustdrüse, mittelgradige,
herdförmig schwere nukleäre Atypien, etwa in gleicher Größe ductales Carcinoma in situ
mit schweren Atypien, DCIS III sowie Nekrosen und Verkalkungen bis 4mm (Resektat linke
Mamma oben außen); immunhistologisch starke Expression des Östrogenrezeptors und des
Progesteronrezeptors jeweils in etwa 85% der Tumorzellen.<br>
Immunreaktiver Score: Östrogenrezeptor 12, Progesteronrezeptor 12. <br>
Kleine Wachstumsfraktion (Ki-67 um 10 %). <br>
Onkoprotein C-erbB-2 (Her-2-Neu-Index: 0). Größter Durchmesser der invasiven Komponente
und der in-situ-Komponente jeweils etwa 23mm. Abstand der invasiven Komponente von der
nächstgelegenen Abtragungsebene (posterior) 3,2mm, Abstand von anterior 12mm, von cranial
20mm, von caudal 23mm, von medial 30mm, von lateral 13mm. Die in-situ-Komponente breitet
sich zumindest bis an die posteriore Abtragungsebene aus. <br>
<br>
4. Neun tumorfreie Lymphknoten (linke Axilla).<br>
<br>
5. Tumorfreies Fettgewebe (linke Mamma, Nachresektat lateral).<br>
<br>
6. Überwiegend tumorfreies Fettgewebe mit kleinen Anteilen von tumorfreiem
Brustdrüsengewebe (linke Mamma, Nachresektat mamillenwärts).<br>
<br>
7. Überwiegend tumorfreies Fettgewebe mit kleinen Anteilen von tumorfreiem
Brustdrüsengewebe (linke Mamma, Nachresektat cranial).
</text>
</section>
</component>
<component>
<section>
<code code="PathoBerichtText" codeSystem="1.2.276.0.76.5.??????"/>
<title></title>
<text>
Auch nach Wiederholung lässt sich die Wachstumsfraktion in dem kleinen Tumor auf der
rechten Seite nicht darstellen, vermutlich wurde das Antigen durch die schwere thermische
Schädigung zerstört.
</text>
</section>
</component>
</StructuredBody>
Vokabeldomänen
Einleitung
Dieser Abschnitt dient der Trennung von verwendeten Codes und der normativen Spezifikation. Damit lassen sich die Codes aktualisieren, ohne dass die Spezifikation überarbeitet werden muss.
Dieser Abschnitt ist deshalb nur informativ. Die jeweils aktuellen Codes sind deshalb zu erfragen.
Überblick über die Codierschemata
| Vokabeldomäne/ Codiersystem | OID | Kurzbezeichnung | Diagnosen | Lokalisationen |
|---|---|---|---|---|
| ICD10GM | ||||
| ICD-10 GM Version 2013 | ||||
| ICD-10 GM Version 2012 | 1.2.276.0.76.5.??? | icd10gm2012 | x | |
| ICD-10 GM Version 2011 | 1.2.276.0.76.5.??? | icd10gm2011 | x | |
| ICD-10 GM Version 2010 | 1.2.276.0.76.5.384 | icd10gm2010 | x | |
| ICD-10 GM Version 2009 | 1.2.276.0.76.5.356 | icd10gm2009 | x | |
| ICD-10 GM Version 2008 | 1.2.276.0.76.5.330 | icd10gm2008 | x | |
| ICD-10 GM Version 2007 | 1.2.276.0.76.5.318 | icd10gm2007 | x | |
| ICD-10 GM Version 2006 | 1.2.276.0.76.5.311 | icd10gm2006 | x | |
| ICD-O | ||||
| ICD-O-3 | icd-o-3 | |||
| ICD-O-DA-1978 | ||||
| ICD-O-DA-2002 | ||||
| TNM | ||||
| TNM: C-Faktor | 1.2.276.0.76.5.341 | c-faktor-tumor | ||
| TNM-Qualifier | 1.2.276.0.76.5.340 | tnm-qualifier | ||
| TNM: Metastasen | 1.2.276.0.76.5.339 | metastasen | ||
| TNM: Nodus | 1.2.276.0.76.5.338 | nodus-tnm | ||
| TNM: Ausdehnung | 1.2.276.0.76.5.337 | ausdehnung-tnm | ||
| TNM: Differenzierung | 1.2.276.0.76.5.336 | diff-grading-tumor | ||
| TNM: Dignität | 1.2.276.0.76.5.335 | dignitaet-tumor | ||
| TNM: Tumordiagnosen | 1.2.276.0.76.5.334 | tumordiagnosen | ||
| Alpha-ID | ||||
| Alpha-ID 2013 | ||||
| Alpha-ID 2012 | 1.2.276.0.76.5.??? | alphaid2012 | x | |
| Alpha-ID 2011 | 1.2.276.0.76.5.??? | alphaid2011 | x | |
| Alpha-ID 2010 | 1.2.276.0.76.5.383 | alphaid2010 | x | |
| Alpha-ID 2009 | 1.2.276.0.76.5.355 | alphaid2009 | x | |
| Alpha-ID 2008 | 1.2.276.0.76.5.329 | alphaid2008 | x | |
| Alpha-ID 2007 | 1.2.276.0.76.5.316 | alphaid2007 | x | |
| Alpha-ID 2006 | 1.2.276.0.76.5.309 | alphaid2006 | x | |
| MeSH | ||||
| MeSH | 2.16.840.1.113883.6.177.5 | MSHGER | x | x |
| Kodiersysteme | ||||
| Snomed CT | 2.16.840.1.113883.6.96 | SNOMED CT | x | x |
| ID Macs | 1.2.276.0.76.5.305 | id_macs | x | x |
| LOINC | 2.16.840.1.113883.6.1 | loinc | x | |
| .. | ||||
| 1.2.276.0.76.5.342 | Typisierung-diagnose | x | x |
Tabelle 5: Codierschemata
Diese Tabelle sollte ausgelagert werden, da sie sonst mehrfach gepflegt werden muss.
generische Codes für Attribut-Wert-Paare
Befundinterpretation im Kontext
| Code | Codename | Bedeutung |
|---|---|---|
| negativ | nicht zutreffend (z.B.für Malignität) | |
| positiv | zutreffend (z.B. für Malignität) | |
| zweifelhaft | nicht sicher zutreffend (z.B. für Malignität) |
Tabelle 19: Befundinterpretation im Kontext mit Fragestellung (OID 1.2.276.0.76.5.??????)
Hier stellt sich die Frage, ob bestimmte Attribute nicht auch über boolesche Werte abgebildet werden können?
z.B. in folgendem Beispiel
| Code | Codename | ja | nein | unsicher | nicht bestimmbar | keine Aussage |
|---|---|---|---|---|---|---|
| Übereinstimmung mit klinischer Fragestellung | x | x | x | x | ||
| Übereinstimmung mit Referenzdiagnose | x | x | x | x | ||
| etc. |
Tabelle 20: Befundinterpretation im Kontext mit Fragestellung (OID 1.2.276.0.76.5.??????)
Codes aus IHE Anatomy Pathology Report
Codes für Specimen Types
nach IHE (IHE APSR Trial Implementation, March 31, 2011) ist ein Specimen collection procedure generic template das Elterntemplate für jedes organspezifische Template. Jedes organspezifische Template hat Vokabularbegrenzungen, die für das jeweilige Organ spezifisch sind:
Ein Value Set ist gebunden an ein Prozedur Code-Element, das die verschiednen Prozeduren, die an diesem Organ möglich sind, auflistet.
Ein zweiter Value Set ist gebunden and das Prozedut-Target-Site Code-Element, das die möglichen präzisen Lokalisationen an diesem spezifischen Organ auflistet.
z.B.
| (PathLex)Code | Codename | Bedeutung |
|---|---|---|
| 2257 | Breast-Specimen-Specimen collection procedure | Excision with wire-guided localization |
| 2256 | Breast-Specimen-Specimen collection procedure | Excision without wire-guided localization |
| 662 | Breast-Specimen-Specimen collection procedure | Total mastectomy (including nipple and skin) |
| 666 | Breast-Specimen-Target site | Lower inner quadrant |
| 663 | Breast-Specimen-Target site | Upper outer quadrant |
Tabelle 24: HL7 Table 0487 resp. 0700 (OID 1.2.276.0.76.5.??????)
Codes für Specimen Reject Reason
| Code | Codename | Bedeutung |
|---|---|---|
| EX | expired | verfallen |
| QS | quantity not sufficient | Menge nicht ausreichend |
| RB | broken container | Einsendegefäß zerbrochen |
| RE | missing collection date | fehlende Angabe zum Entnahmedatum |
| R | missing patient ID | fehlender Patientencode |
| RE | missing patient name | fehlender Patientenname |
| etc. |
Tabelle 25: HL7 Table 0490 (OID 1.2.276.0.76.5.??????)
LOINC Codes
| LOINC Code | LOINC Code Name |
|---|---|
| 22637-3 | Path report.final diagnosis |
| 33746-9 | Pathologic findings |
| 22636-5 | Path report.relevant Hx |
| 22633-2 | Path report.site of origin |
| 22634-0 | Path report.gross description |
| 22635-7 | Path report.microscopic observation |
| 22638-1 | Path report.comments |
| 22639-9 | Path report.supplemental reports |
Tabelle 26: LOINC Codes (OID 2.16.840.1.113883.6.1)
Cancer Check Lists
| OID | Name |
|---|---|
| 1.3.6.1.4.1.19376.1.8.1.1.1 | Generic APSR |
| 1.3.6.1.4.1.19376.1.8.1.1.2.1 | Breast APSR |
| 1.3.6.1.4.1.19376.1.8.1.1.2.2 | Colonic APSR |
| 1.3.6.1.4.1.19376.1.8.1.1.2.3 | Prostate APSR |
| 1.3.6.1.4.1.19376.1.8.1.1.2.x | etc. |
Tabelle 27: Generische und Organspezifische Cancer Check Lists (OID 1.3.6.1.4.1.19376.1.8.1.1.2)
Generische Cancer Check List und Value set (ohne Organspezifität)
zum Teil werden die Sachverhalte auch vom Diagnoseleitfaden abgebildet (z.B. TNM). Doppelungen zulassen??
| Name | IHE_PAT_elementTemplates _Element_template_ID | IHE_PAT_Element_name | ValueSetID |
|---|---|---|---|
| Material | 1.3.6.1.4.1.19376.1.8.1.4.1 | Generic-Specimen Collection Procedure | 1.3.6.1.4.1.19376.1.8.5.371 |
| RR_Infiltration_INV | 1.3.6.1.4.1.19376.1.8.1.4.140 | Generic-Infiltrating malignant neoplasm-Margins involvement | BL |
| RR_Infiltration_CIS | 1.3.6.1.4.1.19376.1.8.1.4.141 | Generic-In situ neoplasm-Margins involvement | BL |
| RR_Infiltration | 1.3.6.1.4.1.19376.1.8.1.4.142 | Generic-Lesion-Margins involvement | BL |
| Fokalität_INV | 1.3.6.1.4.1.19376.1.8.1.4.143 | Generic-Infiltrating malignant neoplasm-Lesion focality | 1.3.6.1.4.1.19376.1.8.5.5 |
| TNM-Deskriptoren | 1.3.6.1.4.1.19376.1.8.1.4.144 | Generic-Infiltrating malignant neoplasm-TNM Descriptors | 1.3.6.1.4.1.19376.1.8.5.6 |
| Abstand_Nächster_RR_INV | 1.3.6.1.4.1.19376.1.8.1.4.145 | Generic-Infiltrating malignant neoplasm-Distance of lesion from closest uninvolved margin | PQ |
| Abstand_Nächster_RR_CIS | 1.3.6.1.4.1.19376.1.8.1.4.146 | Generic-In situ neoplasm-Distance of lesion from closest uninvolved margin | PQ |
| Lymphgefäßinvasion | 1.3.6.1.4.1.19376.1.8.1.4.147 | Generic-Infiltrating malignant neoplasm-Lymph-vascular invasion | BL |
| Lymphknotensampling | 1.3.6.1.4.1.19376.1.8.1.4.148 | Generic-Infiltrating malignant neoplasm-Lymph node sampling | BL |
| Ausdehnung | 1.3.6.1.4.1.19376.1.8.1.4.149 | Generic-Infiltrating malignant neoplasm-Extent | 1.3.6.1.4.1.19376.1.8.5.328 |
| Histol_Grad_2Stufig | 1.3.6.1.4.1.19376.1.8.1.4.150 | Generic-Infiltrating malignant neoplasm-Histologic grade (Two-Tier Grading System) | 1.3.6.1.4.1.19376.1.8.5.245 |
| Histol_Grad_WHO | 1.3.6.1.4.1.19376.1.8.1.4.151 | Generic-Infiltrating malignant neoplasm-Histologic grade (WHO) | 1.3.6.1.4.1.19376.1.8.5.246 |
| Perineuralscheideninvasion | 1.3.6.1.4.1.19376.1.8.1.4.152 | Generic-Infiltrating malignant neoplasm-Perineural invasion | BL |
| Lokalisation_RR | 1.3.6.1.4.1.19376.1.8.1.4.153 | Generic-Infiltrating malignant neoplasm-Margin site | 1.3.6.1.4.1.19376.1.8.5.192 |
| Lokalisation_Läsion_INV | 1.3.6.1.4.1.19376.1.8.1.4.154 | Generic-Infiltrating malignant neoplasm-Lesion site | 1.3.6.1.4.1.19376.1.8.5.329 |
| Lokalisation_Lymphknoten | 1.3.6.1.4.1.19376.1.8.1.4.155 | Generic-Infiltrating malignant neoplasm-Lymph node site | 1.3.6.1.4.1.19376.1.8.5.330 |
| Lymphknoten_befallen | 1.3.6.1.4.1.19376.1.8.1.4.156 | Generic-Infiltrating malignant neoplasm-Number of lymph nodes involved | INT |
| Lymphknoten_untersucht | 1.3.6.1.4.1.19376.1.8.1.4.157 | Generic-Infiltrating malignant neoplasm-Number of lymph nodes examined | INT |
| pM | 1.3.6.1.4.1.19376.1.8.1.4.158 | Generic-Infiltrating malignant neoplasm-pM | 1.3.6.1.4.1.19376.1.8.5.9 |
| pN | 1.3.6.1.4.1.19376.1.8.1.4.159 | Generic-Infiltrating malignant neoplasm-pN | 1.3.6.1.4.1.19376.1.8.5.8 |
| Probengewicht_INV | 1.3.6.1.4.1.19376.1.8.1.4.160 | Generic-Infiltrating malignant neoplasm-Specimen weight | PQ |
| pT | 1.3.6.1.4.1.19376.1.8.1.4.161 | Generic-Infiltrating malignant neoplasm-pT | 1.3.6.1.4.1.19376.1.8.5.7 |
| Behandlungseffekt | 1.3.6.1.4.1.19376.1.8.1.4.162 | Generic-Infiltrating malignant neoplasm-Treatment effect | 1.3.6.1.4.1.19376.1.8.5.10 |
| Läsionsgröße_Zusatzdimension | 1.3.6.1.4.1.19376.1.8.1.4.163 | Generic-Infiltrating malignant neoplasm-Lesion size, additional dimension | PQ |
| Läsionsgröße_größteDimension | 1.3.6.1.4.1.19376.1.8.1.4.164 | Generic-Infiltrating malignant neoplasm-Lesion size, largest dimension | PQ |
| Probengröße_Zusatzdimension_INV | 1.3.6.1.4.1.19376.1.8.1.4.165 | Generic-Infiltrating malignant neoplasm-Specimen size, additional dimension | PQ |
| Probengröße_größteDimension_INV | 1.3.6.1.4.1.19376.1.8.1.4.166 | Generic-Infiltrating malignant neoplasm-Specimen size, largest dimension | PQ |
| HistologischerTyp_INV | 1.3.6.1.4.1.19376.1.8.1.4.167 | Generic-Infiltrating malignant neoplasm-Histologic type | 1.3.6.1.4.1.19376.1.8.5.331 |
| MakroskopischerTyp_INV | 1.3.6.1.4.1.19376.1.8.1.4.168 | Generic-Infiltrating malignant neoplasm-Macroscopic type | 1.3.6.1.4.1.19376.1.8.5.187 |
| Lokalisation_Läsion_CIS | 1.3.6.1.4.1.19376.1.8.1.4.169 | Generic-In situ neoplasm-Lesion site | 1.3.6.1.4.1.19376.1.8.5.184 |
| HistologischerTyp_CIS | 1.3.6.1.4.1.19376.1.8.1.4.170 | Generic-In situ neoplasm-Histologic type | 1.3.6.1.4.1.19376.1.8.5.183 |
| Fokalität | 1.3.6.1.4.1.19376.1.8.1.4.171 | Generic-Lesion-Lesion focality | 1.3.6.1.4.1.19376.1.8.5.3 |
| Lokalisation | 1.3.6.1.4.1.19376.1.8.1.4.172 | Generic-Lesion-Lesion site | 1.3.6.1.4.1.19376.1.8.5.332 |
| HistologischerTyp | 1.3.6.1.4.1.19376.1.8.1.4.173 | Generic-Lesion-Histologic type | 1.3.6.1.4.1.19376.1.8.5.333 |
| Probenintegrität | 1.3.6.1.4.1.19376.1.8.1.4.174 | Generic-Specimen-Specimen integrity | 1.3.6.1.4.1.19376.1.8.5.2 |
Tabelle 28: Codeliste für Generische Cancer Check List (ohne Organspezifität)(OID 1.3.6.1.4.1.19376.1.8.1.1.1)
| ValueSetID | PathLex_Code | PathLex_Value |
|---|---|---|
| 1.3.6.1.4.1.19376.1.8.5.371 | ?? | ?? |
| 1.3.6.1.4.1.19376.1.8.5.5 | 801 | Single focus (Solitary - Unifocal) |
| 1.3.6.1.4.1.19376.1.8.5.5 | 802 | Multiple foci (Multifocal - (Ipsilateral – Bilateral))(specify location) |
| 1.3.6.1.4.1.19376.1.8.5.6 | 1597 | Multiple primary tumors |
| 1.3.6.1.4.1.19376.1.8.5.6 | 1598 | Recurrent |
| 1.3.6.1.4.1.19376.1.8.5.6 | 1599 | Post-treatment |
| 1.3.6.1.4.1.19376.1.8.5.328 | ?? | ?? |
| 1.3.6.1.4.1.19376.1.8.5.245 | 623 | High grade |
| 1.3.6.1.4.1.19376.1.8.5.245 | 624 | Low grade |
| 1.3.6.1.4.1.19376.1.8.5.246 | 569 | G1: Well differentiated |
| 1.3.6.1.4.1.19376.1.8.5.246 | 570 | G2: Moderately differentiated |
| 1.3.6.1.4.1.19376.1.8.5.246 | 571 | G3: Poorly differentiated" |
| 1.3.6.1.4.1.19376.1.8.5.192 | ?? | ?? |
| 1.3.6.1.4.1.19376.1.8.5.329 | ?? | ?? |
| 1.3.6.1.4.1.19376.1.8.5.330 | ?? | ?? |
| 1.3.6.1.4.1.19376.1.8.5.9 | 1433 | Concept pM UICC TNM 7ème édition |
| 1.3.6.1.4.1.19376.1.8.5.8 | 1432 | Concept pN UICC TNM 7ème édition |
| 1.3.6.1.4.1.19376.1.8.5.7 | 1431 | Concept pT UICC TNM 7ème édition |
| 1.3.6.1.4.1.19376.1.8.5.10 | 734 | No residual tumor (complete response, grade 0) |
| 1.3.6.1.4.1.19376.1.8.5.10 | 735 | Moderate response (grade 2) |
| 1.3.6.1.4.1.19376.1.8.5.10 | 736 | No definite response identified (grade 3, poor or no response) |
| 1.3.6.1.4.1.19376.1.8.5.10 | 2271 | Marked response (grade 1, minimal residual cancer) |
| 1.3.6.1.4.1.19376.1.8.5.331 | ?? | ?? |
| 1.3.6.1.4.1.19376.1.8.5.187 | ?? | ?? |
| 1.3.6.1.4.1.19376.1.8.5.184 | ?? | ?? |
| 1.3.6.1.4.1.19376.1.8.5.183 | ?? | ?? |
| 1.3.6.1.4.1.19376.1.8.5.3 | 799 | Single focus (Solitary - Unifocal) |
| 1.3.6.1.4.1.19376.1.8.5.3 | 800 | Multiple foci (Multifocal - (Ipsilateral – Bilateral))(specify location) |
| 1.3.6.1.4.1.19376.1.8.5.332 | ?? | ?? |
| 1.3.6.1.4.1.19376.1.8.5.333 | ?? | ?? |
| 1.3.6.1.4.1.19376.1.8.5.2 | 797 | Intact specimen(s) (Unopened, Capsule intact, Single intact specimen, Multiple intact, designated specimens (margins can be evaluated)) |
| 1.3.6.1.4.1.19376.1.8.5.2 | 798 | Non intact specimen (Open, Capsule ruptured specimen, Multiple non designated specimens (Fragmented, Morcellated)(margins cannot be evaluated with certainty) |
Tabelle 29: Value sets für Generische Cancer Check List (ohne Organspezifität)(OID 1.3.6.1.4.1.19376.1.8.2.1)
Cancer Check List und Value set für Mammakarzinome
| Name_LL | IHE_PAT_elementTemplates _Element_template_ID | IHE_PAT_Element_name | ValueSetID |
|---|---|---|---|
| DCIS_CRM_DIST | 1.3.6.1.4.1.19376.1.8.1.4.419 | Breast-In situ neoplasm-Distance of lesion from closest uninvolved margin | PQ |
| DCIS_GRAD | 1.3.6.1.4.1.19376.1.8.1.4.444 | Breast-In situ neoplasm-Histologic grade of ductal carcinoma in situ (DCIS) | 1.3.6.1.4.1.19376.1.8.5.23 |
| DCIS_Typ | 1.3.6.1.4.1.19376.1.8.1.4.446 | Breast-In situ neoplasm-Histologic type | 1.3.6.1.4.1.19376.1.8.5.254 |
| DCIS_DIMEN_LARG | 1.3.6.1.4.1.19376.1.8.1.4.442 | Breast-In situ neoplasm-Lesion size, largest dimension | PQ |
| DCIS_MARG_INVOLV | 1.3.6.1.4.1.19376.1.8.1.4.414 | Breast-In situ neoplasm-Lesion size, largest dimension | BL |
| DCIS_NECROSIS | 1.3.6.1.4.1.19376.1.8.1.4.429 | Breast-In situ neoplasm-Necrosis of ductal carcinoma in situ (DCIS) | 1.3.6.1.4.1.19376.1.8.5.253 |
| INV_CRM_DIST | 1.3.6.1.4.1.19376.1.8.1.4.418 | Breast-Infiltrating malignant neoplasm-Distance of lesion from closest uninvolved margin | PQ |
| INV_ER | 1.3.6.1.4.1.19376.1.8.1.4.439 | Breast-Infiltrating malignant neoplasm-Estrogen receptor | 1.3.6.1.4.1.19376.1.8.5.31 |
| INV_PgR | 1.3.6.1.4.1.19376.1.8.1.4.438 | Breast-Infiltrating malignant neoplasm-Progesterone receptor | 1.3.6.1.4.1.19376.1.8.5.34 |
| INV_HER2_ISH | 1.3.6.1.4.1.19376.1.8.1.4.416 | Breast-Infiltrating malignant neoplasm-HER2/neu (FISH method) | 1.3.6.1.4.1.19376.1.8.5.32 |
Tabelle 30: Codeliste für Cancer Check List Mammakarzinom(OID 1.3.6.1.4.1.19376.1.8.1.1.2.1)
| ValueSetID | PathLex_Code | PathLex_Value |
|---|---|---|
| 1.3.6.1.4.1.19376.1.8.5.23 | 743 | low grade |
| 1.3.6.1.4.1.19376.1.8.5.23 | 744 | intermediate grade |
| 1.3.6.1.4.1.19376.1.8.5.23 | 745 | high grade |
| 1.3.6.1.4.1.19376.1.8.5.254 | 2308 | Ductal carcinoma in situ with microinvasion |
| 1.3.6.1.4.1.19376.1.8.5.254 | 2309 | Lobular carcinoma in situ with microinvasion |
| 1.3.6.1.4.1.19376.1.8.5.254 | 2557 | DCIS Comedo |
| 1.3.6.1.4.1.19376.1.8.5.254 | 2558 | DCIS Paget disease (DCIS involving nipple skin) |
| 1.3.6.1.4.1.19376.1.8.5.254 | 2559 | DCIS Cribriform |
| 1.3.6.1.4.1.19376.1.8.5.254 | 2560 | DCIS Micropapillary |
| 1.3.6.1.4.1.19376.1.8.5.254 | 2561 | DCIS Papillary |
| 1.3.6.1.4.1.19376.1.8.5.254 | 2562 | DCIS Solid |
| 1.3.6.1.4.1.19376.1.8.5.253 | 2168 | Not identified |
| 1.3.6.1.4.1.19376.1.8.5.253 | 2169 | Present, focal (small foci or single cell necrosis) |
| 1.3.6.1.4.1.19376.1.8.5.253 | 2170 | Present, central (expansive “comedo” necrosis) |
| 1.3.6.1.4.1.19376.1.8.5.31 | 1602 | No immunoreactive tumor cells present |
| 1.3.6.1.4.1.19376.1.8.5.31 | 2269 | Immunoreactive tumor cells present (> = 1%) (Specify Quantitation) |
| 1.3.6.1.4.1.19376.1.8.5.31 | 2270 | Less than 1% immunoreactive cells present |
| 1.3.6.1.4.1.19376.1.8.5.34 | 740 | No immunoreactive tumor cells present |
| 1.3.6.1.4.1.19376.1.8.5.34 | 2267 | Immunoreactive tumor cells present (> = 1%) (Specify Quantitation) |
| 1.3.6.1.4.1.19376.1.8.5.34 | 2268 | Less than 1% immunoreactive cells present |
| 1.3.6.1.4.1.19376.1.8.5.32 | 741 | Amplified (HER2 gene copy >6.0 or ratio >2.2) |
| 1.3.6.1.4.1.19376.1.8.5.32 | 742 | Not amplified (HER2 gene copy <4.0 or ratio <1.8) |
| 1.3.6.1.4.1.19376.1.8.5.32 | 1925 | Equivocal (HER2 gene copy 4.0 to 6.0 or ratio 1.8 to 2.2) |
| 1.3.6.1.4.1.19376.1.8.5.x | yy | etc. |
Tabelle 31: Value sets für Cancer Check List Mammakarzinom(OID 1.3.6.1.4.1.19376.1.8.2.1)
Cancer Check List und Value set für kolorektale Karzinome
| Name_LL | IHE_PAT_elementTemplates _Element_template_ID | IHE_PAT_Element_name | ValueSetID |
|---|
TBD
Tabelle 32: Codeliste für Cancer Check List kolorektale Karzinome(OID 1.3.6.1.4.1.19376.1.8.1.1.2.2)
| ValueSetID | PathLex_Code | PathLex_Value |
|---|
TBD
Tabelle 33: Value sets für Cancer Check List kolorektale Karzinome(OID 1.3.6.1.4.1.19376.1.8.2.2)
Cancer Check List und Value set für Prostatakarzinome
| Name_LL | IHE_PAT_elementTemplates _Element_template_ID | IHE_PAT_Element_name | ValueSetID |
|---|
TBD
Tabelle 34: Codeliste für Cancer Check List Prostatakarzinome(OID 1.3.6.1.4.1.19376.1.8.1.1.2.3)
| ValueSetID | PathLex_Code | PathLex_Value |
|---|
TBD
Tabelle 35: Value sets für Cancer Check List Prostatakarzinome(OID 1.3.6.1.4.1.19376.1.8.2.3)
Anhang A: Diverses
Offene Punkte
- Codesysteme vervollständigen
- fehlende Abschnitte:
- n.n.
- Elemente und Attribute vollständig auflisten (inkl. Wikifizierung)
- Abgleich mit IHE Anatomic Pathology APSR und HL7 Anatomic Pathology
- ..
Beispieldokument
CDA-Header
<?xml version="1.0" encoding="UTF-8"?>
<?xml-stylesheet type="text/xsl" href="vhitg-cda-v3.xsl"?>
<ClinicalDocument xmlns="urn:hl7-org:v3"
xmlns:sciphox="urn::sciphox-org/sciphox"
xmlns:xsi="http://www.w3.org/2001/XMLSchema-instance"
xsi:schemaLocation="urn:hl7-org:v3 CDA.xsd">
<typeId root="2.16.840.1.113883.1.3" extension="POCD_HD000040"/>
<id extension="60467,36049" root="1.2.276.0.58"/>
<code code="11488-4" codeSystem="2.16.840.1.113883.6.1"
displayName="Consultation note"/>
<title>Pathologisch-anatomische Begutachtung mit kritischer Stellungnahme </title>
<effectiveTime value="20100224"/>
<confidentialityCode code="N" codeSystem="2.16.840.1.113883.5.25"/>
<languageCode code="de"/>
<setId extension="D1" root="2.16.840.1.113883.3.933"/>
<versionNumber value="1"/>
<recordTarget>
<!--- Patienten-Daten -->
<patientRole>
<id extension="6" root="2.16.840.1.113883.3.933"/>
<addr>
<streetName>Steinstr.</streetName>
<houseNumber>12</houseNumber>
<postalCode>30156</postalCode>
<city>Hamburg</city>
</addr>
<telecom use="WP" value="tel:040-555-12345"/>
<telecom use="HP" value="tel:040-222-76543"/>
<patient>
<name>
<prefix>Dr.</prefix>
<given>Alfred</given>
<family>Hafer</family>
</name>
<administrativeGenderCode code="M" codeSystem="2.16.840.1.113883.5.1"/>
<birthTime value="19450601"/>
<birthplace>
<place>
<addr>
<city>Sassnitz</city>
</addr>
</place>
</birthplace> </patient>
<providerOrganization>
<telecom use="WP" value="tel:06151-1111111"/>
<telecom use="WP" value="fax:06151-2222222"/>
<addr>
<streetName>Musterstr.</streetName>
<houseNumber>1</houseNumber>
<postalCode>64283</postalCode>
<city>Darmstadt</city>
</addr>
</providerOrganization>
</patientRole>
</recordTarget>
<author>
<!--- author -->
<time value="20060924"/>
<assignedAuthor>
<id extension="6319123" root="2.16.840.1.113883.3.933"/>
<assignedPerson>
<name>
<prefix qualifier="AC">Dr.med.</prefix>
<given>Hans</given>
<family>Topp-Gluecklich</family>
</name>
</assignedPerson>
<representedOrganization>
<name>Praxis Dr.med. Gluecklich </name>
<telecom use="WP" value="tel:06151-1111111"/>
<telecom use="WP" value="fax:06151-2222222"/>
<addr>
<streetName>Musterstr.</streetName>
<houseNumber>1</houseNumber>
<postalCode>64283</postalCode>
<city>Darmstadt</city>
</addr>
</representedOrganization>
</assignedAuthor>
</author>
<custodian>
<!--- Organisation von der das Dokument stammt -->
<assignedCustodian>
<representedCustodianOrganization>
<id extension="M345" root="1.2.276.0.58"/>
<name>Praxis Dr.med. Gluecklich </name>
<telecom nullFlavor="UNK"/>
<addr>
<streetName>Musterstr.</streetName>
<houseNumber>1</houseNumber>
<postalCode>64283</postalCode>
<city>Darmstadt</city>
</addr>
</representedCustodianOrganization>
</assignedCustodian>
</custodian>
<informationRecipient typeCode="PRCP">
<!--- Empfaenger -->
<intendedRecipient classCode="PUB">
<id extension="21233445" root="2.16.840.1.113883.3.933"/>
<receivedOrganization>
<name>Institut für Onkologie des Krankenhauses XYZ</name>
<telecom use="WP" value="fax:02431/901-6210"/>
<addr>
<streetName>Postbox 11 52</streetName>
<postalCode>24099</postalCode>
<city>Kiel</city>
</addr>
</receivedOrganization>
</intendedRecipient>
</informationRecipient>
<legalAuthenticator>
<!--- legalAuthenticator -->
<time value="20060721"/>
<signatureCode code="S"/>
<assignedEntity>
<id extension="6319123" root="2.16.840.1.113883.3.933"/>
<assignedPerson>
<name>
<prefix qualifier="AC">Dr.med.</prefix>
<given>Hans</given>
<family>Topp-Gluecklich</family>
</name>
</assignedPerson>
<representedOrganization>
<name>Praxis Dr.med. Gluecklich </name>
<telecom use="WP" value="fax:061512222222"/>
<addr>
<streetName>Musterstr.</streetName>
<houseNumber>1</houseNumber>
<postalCode>64283</postalCode>
<city>Darmstadt</city>
</addr>
</representedOrganization>
</assignedEntity>
</legalAuthenticator>
<component>
<structuredBody>
... (s.u.)
</structuredBody>
</component>
</ClinicalDocument>
CDA-Body
<component>
<structuredBody>
<component> <!-- Anrede -->
<section>
<code code="X-SALUT" codeSystem="2.16.840.1.113883.6.1" />
<text>
<paragraph>Sehr geehrter Herr Kollege Dr. Heitmann,</paragraph>
<paragraph>Vielen Dank für die freundliche Überweisung
des Patienten Paul Pappel, geb. 12. Dez. 1955.
</paragraph>
</text>
</section>
</component>
<component> <!-- Fragestellung -->
<section>
<code code="X-RFR" codeSystem="2.16.840.1.113883.6.1" />
<title>Fragestellung</title>
<text>Tod verursacht durch onkologische Erkrankung?</text>
</section>
</component>
<component>
<!-- Diagnose mit ICD Komponente -->
<section>
<code code="27754-1" codeSystem="2.16.840.1.113883.6.1"
codeSystemName="LOINC"/>
<title>22.02.2010: Diagnosen mit ICD 10</title>
<text>
<paragraph> Diagnose: <content ID="diag-1">????????</content>
</paragraph>
</text>
<entry>
<observation classCode="OBS" moodCode="EVN">
<code code="DISDX" codeSystem="2.16.840.1.113883.3.7.1.16"
codeSystemName="LOINC" displayName="Entlassdiagnosen"/>
<statusCode code="completed"/>
<effectiveTime>
<low value="20050829"/>
</effectiveTime>
<value xsi:type="CD" code="O????"
codeSystem="1.2.276.0.76.5.?????"
codeSystemName="ICD10gm2010"
displayName="?????">
<originalText>
<reference value="\#diag-1"/>
</originalText>
<qualifier>
<name code="8" codeSystem="2.16.840.1.113883.3.7.1"
displayName="Diagnosesicherheit"/>
<value code="G" codeSystem="2.16.840.1.113883.3.7.1.8"
displayName="Gesichert"/>
</qualifier>
</value>
</observation>
</entry>
</section>
</component>
<component>
<section>
<!--
<templateID root="1.2.276.0.76.3.1.81.1.4.xxx"/>
-->
<code code="??????????" codeSystem="1.2.276.0.76.3.1.81.1.3.4.xxxx
codeSystemName="xxxx"/>
<title>xxxx</title>
<text>xxxxxx</text>
</section>
</component>
<component>
<!-- Empfehlung -->
<section>
<code code="????????" codeSystem="2.16.840.1.113883.6.1"
codeSystemName="LOINC"/>
<title>Weitergabe</title>
<text>Geben Sie diesen Befund an den behandelnden Arzt weiter.</text>
</section>
</component>
<component> <!-- Schlusstext -->
<section>
<text> Mit freundlichen, kollegialen Grüßen </text>
</section>
</component>
</structuredBody>
</component>
Referenzen/Literatur
| DIMDI, Alpha_Id: | Alpha-ID - Die Identifikationsnummer, http://www.dimdi.de/static/de/ehealth/alpha-id/index.htm |
| DIMDI, Verschl: | Anleitung zur Verschlüsselung, http://www.dimdi.de/static/de/klassi/diagnosen/icd10/icdsgbv20.htm |
| DIMDI, Basis: | Basiswissen Codieren, DIMDI 2004 |
| BMGS, 2004: | ICD-10-Bekanntmachung des BMGS, http://www.zi-berlin.de/Zi_ICD10Browser/zi_icd_10_browser.htm |
| InEK, Codierrichtlinien: | Deutsche Codierrichtlinien – Version 2005, Institut für Entgeltsystem im Krankenhaus (InEK gGmbH) 2004, http://www.g-drg.de/service/download/veroeff_2005/DKR2005_Endversion_PDF30_040916_1500.pdf |
| HL7 Datentypen: | HL7 Version 3 Datentypen und CMETs für das Deutsche Gesundheitswesen, www.hl7.de (Publikationen) |
| CDAr2Arztbrief: | Arztbrief auf Basis der HL7 Clinical Document Architecture Release 2 für das deutsche Gesundheitswesen, Version 1.50 vom 12.05.2006, herausgegeben vom VHitG, HL7 Deutschland und der Arbeitsgemeinschaft Sciphox, www.hl7.de (Publikationen)
http://www.hl7.de/download/documents/cdar2-arztbrief/Leitfaden-VHitG-Arztbrief-v150.pdf |
| Wiley: | TNM-System: Wiley Interscience |
Zeitangaben
In einem Bericht tauchen mehrere Zeitangaben auf, die hier einmal in Form einer Übersicht dargestellt werden sollen. Die Angaben in der Bedingung beziehen sich auf die Nummern aus der ersten Spalte, d.h. hierüber wird eine Reihenfolge etabliert:
| # | Datum | Art | Bedingung (bezogen auf #) |
im Krankenhaus | im Labor | HL7 V3 (CDA später) |
|---|---|---|---|---|---|---|
| 1 | Auftragserfassung | Beginn | x | Order | ||
| 2 | Auftragserfassung | Ende | 1 < 2 | x | ||
| 3 | Auftragsfreigabe | TS | 2 < 3 | x | ||
| 4 | Auftragsübermittlung | TS | 3 < 4 | x | ||
| 5 | Probenentnahme | von/bis | x | Specimen Specimen Collection Process | ||
| 6 | Probenversand (Ausgang) | TS | 5 < 6 | x | ||
| 7 | Auftragseingang | TS | 4 < 7 | x | Acknowledgement | |
| 8 | Auftragsbestätigung | TS | 7 < 8 | x | Promise | |
| 9 | Probeneingang | TS | 6 < 9 | x | Specimen-> Specimen Process Step | |
| 10 | Probenuntersuchung | Beginn | 9 < 10 7 < 10 |
x | documentationOf ServiceEvent->
Specimen | |
| 11 | Probenuntersuchung | Ende | 10 < 11 | x | ||
| 12 | Befundung | Beginn | 10 < 12 | x | Specimen-> ObservationEvent
author.time | |
| 13 | Befundung | Ende | 12 < 13 | x | ||
| 14 | Niederschrift Befund | Beginn | 13 < 14 | x | dataEnterer.time | |
| 15 | Niederschrift Befund | Ende | 14 < 15 | x | ||
| 16 | Freigabe Befund | TS | 15 < 16 | x | legalAuthenticator .time | |
| 17 | Übermittlung Befund | TS | 16 < 17 | x | Wrapper | |
| 18 | Befundeingang | TS | 17 < 18 | x | Wrapper | |
| 19 | Befund gelesen | TS | 18 < 19 | x | - |
Freigaben können mehrstufig erfolgen.
Mehrere Berichte in Abhängigkeit des Prozessschrittes