Pathologiebefund auf der Basis von CDA R2
|
|
Dieses Dokument gibt wieder:
Implementierungsleitfaden Pathologiebefund auf der Basis von CDA R2 (08). Die Teilmaterialien gehören der Kategorie cdapath an. |
HL7 Clinical Document Architecture Release 2
für das deutsche Gesundheitswesen
| Abstimmungsdokument | |||
|---|---|---|---|
| Version | Datum | Status | Realm |
| 08 | 2012 | ||
| noch kein download verfügbar | |||
| Kontributoren | ||
|---|---|---|
| Agfa HealthCare GmbH | Bonn | |

|
Vivantes Netzwerk für Gesundheit | Berlin |
| Prof.Dr. Gunter Haroske | Dresden | |
HL7 Deutschland e.V.
Geschäftsstelle Köln
An der Schanz 1
50735 Köln
Implementierungsleitfaden
Pathologie-Befunde auf Basisvon HL7 CDA Rel.2
zur Abstimmung durch die Mitglieder von HL7 Deutschland e.V.
Ansprechpartner:
Dr. Frank Oemig, Agfa HealthCare GmbH (Bonn)
Dokumentinformation
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Impressum
For the Anatomic Pathology Structured Report Version 2.0 (APSR 2.0), an IHE Project, the existing German "Leitfaden Pathologiebefund" has been modified to an IHE Technical Framework document, starting from the IHE reference text APSR, Trial Implementation, to be linked with Art-Decor for the specification of value sets, terminologies, and templates according HL7/CDA.
Dieser Leitfaden wird von einer Arbeitsgruppe des Interoperabilitätsforum in Kooperation mit dem Berufsverband der Pathologen erstellt. Er basiert auf dem Leitfaden "Arztbrief 2014", der parallel hierzu durch eine Arbeitsgruppe des Interoperabilitätsforums erstellt wird.
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Contact
- IHE PaLM, palm@ihe.net
- Prof.Dr. Gunter Haroske, BVDP, Berlin, haroske@icloud.com
- Francois Macary, PHAST, Paris, francois.macary@phast.fr
- Dr. Frank Oemig, Deutsche Telekom Healthcare Solutions GmbH, Bonn, Frank.Oemig@t-systems.de
- Dr. Kai-Uwe Heitmann, hl7@kheitmann.de
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Authors and copyrights, terms of use
Copyright permissions
This document has been developed by Agfa HealthCare GbmH, Bonn, in cooperation with the HL7 user group Germany e.V. The claims for use or publication are not restricted.
The content of this specification is public.
It should be noted that parts of this document are based on the reconciliation package 2 of May 17, 2009 and the HL7 version 3 normative edition 2008, for which © Health Level Seven, Inc. applies. For further information see http://www.hl7.de/ and http://www.hl7.org/.
The extension or rejection of the specification, in whole or in part, shall be notified in writing to the board of the user group and to the editors / authors.
All HL7 specifications, adapted and published to national circumstances, can be used in any type of application software without license and usage fees.
Health Level Seven, Inc. has granted permission to the IHE to reproduce tables from the HL7 standard. The HL7 tables in this document are copyrighted by Health Level Seven, Inc. All rights reserved. Material drawn from these documents is credited where used.
Disclaimer
Although this publication has been compiled with the utmost care, neither HL7 Deutschland e.V. nor the companies involved in the creation can assume any liability for direct or indirect damage that could arise through the content of this specification.
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Foreword
This is a supplement to the IHE Pathology and Laboratory Medicine Technical Framework V8.0. Each supplement undergoes a process of public comment and trial implementation before being incorporated into the volumes of the Technical Frameworks.
This supplement was published on September 27, 2017 for Public Comment, with a comment period extending to mid-November. The current content is the Trial Implementation version, which has taken in consideration the comments received.
This supplement describes changes to the existing technical framework documents.
“Boxed” instructions like the sample below indicate to the Volume Editor how to integrate the relevant section(s) into the relevant Technical Framework volume.
Replace Section X.X by the following:
Where the amendment adds text, make the added text bold underline. Where the amendment removes text, make the removed text bold strikethrough. When entire new sections are added, introduce with editor’s instructions to “add new text” or similar, which for readability are not bolded or underlined.
General information about IHE can be found at: http://www.ihe.net.
Information about the IHE Pathology and Laboratory Medicine domain can be found at: http://ihe.net/IHE_Domains.
Information about the structure of IHE Technical Frameworks and Supplements can be found at: http://ihe.net/IHE_Process and http://ihe.net/Profiles.
The current version of the IHE Technical Framework (if applicable) can be found at: http://ihe.net/Technical_Frameworks.
Comments may be submitted on IHE Technical Framework templates any time at http://ihe.net/ihetemplates.cfm. Please enter comments/issues as soon as they are found. Do not wait until a future review cycle is announced.
Acknowledgements
Following authors mainly contributed to this document:
- Francois Macary, PHAST, Paris
- Dr. Gunter Haroske, Federal Association of German Pathologists, Berlin
- Dr. Frank Oemig, Deutsche Telekom Healthcare Solutions GmbH, Bonn
- Dr. Riki Merrick, APHL, San Francisco
- Dr. Raj Dash, Duke University, Durham
They have to acknowledge the contributions of Dr. Kai U. Heitmann, who managed the cooperation between PaLM and ART-DECOR. It was the very first time that a complete IHE TF document could be developed by means of the ART-DECOR tools and a media wiki.
Inhaltsverzeichnis
- 1 Impressum
- 2 Contact
- 3 Authors and copyrights, terms of use
- 4 Foreword
- 5 Acknowledgements
- 6 Introduction to this Supplement
- 7 Dynamisches Modell / Dynamic Model
- 8 Statisches Modell / Static Model
- 8.1 Übersicht / Survey
- 8.2 Gesamtstruktur
- 8.3 Dokumenttypen / Document types
- 8.3.1 Use Cases
- 8.3.2 Dokumentenstruktur des Pathologiebefundberichts / Structure of the Anatomic Pathology Report
- 8.3.2.1 Sections, Entries und Child Elements
- 8.3.2.2 Allgemeine Beschränkungen des Pathologiebefundes (IHE APSR) gegenüber einem CDA R2 Dokument / General constraints added by IHE PAT to a CDA R2 document
- 8.3.2.3 Spezielle Beschränkungen des Pathologiebefundes gegenüber IHE-APSR (National Extensions) / Special constraints added by the Anatomic Pathology Report to IHE-APSR (National Extensions)
- 8.3.2.4 Allgemeine Struktur des Pathologiebefundes CDA R2
- 8.3.3 Abschnitte des Befundberichtes / Sections of the APSR
- 9 CDA-Header
- 10 CDA Body
- 10.1 Modell
- 10.2 Beispiele für Befunde
- 10.3 Pathologiebefundbericht
- 10.4 Section: Anrede
- 10.5 Section: Vorbefund
- 10.6 Section: Klinische Information
- 10.7 SubSection: Fragestellung
- 10.8 Material
- 10.9 Section: Material und Materialaufbereitung (Procedure Steps)
- 10.10 Section:Färbungen
- 10.11 Entry:Immunhistologie Mikroskopie
- 10.12 Section: Makroskopische Beschreibung
- 10.13 Section: Mikroskopische Beschreibung
- 10.13.1 Definition und Zweck
- 10.13.2 Beispiel
- 10.13.3 Spezifikation
- 10.13.4 Content Module, die in die Section eingebunden werden sollten
- 10.13.4.1 Ergebnisse spezieller Untersuchungsmethoden
- 10.13.4.1.1 Immunhistologie (siehe Entry cdapath:Färbung-Section (Template) )
- 10.13.4.1.2 Elektronenmikroskopie (z.Zt. kein strukturierter Befundvorschlag, d.h. kein Entry??)
- 10.13.4.1.3 Molekularpathologie(z.Zt. kein strukturierter Befundvorschlag, d.h. kein Entry??)
- 10.13.4.1.4 Präparatradiographie(z.Zt. kein strukturierter Befundvorschlag, d.h. kein Entry)
- 10.13.4.1 Ergebnisse spezieller Untersuchungsmethoden
- 10.14 Section: Unterbeauftragung
- 10.15 Section: Diagnostische Schlussfolgerung
- 10.16 Allgemeine Spezifikation aller APSR Entry Content Module
- 10.17 Section: Zusammenfassung: ausführl. kritische gutachterliche Stellungnahme/Epikrise/Kommentar
- 10.18 Section: Grundleiden/Todesursache (klininisch)
- 10.19 Section: Grundleiden/Todesursache (autoptisch)
- 10.20 Section: Äußere Leichenschau
- 10.21 Section: Innere Leichenschau
- 10.22 Section: Weitergabemodus
- 10.23 Section: Abschließende Bemerkungen (Schlusstext)
- 11 Vokabeldomänen
- 12 Anhang A: Diverses
Introduction to this Supplement
This supplement complements volume 1 of the PaLM technical framework with the description of the APSR 2.0 content profile, and volume 3 with the bindings, content modules and value sets, which specify this profile.
Open Issues and Questions
None yet
Closed Issues
APSR-07 – Representing the hierarchy of specimens in an entry : This APSR supplement enables to represent the hierarchy of specimens at the CDA level 3. The operations on specimen and production of child specimens are tracked in the “Procedure Steps” section, which has a level 3 entry.
APSR-10 – Observation related to multiple specimens: For example tumor staging may require combining data from multiple specimens (e.g., a breast excision with positive margins followed by a re-excision with clear margins – in this case the tumor size may be a composite of measurements from both specimens. Another example is staging of ovarian carcinomas with multiple biopsies of pelvis, peritoneum, nodes, omentum, etc.). To accommodate these use cases, each problem organizer as well as each observation may reference any number of specimens using the <specimen> child element. Each of these references may point to a detailed description of the specimen, in the "procedure steps" section.
APSR-11 – Derivative specimens:Specimens derived from primary specimens for ancillary studies, which may be sent to a reference lab or done in another part of the same institution, are included in the scope of this profile. The results produced on a derived specimen are attached to this specimen in the report.
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Dynamisches Modell / Dynamic Model
Übersicht / Survey
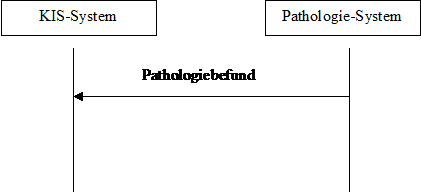
Das dynamische Modell sieht das relativ einfach aus:
The dynamic model looks rather simple:
Abbildung 1: dynamisches Modell Figure 1: dynamic model
Im Prinzip agiert das Pathologiesystem als Content Creator und das KIS-System als Content Consumer. Entsprechend können auch andere Systeme diese beiden Rollen übernehmen.
The Pathology Management System (PMS sive LIS) acts as Content Creator, whereas the Hospital Information System (HIS) plays the role of the Content Consumer. Other systems may play those roles, too.
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Statisches Modell / Static Model
Übersicht / Survey
In diesem Abschnitt wird grob der Aufbau und die Struktur von HL7 CDA R2 Dokumenten erläutert (entnommen aus dem Implementierungsleitfaden des VHitG-Arztbriefes, Kapitel 3).
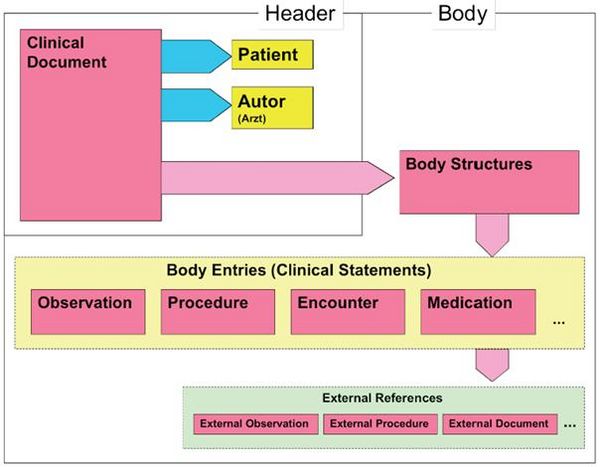
Wie alle Spezifikationen von Nachrichten in HL7 basiert auch die Clinical Document Architecture auf dem RIM und ist als HL7 V3 Modell repräsentiert. Grob gesprochen besteht ein CDA Dokument aus einem Header und einem Body, der wiederum Body Structures und Body Entries aufweist. An die Entries können externe Referenzen (External References) geknüpft sein. Der folgende Überblick zeigt die Hauptkomponenten des CDA R2 Modells auf und in der folgenden Abbildung ist das Ganze in XML-artiger Darstellung gezeigt.
As all HL7 specifications of messages the Clinical Document Architecture (CDA) is based on the Reference Information Model (RIM) and represented as HL7 V3 model. Roughly a CDA document consists of a header and a body, which contains Body Structures and Body Entries. The latter may be linked with external references. The figures 2 and 3 below show the main components of the CDA R2 model also in a xml-like fashion.
Abbildung 2: CDA-RMIM (vereinfachte Darstellung)
Die nachfolgende vereinfachte Graphik zeigt die Darstellung in XML:
Abbildung 3: CDA Level 3 Entries (vereinfachter Ausschnitt)
Die Informationen zum Patienten, zum Dokument selbst, zu den weiteren beteiligten Personen und Organisationen sowie der dokumentierten Episode (Zeitereignisse) sind zum CDA Header zusammengefasst, hochstrukturiert und von der Semantik her festgelegt.
Die Informationen im Header unterstützen einen Austausch klinischer Dokumente über Institutionsgrenzen hinweg. Er trägt Informationen über das Dokument selbst (eine eineindeutige Identifikation, eine Andeutung des Typs des Dokuments), über „Teilnehmer" am Dokument (an der Dokumentation beteiligte Heilberufler, Autoren, und natürlich den Patienten selbst), sowie über Beziehungen zu Dokumenten (zu Anforderungen und anderen Dokumenten). Mit den Informationen des Headers werden Dokumentenmanagementsysteme unterstützt, der Header stellt dafür entsprechende Mechanismen zur Verfügung. Schließlich hat man mit den im CDA Header verfügbaren Informationen die Zusammenführung einer individuellen (lebenslangen) Patientenakte vor Augen.
Information to the patient, to the document itself, to further persons and organizations who participate, as well as to the time period of documentation are comprised in the CDA Header, highly structured and defined by their semantics.
The header information supports the exchange of clinical documents across institution barriers. The header carries information about the document itself (unequivocal identification, type of document), about participants in the document (participating health officials, authors, and of course, the patient), as well as about relations to documents (queries and other documents). With the header information document management systems are supported. Finally, with the header information the compilation of a (livelong) health record of a patient is aimed.
Gesamtstruktur
|
|
Anm.: Die folgende Abbildung muss noch an die im weiteren ausgeführten Gliederungen angepasst werden. |
Abbildung 4: Gesamtstruktur
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Dokumenttypen / Document types
Pathologiebefunde erfassen alle Bereiche der Klinischen Pathologie (synonym "surgical pathology"), d.h. gut- und bösartige Tumoren, nichtneoplastische Läsionen, zytopathologische Befunde und Autopsien / Obduktionen. Zweit-/Nachberichte, Konsiliarberichte und Zytologische Befunde (gynäkologisch und nicht-gynäkologisch) weisen die gleiche Grundstruktur auf und sollten daher keine separaten Dokumententypen sein.
In IHE Anatomic Pathology Technical Framework Supplement Anatomic Pathology Structured Reports (APSR) Trial Implementation Tab. 5.6-1 wird durchgängig 11526-1 als Dokumententyp verwendet.
Obduktionsgutachten sind zwar anders aufgebaut als die Befundberichte zu Biopsien, Resektaten oder zytologischen Präparaten, stimmen aber in ihren Grundelementen überein. Sie sollten daher lediglich als ein spezielles Template behandelt werden. Von IHE wurden Autopsie-/Obduktionsbefunde bisher nicht erörtert.
In IHE-XDS-Registern können eine Klasse und feiner granulierte Typen angegeben werden. In CDA gibt es nur einen „Code“. Wir interpretieren das als den Typ, beim Registrieren kann dann die grobe Klassifizierung einfach ermittelt werden (umgekehrt wäre es schwierig). Einigen wir uns also auf 11526-1 "Pathology study" als Dokumentenklasse, darunter die beiden Dokumententypen 11529-5 und 18743-5.
Anatomic Pathology Reports about benign and malignant tumors and non-neoplastic lesions as well comprise all fields of Clinical Pathology (sive Surgical Pathology). Second opinion reports, additional reports, and cytopathologic reports have the same basic structure and should therefore not handled as separate document types.
In IHE Anatomic Pathology Technical Framework Supplement Anatomic Pathology Structured Reports (APSR) Trial Implementation Tab. 5.6-1 "11526-1" is generally used as document type.
Although Autopsy reports have the same basic elements as Pathology reports, they often show a different basic structure. They should be handled by specialized templates. Autopsy reports are not yet in the scope of IHE-Anatomic Pathology.
In IHE-XDS-Registries a class and more granulated types can be used. In CDA only a code is used. 11526-1 "Pathology study" should be understood as document class, beneath the both types 11529-5 und 18743-5 could be used for a finer granulation.
| Code | Dokumenttyp | Taxonomielevel |
|---|---|---|
| 11526-1 | Pathologisch- anatomische Begutachtung / Pathology study | 3 |
| 11529-5 | Pathologisch- anatomische Begutachtung / Surgical pathology study | 4 |
| 18743-5 | Obduktions-/ Sektionsgutachten / Autopsy report | 4 |
In der Taxonomie der Dokumenttypen finden sich die Spezifikationen für die beiden Dokumententypen im Taxonomielevel 4.
In the taxonomy of document types those specifications are to be found in taxonomy level 4.
Use Cases
Regelfall
Barbara Brust wird durch Dr. Christian Chirurg zur Entfernung eines Brusttumors aufgenommen. Dr. Christian Chirurg ordert die Untersuchung “Mammaexzisat - Pathohistologische Untersuchung” und sendet die Probe(n) zum Institut für Pathologie.
Die Probe(n) triftt im Institut ein. Dr. Peter Pathologe führt die makroskopische Untersuchung und den Zuschnitt durch; wenn nötig, werden Makrobilder aufgenommen. Die Probe(n) werden im Labor für die histologische Untersuchung einschließlich Spezialuntersuchungen aufbereitet, eine Gewebsasservation findet, falls nötig, statt. Während der mikroskopischen Untersuchung werden, falls nötig, Mikrobilder aufgenommen. Nach der mikroskopischen Untersuchung ruft Dr. Peter Pathologe die Content Creator Anwendung für das entsprechende APSR Template auf, füllt es aus, bindet relevante Bilder mit ROI ein und schließt den Fall ab. Nach Diktat und Kontrolle wird der Befund durch die Oberärztin Dr. Pauline Pathologin freigegeben.
Probengewinner ist nicht der Einsender
In Deutschland nicht üblich, Ablauf ansonsten wie Regelfall.
Mehrstufiger Bericht(Zweit-/Nachbericht)
Für Zweit-, besser Nachberichte gilt, dass sie neue Informationen zu einem bereits vorhandenen Befund hinzufügen (nachträgliche Spezialuntersuchungen, Zweitmeinungen, Konsiliarbefunde, Antworten auf klinische Fragestellungen, etc.). Damit besitzen sie die gleiche Struktur. Sie beziehen sich dabei immer auf einen vorhandenen Befund, in dem sie u.U. auch schon angekündigt werden (siehe ELGA-Beispiel). Sie stellen aber ein eigenes Dokument dar. Insofern muss geklärt werden, wie Beziehungen zwischen diesen Dokumenten hergestellt werden. (RPLC ist nicht geeignet, da das für die Versionierung genutzt werden muss. APND stellt schon eher das adäquate Konstrukt dazu dar.) Diese Art der Befunde müssen dann aber nicht separat modelliert werden.
Barbara Brust wird durch Dr. Christian Chirurg zur Entfernung eines Brusttumors aufgenommen. Dr. Christian Chirurg ordert die Untersuchung “Mammaexzisat - Schnellschnitt und Pathohistologische Untersuchung” und sendet die Probe(n) zum Institut für Pathologie.
Die Probe(n) triftt im Institut ein. Dr. Paul Putzig führt die makroskopische Untersuchung und den Zuschnitt durch; wenn nötig, werden Makrobilder aufgenommen. Die Probe(n) für den intraoperativen Schnellschnitt werden im Labor bearbeitet, Frischgewebe für eine Tumorbank wird entnommen. Während der mikroskopischen Untersuchung des Gefrierschnitts fordert Dr. Paul Putzig den Content Creator für das entsprechende APSR Template an, füllt die Felder aus, bindet relevante Bilder und /oder ROIs ein und signiert den vorläufigen Befund. Am nächsten Tag werden die Probe(n)für die histologische Untersuchung einschließlich Spezialuntersuchungen aufbereitet. Während der mikroskopischen Untersuchung werden, falls nötig, Mikrobilder aufgenommen. Nach der mikroskopischen Untersuchung ruft Dr. Peter Pathologe die Content Creator Anwendung für das entsprechende vorläufige APSR Template auf, füllt es aus, bindet relevante Bilder mit ROI ein und schließt den Fall endgültig ab. Nach Diktat und Kontrolle wird der Befund durch die Oberärztin Dr. Pauline Pathologin freigegeben.
Konsiliarbefund
Ein Konsiliarbefund stellt einen eigenständigen Befundbericht mit allen dafür notwendigen Abschnitten dar, der vom konsilsuchenden Auftraggeber (Pathologen) in dessen Erstbefund aufgenommen oder als Nachbericht an einen Erstbefund angefügt wird.
Es gibt verschiedene Situationen, in denen ein Konsiliarbefund angefordert wird:
- Externe Expertenkonsultation(erbeten durch Phillip Pathologe, oft bevor ein engültiger Befund fertiggestellt wird). - Bitte um eine Zweite Meinung(üblicherweise durch den behandelnden Arzt, den Patienten, seiner Familie oder in juristischem Zusammenhang). - Externe Schnittbegutachtung (üblicherweise Routinebegutachtung von Schnitten, die durch Protokolle einer externen Behandlungseinrichtung notwendig wird). Dies kann im Workflow und auch im zu begutachtenden Material variieren.
Anfordernder Pathologe
Phillip Pathologe bittet um eine zweite Meinung im Fall von Peter Patient, bei dem er ein malignes Lymphom diagnostizierte. Er sendet Blöcke oder Schnitte oder WSI zu Dr. Patrizia Pathologin und bittet sie um ihre (Experten-)Meinung zu diesem Material. Er verwendet die Content Creator Anwendung zur Erzeugung einer Konsultationsanfrage zum vorläufigen Befund von Peter Patient. Phillip Pathologe verwendet später seine Content Consumer Anwendung um den Befund von Dr. Patrizia Pathologin zu lesen und in seinen Befundbericht zu integrieren. Er verwendet diesen Befund daher um seinen eigenen Befund abzuschließen und mit seiner Anwendung als Content Creator zu agieren. Dabei wird ein vorläufiger Befundbericht fertiggestellt oder ein Zweit/Nachbericht zum Erstbericht erstellt.
Angefragter Pathologe
Dr. Patrizia Pathologin willigt ein, eine zweite Meinung zum Fall des Peter Patient abzugeben.
Blöcke
Die Proben werden für die mikroskopische untersuchung und notwendige zusätzliche Untersuchungsverfahren bearbeitet. Während des Mikroskopierens werden, falls nötig, Bilder aufgenommen. Zum Abschluss ihrer Untersuchungen ruft Dr. Patrizia Pathologin den Content Creator für das entsprechende APSR Template auf, füllt es aus, bindet Bilder und ROIs ein, und gibt den Befundbericht frei.
Schnitte
Während des Mikroskopierens werden, falls nötig, Bilder aufgenommen. Zum Abschluss ihrer Untersuchungen ruft Dr. Patrizia Pathologin den Content Creator für das entsprechende APSR Template auf, füllt es aus, bindet Bilder und ROIs ein, und gibt den Befundbericht frei.
WSI (Virtual slides)
Zum Abschluss ihrer Untersuchungen ruft Dr. Patrizia Pathologin den Content Creator für das entsprechende APSR Template auf, füllt es aus, bindet Bilder und ROIs ein, und gibt den Befundbericht frei.
Dokumentenstruktur des Pathologiebefundberichts / Structure of the Anatomic Pathology Report
Sections, Entries und Child Elements
IHE verwendet den Begriff der Content Module für unterschiedliche Hierarchie-Ebenen:
Auf der höchsten Ebene der Document Content Module (Dokumententypklasse LOINC 11526-1) ist der generische Anatomic Pathology Structured Report (1.3.6.1.4.1.19376.1.8.1.1.1) als Template für das gesamte Dokument, den Pathologiebefund, vorgesehen. Bisher ca. 20 Organ Specific APSR (1.3.6.1.4.1.19376.1.8.1.1.2.1 bis -2.20) werden als Templates für organspezifische Tumor-APSR in einer Appendix des APSR-Supplements für optionale Verwendung vorgehalten.
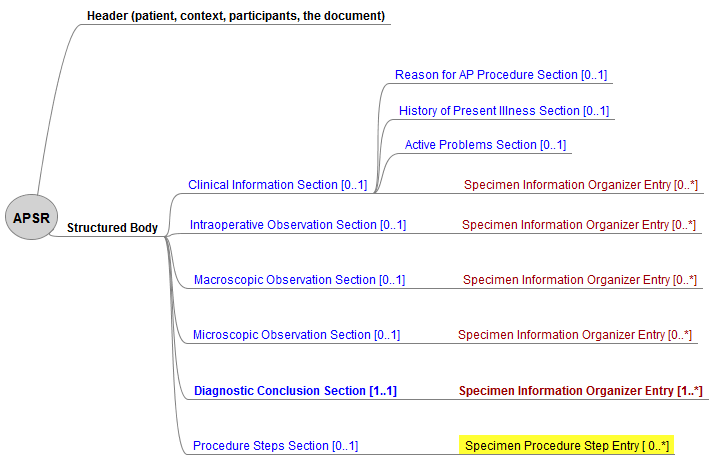
Nach IHE sind 6 Sections als Templates für <section> Elemente vorgesehen.
Diese können teilweise durch Entries als Templates für <entry> Elemete ergänzt werden (vgl. Fig. 4.1.2.1-1 APSR).
Child Elemente sind Templates für Elemente des CDA Headers oder für <section> Elemente oder für Elemente, die in verschiedenen Tiefen unterhalb eines <entry> Elements verschachtelt werden oder für ein Element aus Kombinationen dieser genannten.
Die OID Hierarchie der IHE ist wie folgt:
- 1.3.6.1.4.1.19376.1.8 Pathologiedomäne
- 1.3.6.1.4.1.19376.1.8.1 CDA Content Module
- 1.3.6.1.4.1.19376.1.8.1.1 Document Content Module
- 1.3.6.1.4.1.19376.1.8.1.2 Section Content Module
- 1.3.6.1.4.1.19376.1.8.1.3 Entry Content Module
- 1.3.6.1.4.1.19376.1.8.1.4 Element Content Module
- 1.3.6.1.4.1.19376.1.8.2 Terminologien
- 1.3.6.1.4.1.19376.1.8.2.1 PathLex
- 1.3.6.1.4.1.19376.1.8.5 Value Sets
- 1.3.6.1.4.1.19376.1.8.9 Beispielinstanzen
- 1.3.6.1.4.1.19376.1.8.1 CDA Content Module
Die Grundstruktur eines Befundes im deutschsprachigen Raum ist: Material - Makroskopische Beurteilung - Mikroskopische Beurteilung - Diagnose - Zusammenfassung. Diese wird durch o.g. Struktur vollständig abgebildet.
Der Pathologiebefundbericht als Sonderfall des Arztbriefs kann also weitgehend durch Verwendung von Arztbrief- und IHE-Templates als CDA R2-Dokument gestaltet werden.
At the highest level of Document Content Modules (document type class LOINC 11526-1) the generic Anatomic Pathology Structured Report (1.3.6.1.4.1.19376.1.8.1.1.1) is the template for the entire Pathology report. Up to now 20 Organ Specific APSR (1.3.6.1.4.1.19376.1.8.1.1.2.1 through -2.20) have been reserved as templates for organ-specific tumor-APSR in an appendix of the IHE-APSR Supplement.
This document content modul contains up to 6 Sections as templates for <section> elements.
Those can be added by Entries as templates for <entry> elements (see Fig. 4.1.2.1-1 APSR).
Child Elemente are templates for elements of the CDA header or for <section> elements or for elements, which are nested at various depths below an <entry>, or an element appearing at some combination of these locations.
The OID hierarchy of IHE is as follows:
- 1.3.6.1.4.1.19376.1.8 Pathology domain
- 1.3.6.1.4.1.19376.1.8.1 CDA Content Module
- 1.3.6.1.4.1.19376.1.8.1.1 Document Content Module
- 1.3.6.1.4.1.19376.1.8.1.2 Section Content Module
- 1.3.6.1.4.1.19376.1.8.1.3 Entry Content Module
- 1.3.6.1.4.1.19376.1.8.1.4 Element Content Module
- 1.3.6.1.4.1.19376.1.8.2 Terminologies
- 1.3.6.1.4.1.19376.1.8.2.1 PathLex
- 1.3.6.1.4.1.19376.1.8.5 Value Sets
- 1.3.6.1.4.1.19376.1.8.9 Instances in the examples built by the PAT TF
- 1.3.6.1.4.1.19376.1.8.1 CDA Content Module
The basic structure of an Anatomic Pathology Report in German speaking countries is: Specimen - Macroscopic evaluation - Microscopic evaluation - Diagnosis - Summary. This is completely mirrored by the structure described above.
The Pathology Report as a special (constrained) case of a discharge letter can also be derived from the German HL7 Discharge letter and from IHE-APSR templates as a valid CDA R2 document.
Any AP structured report SHALL be associated with the following metadata:
- typeCode = 11526-1, which is the LOINC code for “Pathology study”.
- formatCode = urn:ihe:pat:apsr:all:2010, with associated codingScheme = 1.3.6.1.4.1.19376.1.2.3 as assigned by the ITI Domain for codes used for the purposes of cross-enterprise document sharing (XDS).
- The media type SHALL be “text/xml”.
Allgemeine Beschränkungen des Pathologiebefundes (IHE APSR) gegenüber einem CDA R2 Dokument / General constraints added by IHE PAT to a CDA R2 document
Im strukturierten Body hat eine Section einen narrativen Block (text Element), der den lesbaren Inhalt der Section repräsentiert und der ein oder mehrere Entries haben KANN. Sections KÖNNEN ineinander verschachtelt sein,
Folgende Einschränkungen gelten:
- Wenn eine Section ein text Element und entry Elemente hat, dann MUSS der maschinenlesbare Inhalt vollständig in lesbaren Inhalt ins text Element transskribiert werden.
- Das text Element KANN Informationen enthalten, die nicht im maschinenlesbaren entry Element enthalten sind.
- Die entry Elemente werden nach Specimen oder Gruppen von Specimen instantiiert. Unterhalb eines entry Elements erfolgt die Organisation der Observations nach dem Problem.
- Das text Element einer Section sollte im Layout ebenfalls nach Specimen oder Gruppe von Specimen gegliedert werden.
- Das Layout des text Elementes obliegt der Content Creator Applikation oder weiteren Einschränkungen durch Nationale Körperschaften. Die Content Creator Applikation MUSS Freitext und aus entry Elementen transskribierten Text separat behandeln und sicherstellen, dass ein Überschreiben von Freitext durch transskribierten Text verhindert wird.
- Zu übertragende Information MÜSSEN Unterscheidungen klar identifizieren hinsichtlich:
- None: Es ist sicher bekannt, dass es keine Information gibt.
- None known: Es ist derzeitig nicht sicher bekannt, ob es keine Information gibt.
- Asked but unknown:Die Information wurde nachgefragt, konnte aber nicht erhalten werden (Ergebnis einer Untersuchung nicht bekannt).
- Unknown: Die Information ist unbekannt oder anderweitig nicht verfügbar.
- Other, not specified: Der Wert gehört nicht zum vereinbarten Wertesatz und wird vom Autor überhaupt nicht mitgeteilt.
- Other, specify: Der Wert gehört nicht zum vereinbarten Wertesatz und wird vom Autor als fremder Wert mitgeteilt.
- Not applicable: Es gibt keinen anwendbaren Wert für den gegebenen Kontext.
Sections, die R oder R2 Status haben und über keine Informationen verfügen, sollten eine dieser Begriffe im text Element verwenden.
Strukturelemente mit R oder R2 Status, die über keine Information verfügen solten ein "nullFlavor" Attribut zur Codierung des Grundes der fehlenden Information verwenden.
| Situation | nullFlavor | HL7 Definition |
|---|---|---|
| Asked but unknown | ASKU | Information wurde gesucht aber nicht gefunden |
| Unknown | UNK | Ein Wert wäre anwendbar, aber nicht vorhanden |
| Other, not specified | OTH | Der Wert ist kein Element aus der betreffenden Wertedomäne |
| Not applicable | NA | Es gibt keinen geeigneten Wert in diesem Kontext |
| Temporarily not available | NAV | Information ist im Moment nicht verfügbar, wird aber erwartet |
Die Situationen "None" und "None known" sind effektive Werte in den zugehärigen Wertesätzen. Die Situation "Other, specify" kann auf zweierlei Art gehandhabt werden:
- das code Attribute wird leer gelassen und die Antwort wird im nichtkodierten originalText Attribut aufgeführt,
- ein kodierter Wert aus einem anderen Codeschema wird eingetragen, wenn die Kodierungsstärke dieses Elements mit "CWE" (coded with extensions)definiert ist. Die Attribute code, display name und codeSystemName beschreiben dann den fremden Code.
Für zusätzliche Untersuchungstechniken wird die Situation "nicht durchgeführt" oder "nichts durchgeführt" durch den nullFlavor=NAV repräsentiert.
In the structured body of a CDA R2 document, a section has a narrative block (the text element), which presents the human-readable content of the section, and MAY have one entry or more. Sections MAY be nested within one another.
The content modules designed by the PAT TF bring or highlight the following constraints:
- When a section has a text element and one or more entry element, the content coded for machine-processing in the entries SHALL be completely transcribed into human-readable content in the textelement.
- Conversely the text element MAY contain pieces of information, which are not available in machine-readable format in any entry element of the section.
- For a document of the Anatomic Pathology domain, the entry elements are instantiated per specimen or per group of specimens observed together. One entry contains in machine-readable format observations of the section related to the same specimen or group of specimens. Beneath an entry, the observations are organized per problem.
- The text element of the section is supposed to be also laid out per specimen or group of specimens and per problem observed.
- The APSR Content Profile leaves the layout of the text element up to the Content Creator applications, or to further constraints brought by national extensions of this profile. However, given that the text element is usually composed of free text (e.g. dictated text), assembled with the text generated from the set of data, machine-encoded in the entry elements below, the Content Creator application MUST handle these two kinds of content, and provide a user interface, which avoids risks of overwriting text automatically derived from the entries with free text typed in by the user (e.g., using forms with dedicated free text areas and distinct protected areas for text generated out of structured data).
- Information that is sent SHALL clearly identify distinctions between:
- None: It is known with complete confidence that there are none. This indicates that the sender knows that there is no relevant information of this kind that can be sent.
- None Known: None known at this time, but it is not known with complete confidence that none exist.
- Asked but unknown: The information was requested but could not be obtained. Used mainly in the context where an observation was made but the result could not be determined.
- Unknown: The information is not known, or is otherwise unavailable.
- Other, not specified: The actual value does not belong to the assigned value set and is not reported at all by the author.
- Other, specify: The actual value does not belong to the assigned value set and the author of the report provides this foreign value anyway.
- Not applicable: No proper value is applicable in this context.
Sections that are required to be present but have no information should use one of the above phrases where appropriate in the text element.
Structural elements that are required but have no information shall provide a "nullFlavor" attribute coding the reason why the information is missing.
| Situation | nullFlavor | HL7 Definition |
|---|---|---|
| Asked but unknown | ASKU | information was sought but not found |
| Unknown | UNK | A proper value is applicable, but not known. |
| Other, not specified | OTH | The actual value is not an element in the value domain of a variable. (e.g., concept not provided by required code system). |
| Not applicable | NA | No proper value is applicable in this context |
| Temporarily not available | NAV | Information is not available at this time but it is expected that it will be available later. |
The two situations “None” and “None known” represent effective values, which are part of the related value sets.The situation “Other, specify” can be handled in two ways in a coded data element:
- Leaving empty the code attribute and providing the non-coded answer in the originalText attribute.
- Providing a value coded from a different coding scheme, when the coding strength of the element is "CWE"(coded with extensions). The attributes code, displayName, codeSystem and codeSystemName then describe the foreign code.
- For ancillary techniques, the situation "not performed" or "none performed" is represented by nullFlavor = NAV.
Spezielle Beschränkungen des Pathologiebefundes gegenüber IHE-APSR (National Extensions) / Special constraints added by the Anatomic Pathology Report to IHE-APSR (National Extensions)
 |
Gibt es derartige weitere Beschränkungen (z.B. Umgang mit Tumorformeln, Value sets bzgl. Organspezifität etc.)?? GH |
Organspezifische Document Content Module mit organspezifischen Beschränkungen in den Specimen Collection Procedures und AP Observations sind nicht vorgesehen.
Im Gegensatz zu IHE_APSR sind die Sections Intraoperative Untersuchung, Makroskopische Beurteilung, Mikroskopische Beurteilung sowie ausführliche gutachterliche Stellungnahme obligatorisch.
Organ specific Document Content Modules with organ specific constraints in Specimen collection procedures and AP Observations are not defined.
In contrast to IHE APSR the sections "Intraoperative Observation", "Macroscopic Observation", and "Microscopic Observation" are mandatory.
Allgemeine Struktur des Pathologiebefundes CDA R2
Die allgemeine Struktur eines Pathologieberichtes hinsichtlich seiner maschinenlesbaren Inhalte gestaltet sich wie von IHE-APSR vorgeschlagen:
 |
Die Abbildung weist eine Abweichung von der XML-Struktur im Beispiel auf: der component-Tag wird hier nicht dargestellt. --Tschrader 23:14, 31. Jul. 2013 (CEST) |
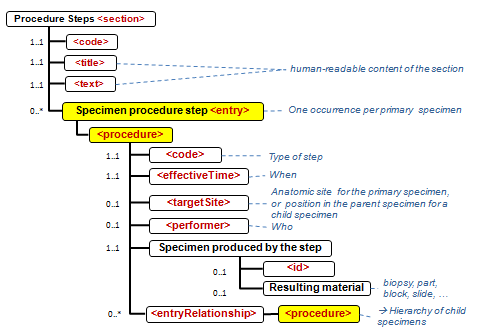
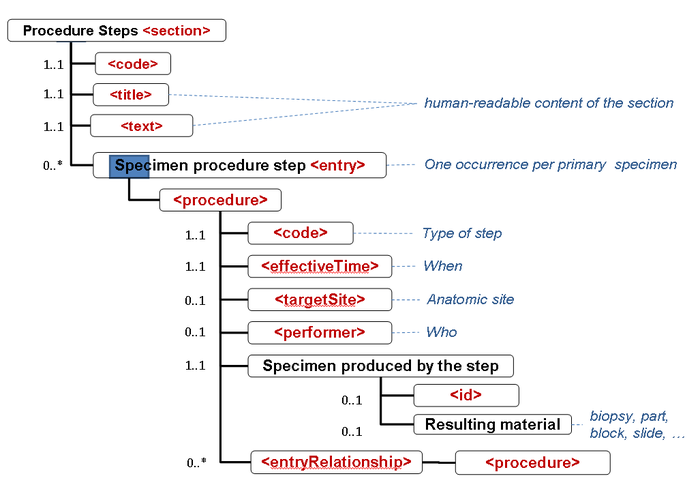
- in den Sections 1 - 5 werden werden keine Specimen collection procedure-Entries genutzt: Jede Observation oder Batterien von Observations werden durch eine Referenz auf das Specimen und das Problem, zu dem sie gehören, gebunden.
- die Informationen zu den Specimen (Proben, Materialien) werden in der (Procedure steps) Materialaufbereitungs Section durch das Specimen procedure step entry zentralisiert, so dass jedes Specimen jeder Hierarchstufe identifiziert werden kann (s.auch cdapath:Materialaufbereitung-Section (Template)).
Abschnitte des Befundberichtes / Sections of the APSR
Die Abschnitte des Befundberichts, untergliedert nach Sections und Entries, sind in nachfolgender Tabelle aufgelistet.
| Lvl | Dokumentabschnitt | Hierarchie | Pathologisch- anatomische Begutachtung/ Erstbericht |
Obduktions-/ Sektions- gutachten |
LOINC | Sektorkommitee | (IHE) OID |
|---|---|---|---|---|---|---|---|
| 1 | Anrede | Section | 0..1 | 0..1 | nein | ||
| 1 | Vorbefunde | Section | 0..1 | 0..1 | nein | ||
| 1 | Klinische Information | Section | 0..1 | 0..1 | 22636-5 | nein | 1.3.6.1.4.1.19376.1.8.1.2.1 |
| 2 | - Fragestellung | SubSection | 0..1 | 0..1 | 42349-1 | nein | 1.3.6.1.4.1.19376.1.5.3.1.3.1 |
| 2 | - Anamnese | SubSection | 0..1 | 0..1 | 10164-2 | nein | 1.3.6.1.4.1.19376.1.5.3.1.3.4 |
| 2 | - "Active problem" | SubSection | 0..1 | 0..1 | 11450-4 | nein | 1.3.6.1.4.1.19376.1.5.3.1.3.6 |
| 1 | Intraoperative Untersuchung | Section | 0..* | tbd | ja | 1.3.6.1.4.1.19376.1.8.1.2.2 | |
| 1 | Makroskopische Beschreibung | Section | 1..* | 22634-0 | ja | 1.3.6.1.4.1.19376.1.8.1.2.3 | |
| 1 | Mikroskopische Beschreibung | Section | 1..* | 1..1 | 22635-7 | ja | 1.3.6.1.4.1.19376.1.8.1.2.4 |
| 1 | Diagnose | Section | 1..* | 22637-3 | ja | 1.3.6.1.4.1.19376.1.8.1.2.5 | |
| 2 | - ausführliche kritische gutachterliche Stellungnahme/Epikrise/Kommentar | SubSection | 0..1 | 0..1 | 35660-0 | ja | 1.3.6.1.4.1.19376.1.8.1.2.7 |
| 1 | Materialaufbereitung | Section | 1..* | 1..1 | 46059-2 | ja | 1.3.6.1.4.1.19376.1.8.1.2.6 |
| 1 | Äußere Leichenschau | Section | 1..1 | ||||
| 1 | Innere Leichenschau | Section | 1..1 | ||||
| 1 | Unterbeauftragung | Section | 0..* | 0..* | ja | ||
| 1 | Diagnose(n) konsiliarischer Untersuchungen | Section | 0..* | 0..* | ja | ||
| 1 | Schlusstext | Section | 0..1 | 0..1 | nein |
Tabelle 1: Abschnitte des Befundberichts, deren Inhalt und zugehörige LOINC-Codes
(Level gibt hier die Schachtelung der Spezifikation an, d.h. ein höher Level bedeutet die Inklusion in einer vorhergehenden Section. Das ist kein Aussage über den CDA-Level, der den Grad der Kodierung angibt.)
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
CDA-Header
Die Regelungen zum Header können aus dem VHitG-Arztbrief, Kapitel 7, vollständig übernommen werden. Einzig die Liste der teilnehmenden Personen (participants) ist um den Einsender zu ergänzen, der in der Regel auch der beabsichtigte Empfänger des Dokuments (informationRecipient) ist. Er ist nicht identisch mit einem Ein- oder Überweiser.
IHE_APSR_TF_Supplement-2011-03-31 schlägt dafür noch zwei weitere participant-Rollen vor:
- Ordering physician (participant[@typeCode="REF"]/templateId[@root="1.3.6.1.4.1.19376.1.3.3.1.6"], HL-7 ORC-12, OBR-16, ORC-9
- Specimen collector (participant[@typeCode="DIST"]/templateId[@root="1.3.6.1.4.1.19376.1.8.1.4.1"], HL-7 OBR-10, SPM-17.
Beide in der usage-Kategorie R2 (required, if known)
Weiterhin gibt es den Dokumentenstatus, der durch das Code Element lab:statusCode unter documentationOf/serviceEvent als Extension zum CDA R2 Standard repräsentiert wird, um einen "vorläufigen Befund (lab:statusCode@code="active") von einem endgültigen Befund (lab:statusCode@code="completed") zu unterscheiden. Diese Extension wird geschützt durch einen Namespace assoziiert mit dem ClinicalDocument Element zum Präfix lab: <Clinical Document xmlns:lab="urn:oid:1.3.6.1.4.1.19376.1.3.2" … >
 |
GH: Ist das schon berücksichtigt? |
Die weiteren Ausführungen erübrigen sich damit bis auf die Dokumententypisierung!
In diesem Abschnitt werden die Elemente des CDA Headers erläutert, die zwingend in den CDA HL7 R2 Arztbrief einzubinden sind.
Alle XML Arztbriefe beginnen mit dem Wurzelelement ClinicalDocument und der vorgeschriebene Zeichensatz ist UTF-8.
Daraus ergibt sich folgende Struktur, die wie aufgeführt umzusetzen ist. Dabei sind fett gedruckte Bereiche unverändert einzubauen.
<?xml version="1.0" encoding="UTF-8"?>
<ClinicalDocument
xmlns="urn:hl7-org:v3"
xmlns:voc="urn:hl7-org:v3/voc"
xmlns:xsi="http://www.w3.org/2001/XMLSchema-instance">
<typeId root="2.16.840.1.113883.1.3" extension="POCD_HD000040"/>
<id extension="13234453645" root="2.16.840.1.113883.2.6.15.3.427.1"/>
<title>Pathologisch anatomische Begutachtung [mit kritischer Stellungnahme]</title>
<code code="11526-1" codeSystem="2.16.840.1.113883.6.1"/>
<effectiveTime value="200509241634"/>
<!-- CDA Header -->
... siehe Beschreibung CDA R2 Header
<!-- CDA Body -->
<component>
<structuredBody>
... siehe Beschreibung CDA R2 Body
</structuredBody>
</component>
</ClinicalDocument>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Patient
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.7 ???? | |
| General Description | In diesem Abschnitt im CDA Header wird der Patient beschrieben/erfasst. Dieser setzt sich zusammen aus einer Patientenrolle sowie dem Patienten selbst. Diese werden im recordTarget zusammengeführt. | |
| LOINC Code | Opt. | Description |
| ???? | O | |
Im CDA-Header muss mindestens eine Patientenrolle beschrieben sein, die genau von einer Person gespielt wird.
| Lvl | RIM | Name | Desc | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|---|
| 1 | part | recordTarget | Patient | 1..1 | required | ||
| 2 | role | patientRole | Patient | 1..1 | required | Die Rolle des Patienten wird durch eine Person gespielt.
| |
| 3 | role | id | ID des Patienten | 1..1 | required | Verpflichtend muss in diesem Bereich die Patientenidentifikationsnummer angegeben werden. Diese setzt sich zusammen aus dem @extension Attribut, das die ID des Patienten enthält sowie dem @root Attribut, das die OID des Systems enthält, das die ID vergeben hat.
| |
| 4 | role | extension | ID des Patienten | 1..1 | required | ||
| 4 | role | root | ID des Patienten | 1..1 | required |
| |
| 3 | ent | Person | 0..1 | optional | Patient | ||
| 4 | ent | name | Name des Patienten | 0..1 | optional | In dem Attribut @name ist der Name des Patienten untergebracht. Der Name wird wiederrum unterteilt in die @given und @family Attribute, die den Vornamen und den Familiennamen des Patienten enthalten. |
Ein kompletter recordTarget ist im Folgenden angegeben.
<recordTarget>
<!--- Patienten-Daten -->
<patientRole>
<id extension="6245" root="2.16.840.1.113883.3.933"/>
<patient>
<name>
<prefix>Dr.</prefix>
<given>Paul</given>
<family>Pappel</family>
</name>
</patient>
</patientRole>
</recordTarget>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Autor
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.7 ???? | |
| General Description | Autor des Berichts | |
| LOINC Code | Opt. | Description |
| ???? | O | |
| Lvl | RIM | Name | Desc | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|---|
| 1 | part | author | Autor | 1..1 | required | Autor: Neben dem Patienten muss ein Autor (author) angegeben werden, welcher das Dokument verfasst hat. | |
| 2 | part | time | Zeitpunkt | TS | 1..1 | required | Im verpflichtend anzugebenden @time Attribut wird der Zeitpunkt der Dokumentation angegeben. |
| 2 | role | author | Autor | 1..1 | required | Informationen über den Autor werden in der assignedAuthor Klasse angegeben. | |
| 4 | ent | name | Name des Autors | PN | 0..1 | optional | In dem Attribut @name ist der Name des Autors untergebracht. Der Name wird wiederum unterteilt in die @given und @family Attribute, die den Vornamen und den Familiennamen des Autors enthalten. |
Ein kompletter author ist im Folgenden angegeben.
<author>
<time value="20050829"/>
<assignedAuthor>
<id extension="190388km89" root="2.16.840.1.113883.3.24535"/>
<assignedPerson>
<name>
<prefix>Dr.med.</prefix>
<given>Theo</given>
<family>Phyllin</family>
</name>
</assignedPerson>
</assignedAuthor>
</author>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Verwaltende Organisation
| Template-Metadaten | |
| Template-Typ | Header |
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.7 ???? |
| generischeres Template | |
| genutztes Templates | |
| nutzende Templates | |
| abgeleitete Templates | |
| Schwester-Templates | |
| Generelle Beschreibung | Dieses Template spezifiziert, wer das Dokument verwaltet. |
| allg. Erläuterung | |
| Verhältnis zu IHE | |
| Ballotierungsstatus | in Arbeit |
| Erweiterbarkeit | offen oder geschlossen |
| Lvl | RIM | Name | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|
| 1 | part | custodian | 1..1 | R | verwaltende Organisation | |
| 2 | part | @typeCode | 1..1 | F | "CST" | |
| 2 | role | assignedCustodian | 1..1 | M | ||
| 3 | role | @classCode | 1..1 | F | ASSIGNED | |
| 3 | ent | representedCustodianOrganization | 1..1 | M | Organisation | |
| 4 | role | id | 1..1 | M | Identifikation
| |
| 4 | role | name | 0..1 | O | Name der Organisation |
Die Organisation (custodian), die für die Verwaltung des Dokuments verantwortlich ist, muss verpflichtend in der entsprechenden Klasse wiedergegeben werden.
Die Organisation muss mindestens mit einer ID gekennzeichnet werden.
Ein kompletter custodian ist im Folgenden angegeben.
<custodian typeCode="CST">
<assignedCustodian classCode="ASSIGNED">
<representedCustodianOrganization>
<id extension="175648374" root="1.2.276.0.76.4.5"/>
<name>
...
</name>
</representedCustodianOrganization>
</assignedCustodian>
</custodian>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
CDA Body
Die eigentliche klinische Dokumentation wird im so genannten CDA Body festgehalten. Im Vordergrund steht hier „lesbarer" (narrativer) Text, der verpflichtender Bestandteil von CDA R2 Dokumenten ist und die Interoperabilität zwischen den menschlichen Kommunikationspartnern garantiert. Hier sind Möglichkeiten gegeben, diesen Text grob zu strukturieren, wie man dies von den Möglichkeiten der Textverarbeitung her kennt. Zur Strukturierung stellt die Standardspezifikation eine Reihe von XML-Elementen zur Verfügung, die als Body Structures zusammengefasst werden können. Der Body enthält ein oder mehrere Abschnitte (sections). Diese können auch ineinander geschachtelt sein, so wie Kapitel und Unterkapitel in einem Buch. Zudem sind Strukturierungen im textuellen Bereich im Sinne von Tabellen oder Listen möglich.
- Abschnitte <section>
- Paragrafen <paragraph>
- Kennzeichnung von bestimmten Inhalten <content>
- Überschriften <caption>
- Tabellen <table>
- Listen <list>
Sections enthalten immer einen narrativen Block und erfüllen damit eine der oben genannten Maximen von CDA: die Mensch-zu-Mensch-Interoperabilität, die Lesbarkeit der Informationen für den Menschen. Im narrativen Block, durch das Textattribut in der section-Klasse repräsentiert, wird eingebetteter Text innerhalb eines Abschnittes angegeben. Dabei kann mit oben genanntem <content> Element bestimmter Inhalt gesondert gekennzeichnet werden.
Zusammengefasst werden im Textblock (teils so auch schon in CDA Release 1 realisiert) u.a. folgende Möglichkeiten der Struktur- und Formgebung des fließenden Textes gegeben:
- Zeilenumbrüche <br>
- Stilistische Angaben (unterstreichen, fett, kursiv etc.)
- Hoch- und Tiefstellung von Text
- Fußnoten
- Symbole
- Revisionsmarken im Text wie <delete>, <insert>
Mit den beschriebenen Body Strukturen können CDA Entries verbunden sein. Diese repräsentieren den „computerlesbaren Teil" innerhalb eines Dokumentenabschnitts. Body Entries sind im Prinzip eine Auswahl aus Klassen mitsamt Attributen aus dem HL7 Referenz-Informationsmodell (RIM).
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
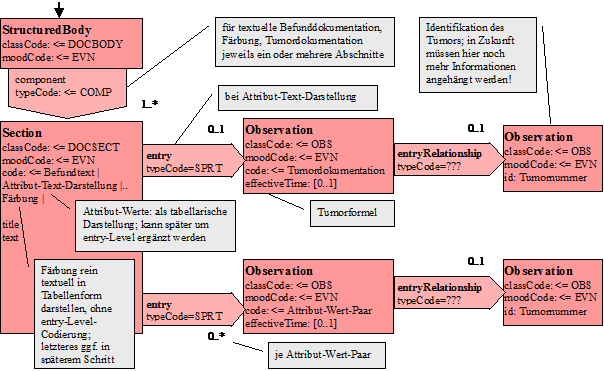
Modell
Nachfolgend ist das CDA-Modell angegeben, so wie es für den Pathologie-Bericht instanziiert wird:
Abbildung 5: Level-3-Modell
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Beispiele für Befunde
Zu Beginn ein relativ einfaches und kurzes Beispiel:
Material:
Zystenbalg Regio 38
Makroskopische Beurteilung:
Zusammen pampelmusenkerngroße (Durchmesser 12 mm) membranöse festelastische grauweißliche Gewebsstücke.
Mikroskopische Beurteilung:
Partiell durch ein sehr schmales, nicht verhorntes Plattenepithel ausgekleideter Balganteil einer odontogenen Zyste mit einzelnen Malassezschen Epithelnestern und herdbetonter sehr schütterer rundzelliger entzündlicher subepithelialer Infiltration. Eingesprengt wenig Hartmaterial.
Diagnose:
Follikuläre Zyste. Kein Anhalt für Malignität oder Spezifität am vorliegenden Material.
Unterschrift
Material:
Sonographisch gestützte Stanzbiopsie Mamma re. 10 Uhr
Makroskopische Beurteilung:
Mehrere (gemäß klinischer Angabe fünf) zusammen 51 mm lange weiche bis mittelfeste teils grauweißliche, teils graugelbliche Punktionszylinder von max. Bleistiftminenstärke.
Mikroskopische Beurteilung: (HE, CK 5/14, CK 7):
Alle gewonnenen Punktionszylinder wurden vollständig gebettet und mit 13 Schnittstufen untersucht. Sie bestehen aus lipomatös und fibrolipomatös transformiertem Brustdrüsengewebe und lassen in mindestens sechs Punktionszylinderteilstücken neben atrophischen Drüsenlobuli und ektatischen Milchgangsanschnitten sowie kleineren Adenoseherden (mikrozystische und blunt-duct-Adenosen) in wechselnder Dichte in ein hyalinelastotisch transformiertes Stroma eingebettete unterschiedlich großkalibrige Tubuli erkennen, diese vielfach mit Abknickungen, die ausgekleidet werden von einem einreihigen, überwiegend gering, örtlich mäßig atypischen flach kubischen Epithel ohne nennenswerte mitotische Aktivität und fehlende basale Myoepithellage in der CK 5/14-Reaktion, vereinbar mit Tumorinfiltraten eines tubulären Mammakarzinoms (Malignitätsgrad 1 nach Elston und Ellis; 1+2+1); vgl. auch EH-Nr. 7082/12.
Diagnose: Tubuläres Karzinom der Mamma.
Klassifikation nach NHSBSP: B 5b
Zum Ergebnis der noch ausstehenden Hormonrezeptorbestimmung und des HER-2/neu- Status erfolgt ein Nachbericht. Telefonische Vorabinformation am 31.05.2012.
Unterschrift
1. Nachbericht:
Ergebnis der immunhistochemischen Hormonrezeptorbestimmung:
Östrogenrezeptoren: über 80 % positiver Zellkerne, vorherrschende Färbeintensität: stark, IRS 12 nach Remmele und Stegner.
Progesteronrezeptoren: über 80 % positiver Zellkerne, vorherrschende Färbeintensität: stark, IRS 12 nach Remmele und Stegner.
Der Tumor ist endokrin-responsiv.
Ergebnis der in-situ-Hybridisierung (BDISH) des HER2-neu-Gens:
Nach einer in-situ-Hybridisierung des Genlocus c-erb-B2 und des Zentromer von Chromosom 17 wurden die ISH-Signale von 30 Tumorzellen ausgezählt. Die untersuchten invasiven Tumorzellen zeigen ein mittleres Verhältnis 1,19 (Gen/Zentromer). Es liegt also keine Amplifikation des HER2-neu Lokus vor (ZytoDot 2C SPEC Her- 2/CEN17 Probe Kit).
Unterschrift
Material:
1. Sentinel-LK re. 2. Segmentresektat Mamma re.
Makroskopische Beurteilung:
1. Backpflaumengroßes (40 x 20 x 15 mm messendes) Fettgewebsstück, darin
eingeschlossen drei, max. gewürzkorngroße (größter Durchmesser 6 mm) mittelfeste
grauweißliche Gewebsknoten.
2. Auf Styroporplatte nadelfixiertes, zweifach fadenmarkiertes fettgewebsreiches
Mammaresektat aus dem zentralen Drüsenkörper von 119 g in einer Ausdehnung
von 6,5 x 7 x 4 cm mit zentral aufsitzender, 4 cm durchmessender Mamille/Areole.
Mamille und unmittelbar retromamilläres Drüsenkörpergewebe in einer Ausdehnung
von 12 mm derb knotig grauweiß induriert. Auf der Mamille selbst aufgelagert
eine kleine Kruste.
Das Resektat wird von lateral beginnend in neun Scheiben lamelliert. Weitere
verdächtige Knotenbildungen kommen nicht zur Darstellung.
Sicherheitszonen der beschriebenen Knotenbildungen zu allen Resektionsrändern
mindestens 10 mm bzw. deutlich darüber.
Schnellschnittdiagnose:
1. Alle drei Sentinellymphknoten im Schnellschnitt metastasenfrei.
2. Karzinom unmittelbar retromamillär, Sicherheitszonen nach allen Seiten
mindestens 10 mm bzw. deutlich darüber.
Telefonische Befundübermittlung am 15.06.2012, 11.59 Uhr an Herrn CA Dr. XY.
Mikroskopische Beurteilung:
1. Alle drei Sentinellymphknoten, die vollständig gebettet und in Stufenserienschnitten
mit jeweils 12 Schnittstufen aufgearbeitet wurden, sind metastasenfrei.
Man erkennt kleinherdige lipomatöse Transformationen und narbige Fibrosierungen.
Umgebendes Fettgewebe unauffällig.
2. Das Mammaresektat wurde unter Berücksichtigung der Topografie und Markierungen mit 13 Paraffinblöcken mit jeweils Schnittstufen untersucht. In der Haut der Mamillenregion bzw. unmittelbar retromamillär unter Einbeziehung der lokalisationstypisch größeren Ductus und Sinus lacteripheri Tumorgewebe des in der Stanzbiopsie vordiagnostizierten invasiven Mammakarzinoms (vgl. EH-Nr. 7226/12), jetzt als gemischt invasiv duktales und lobuläres Mammakarzinom zu klassifizieren (Malignitätsgrad 2 nach Elston und Ellis; 3+2+1) mit vordergründig dissolut einzelzelligem Wachstumsmuster mit Ausbildung sog. indian files und Targetstrukturen um unbeeinträchtigte Milchgänge, aber auch tubulären Differenzierungen und kleiner intraduktaler Komponente, die an umschriebener Stelle den invasiven Karzinomanteil nach dorsal um 1 mm überschreitet. Es besteht eine wechselnd stark ausgeprägte Stromasklerose mit örtlich schütter rundzellig entzündlicher Stromareaktion und man sieht in einem der Schnittpräparate eine umschriebene, herdförmig eingeblutete narbige Fibrose mit reaktiven Veränderungen der bedeckenden Epidermis bei Zustand nach vorausgegangener Punchbiopsie. Karzinomentfernung im Gesunden, Sicherheitszonen nach allen Seiten jeweils deutlich über 10 mm. Breit anhängender Saum tumorfreien Brustdrüsengewebes betont lipomatös transformiert ohne sonstigen nennenswerten pathologischen Befund.
Zusammenfassung:
Ungewöhnlich oberflächlich retromamillär bzw. in der Haut der Mamillenregion rechts gelegenes gemischt invasiv duktales und lobuläres Mammakarzinom. Karzinomentfernung im Gesunden (Sicherheitszonen nach allen Seiten über 10 mm). Drei metastasenfreie Sentinellymphknoten.
Unterbeauftragte Untersuchung:
Unfixiertes Tumorgewebe wurde zur uPA/PAI-1-Bestimmung an das Institut für Pathologie des UKD gesandt. Über das Ergebnis wird nachberichtet.
Tumorklassifikation:
TNM (UICC, 7. Auflage):
pT1c pN0 (0/3sn), R0, L0, V0
Grading:
G 2
ICD-O-3:
C 50.0, M 8522/3
Unterschrift
1. Nachbericht:
Ergebnis der uPA/PAI-1-Analyse:
Methodik: Am unfixierten Gewebe des invasiven Mammakarzinoms wurde nach Kryokonservierung mit dem FEMTELLE® ELISA-Test die Aktivität des Plasminogenaktivators vom Urokinasetyp (uPA) und des Plasminogen-Inhibitors (PAI-1) bestimmt (Institut für Pathologie des UKD, Prof. Dr. XYZ).
Untersuchungsergebnis:
uPA: 6,27 ng/mg Protein
PAI-1: 12,4 ng/mg Protein
Bewertung:
Bei diesem Ergebnis kann bei nodal-negativen Mammakarzinom, Malignitätsgrad 2 nach Elston und Ellis entsprechend der Interdisziplinären S3-Leitlinie für die Diagnostik, Therapie und Nachsorge des Mammakarzinoms von einem hohen Rezidivrisiko ausgegangen werden.
Unterschrift
Material:
Zystoprostatektomiepräparat, Absetzungsrand Harnröhre tumorfrei?
Makroskopische Beurteilung:
Zystoprostatektomiepräparat bestehend aus der im eröffneten Zustand 6,5 x 5,5 x 3 cm messenden Harnblase, der 5,0 x 3,5 x 3,0 cm messenden Prostata, einem 7 cm langen und maximal 0,3 cm durchmessenden Ureteranteil rechts, einem maximal 7 cm langen und maximal 0,3 cm durchmessenden Ureteranteil links, einem 8 cm langen Ductus deferens-Anteil der rechten Seite von maximal 4 mm Durchmesser sowie einem 8 cm langen Ductus deferens-Anteil der linken Seite von maximal 4 mm Durchmesser sowie der maximal 3 cm durchmessenden rechten Samenblase und der maximal 3 cm durchmessenden linken Samenblase. Der Harnrˆhrenabsetzungsrand ist fadenmarkiert. Im Bereich der rechten Harnblasenseitenwand/-hinterwand zeigt sich ein maximal 2,5 cm durchmessender ulkusartiger Defekt (a). Eindeutiges Tumorgewebe lässt sich makroskopisch nicht abgrenzen. Die Prostata ist von kleinknotigem Aufbau und weist kleinzystische Hohlraumbildungen auf. Im perivesikalen Weichgewebe lassen sich keine Gewebsknoten makroskopisch abgrenzen.
Schnellschnittdiagnose:
Tumorfreier Harnröhrenabsetzungsrand.Telefonische Befundübermittlung an Herrn CA Prof. Dr. Urologe um 10:54 Uhr am 27.03.2013.
Mikroskopische Beurteilung:
Das Zystoprostatektomiepräparat wurde mit 18 Paraffinblöcken und zahlreichen Schnittstufen histologisch untersucht. Im oben beschriebenen Bereich der Harnblase (a) finden sich partiell ulzerierte papilläre und solide urotheliale Karzinomstrukturen, die eine mäßige bis starke Kernpolymorphie aufweisen. Tumorinfiltration aller Harnblasenwandschichten, örtlich kleinherdig bis in das unmittelbar angrenzende perivasikale Fettgewebe reichend. Verschiedentlich Lymphangiosis carcinomatosa. In der angrenzenden Harnblasenschleimhaut sowie in der linken Harnblasenseitenwand und im Harnblasenscheitel fokale urotheliale Dysplasien aller Schweregrade bis zu einem Carcinoma in situ. Der Ureter am Resektionsrand beiderseits ist tumorfrei. In der Pars prostatica urethrae multifokales Carcinoma in situ mit Übergreifen auf von Brunnsche Zellnester. Hier keine Tumorinfiltration durch das Urothelkarzinom. Der Harnröhrenabsetzungsrand ist auch im paraffingebetteten Material tumorfrei. Im rechten Prostataseitenlappen finden sich herdförmig azinäre und tubuläre Karzinomstrukturen, die zwischen nicht neoplastische Drüsen infiltrieren sowie eine geringe bis mäßige Kernpolymorphie und prominente Nukleolen aufweisen. Apex prostatae sowie Ductus deferens und Samenblase beiderseits sind tumorfrei.
Diagnose:
Schlecht differenziertes Urothelkarzinom der Harnblase, im Gesunden entfernt. Verschiedentlich Lymphangiosis carcinomatosa. In der Tumorumgebung und in der Pars prostatica urethrae multifokales Carcinoma in situ.
Tumorklassifikation: (im Zusammenhang mit EH-Nr. 4259/13):
TNM (UICC, 7. Auflage): pT3a pN0 (0/17), L1, V0, R0
Grading: G 3, high grade
ICD-O-3: C 67, M 8130/33
Mäßig differenziertes azinäres Adenokarzinom im rechten Prostataseitenlappen, im Gesunden entfernt.
Tumorklassifikation: (im Zusammenhang mit EH-Nr. 4259/13):
TNM (UICC, 7. Auflage): pT2a pN0 (0/17), L0, V0, R0
Grading: Gleason-Score: 3+3=6, G IIa nach Helpap
ICD-O-3: C 61, M 8140/3
Unterschrift
PathoBerichtText
Wiederholung der Ki-67-Färbung von der 2. Fraktion (rechts).
Beurteilung
1. Isolierte Tumorzellen in einem funktionsgesteigerten Lymphknoten (linke Axilla Sentinellymphknoten 544cps).
2. Teils glanduläres, teils solides, invasives ductales Adenocarcinom der Brustdrüse, geringe nukleäre Atypien sowie ductales Carcinoma in situ mit geringen Atypien, DCIS I und Mikrokalk bis 0,2mm (rechte Mamma oben zwischen den Quadranten, Resektat); immunhistologisch fokal starke Expression des Östrogenrezeptors in etwa 60% der Tumorzellen.
Score nach Elston und Ellis: 4.
Immunreaktiver Score: Östrogenrezeptor 9, Progesteronrezeptor 0.
Onkoprotein C-erbB-2 Index: 0
Größter Durchmesser des invasiven Carcinoms etwa 9mm, das invasive Carcinom reicht zumindest an die craniale Abtragungsebene im peripheren (cranialen) Pol. Durchmesser der in situ Komponente etwa 35mm, Abstand von der nächstgelegenen (posterioren) Abtragungsebene etwa 0,4mm, die in situ Komponente breitet sich allerdings ebenfalls in den peripheren (cranialen) Pol aus und erreicht damit dort mindestens die Abtragungsebene.
3. Weitgehend solides, invasives ductales Adenocarcinom der Brustdrüse, mittelgradige, herdförmig schwere nukleäre Atypien, etwa in gleicher Größe ductales Carcinoma in situ mit schweren Atypien, DCIS III sowie Nekrosen und Verkalkungen bis 4mm (Resektat linke Mamma oben außen); immunhistologisch starke Expression des Östrogenrezeptors und des Progesteronrezeptors jeweils in etwa 85% der Tumorzellen. Immunreaktiver Score: Östrogenrezeptor 12, Progesteronrezeptor 12. Kleine Wachstumsfraktion (Ki-67 um 10 %). Onkoprotein C-erbB-2 (Her-2-Neu-Index: 0). Größter Durchmesser der invasiven Komponente und der in-situ-Komponente jeweils etwa 23mm. Abstand der invasiven Komponente von der nächstgelegenen Abtragungsebene (posterior) 3,2mm, Abstand von anterior 12mm, von cranial 20mm, von caudal 23mm, von medial 30mm, von lateral 13mm. Die in-situ-Komponente breitet sich zumindest bis an die posteriore Abtragungsebene aus.
4. Neun tumorfreie Lymphknoten (linke Axilla).
5. Tumorfreies Fettgewebe (linke Mamma, Nachresektat lateral).
6. Überwiegend tumorfreies Fettgewebe mit kleinen Anteilen von tumorfreiem Brustdrüsengewebe (linke Mamma, Nachresektat mamillenwärts).
7. Überwiegend tumorfreies Fettgewebe mit kleinen Anteilen von tumorfreiem Brustdrüsengewebe (linke Mamma, Nachresektat cranial).
Auch nach Wiederholung lässt sich die Wachstumsfraktion in dem kleinen Tumor auf der rechten Seite nicht darstellen, vermutlich wurde das Antigen durch die schwere thermische Schädigung zerstört.
Pathologiebefundbericht
| Template ID | 1.3.6.1.4.1.19376.1.8.1.1.1 (AP APSR 6.2.3.1) | |
| General Description | In diesem Template werden die Daten zum Pathologiebefundbericht übermittelt. | |
| Status | identisch zu IHE | |
| LOINC Code | Opt. | Description |
| 11526-1 | R | Pathology Study |
Definition und Zweck
Dieses Template definiert die Grundlage für die Einschränkungen jedes strukturierten Pathologiebefundberichts unabhängig von Organ oder Diagnose. Es ist damit das generische Template für jeden strukturierten Pathologiebefund in allen Gebieten der Pathologie (z.B. Entzündung, Degeneration, Kreislaufstörung, gut- und bösartige Tumoren) sowie Zytopathologie.
Die Hierarchie von Sections und Entries ist in der IHE-Abb. dargestellt. Die einzige obligatorische Section (R) ist die Diagnostische Schlussfolgerung Section, der einzige obligatorische Entry ist der Specimen Information organizer Entry in dieser Section.
CDA APSR_R2 (IHE): Hierarchy of the body for APSR document content module
Die Sections 1 bis 5 zeigen folgende Grundstruktur:
Die Section 6 (Materialbearbeitung / Procedure Steps) weicht davon ab:
APSR_Common_Structure_Section6.PNG
Beispiel
<ClinicalDocument xmlns='urn:hl7-org:v3'>
<typeId extension="POCD_HD000040" root="2.16.840.1.113883.1.3"/>
<templateId root='1.3.6.1.4.1.19376.1.8.1.1.1'/>
<id root='1.3.6.1.4.1.19376.1.8.9' extension='123'/>
<code code='11526-1' displayName='Pathology study'
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title>Anatomic pathology structured report</title>
<effectiveTime value='20100114153000-0700'/>
<confidentialityCode code='N' displayName='Normal' codeSystem='2.16.840.1.113883.5.25'/>
<languageCode code='en-US'/>
<!-- one patient -->
<recordTarget><patientRole> .. </patientRole></recordTarget>
<!-- one or more author -->
<author> .. </author>
<!-- one or more transcriptionists -->
<dataEnterer> .. </dataEnterer>
<!-- one or more person who provided useful information as input to this document -->
<informant> .. </informant>
<!-- the organization (laboratory) issuing this report and in charge with its lifecycle -->
<custodian> .. </custodian>
<!-- zero or more intended recipient other than the ordering physician (« copy to ») -->
<informationRecipient> .. </informationRecipient>
<!-- the person (lab director) legally responsible for this report, who may have signed it -->
<legalAuthenticator> .. </legalAuthenticator>
<!-- one or more pathologists who validated and/or corrected the content -->
<authenticator> .. </authenticator>
<!-- the ordering physician, and the date-time the order was issued -->
<participant typeCode='REF'> .. </participant>
<!-- zero or more distinct specimen collector, and the date-time the specimen was collected -->
<participant typeCode='DIST'> .. </participant>2
<inFulfillmentOf> .. </inFulfillmentOf>
<!-- The service documented and the primary laboratory having performed it -->
<documentationOf> .. </documentationOf>
<!-- zero or one encompassing encounter -->
<component><encompassingEncounter> .. </encompassingEncounter></component>
<!-- The body of the report -->
<component>
<structuredBody>
<component>
<section>
<templateId root='1.3.6.1.4.1.19376.1.8.1.2.1'/>
<code code='22636-5' displayName=’Pathology report relevant history'
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title>CLINICAL INFORMATION</title>
<text>
Tissue submitted: left breast biopsy and apical axillary tissue
</text>
<component>
<section>
<templateId root= '1.3.6.1.4.1.19376.1.5.3.1.3.1'/>
<code code='42349-1' displayName= ‘Reason for referral’
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title>Reason for anatomic pathology procedure</title>
<text>Breast mass - left breast</text>
</section>
</component>
<component>
<section>
<templateId root='1.3.6.1.4.1.19376.1.5.3.1.3.4'/>
<code code=’10164-2’ displayName= ‘History of present illness’
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title> History of present illness </title>
<text>Carcinoma of breast. Post operative diagnosis: same.
left UOQ breast mass.
</text>
</section>
</component>
</section>
</component>
<component>
<section>
<templateId root='1.3.6.1.4.1.19376.1.8.1.2.2'/>
<code code='TBD' displayName='intraoperative section in anatomicpathology report'
codeSystem='2.16.840.1.113883.6.1'codeSystemName='LOINC'/>
<title>INTRAOPERATIVE OBSERVATION</title>
<text> </text>
<entry> </entry>
</section> </component>
<component>
<section>
<templateId root='1.3.6.1.4.1.19376.1.8.1.2.3'/>
<code code='22634-0' displayName='Pathology report gross observation'
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title>MACROSCOPIC OBSERVATION</title>
<text> </text>
<entry> <entry/>
:
</section>
</component>
<component>
<section>
<templateId root='1.3.6.1.4.1.19376.1.8.1.2.4'/>
<code code='22635-7' displayName='Pathology report microscopic observation'
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title>MICROSCOPIC OBSERVATION</title>
<text> </text>
<entry> <entry/>
:
</section>
</component>
<component>
<section>
<templateId root='1.3.6.1.4.1.19376.1.8.1.2.5'/>
<code code='22637-3' displayName='Pathology report diagnosis'
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title>DIAGNOSTIC CONCLUSION SECTION</title>
<text> </text>
<entry> <entry/>
:
</section>
</component>
<component>
<section>
<templateId root='1.3.6.1.4.1.19376.1.8.1.2.6'/>
<code code='46059-2' displayName=' Special treatments and procedures section '
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title>PROCEDURE STEPS SECTION</title>
<text> </text>
<entry> <entry/>
:
</section>
</component>
</structuredBody>
</component>
</ClinicalDocument>
Spezifikation
Die Spezifikation des Header-Abschnitts ist in jenem Abschnitt beschrieben.
 |
Inwieweit die sehr umfangreiche Header-Spezifikation von IHE damit weiter eingeschränkt werden soll, muss entschieden werden. GH |
In folgender Tabelle ist die Spezifikation des Body ausgeführt.
| Datenelement | Verw. | Kard. | Path and Constraints (Xpath + indentation) | Vokabular / Quelle | Bemerk. | DT | HL7 V2.5.1 |
| Report body | R | [1..1] | component/structuredBody | ||||
| Anrede Section | O | [0..1] | component/section
templateId[@root="2.999.889.777.22.77.10.10"] |
11 | |||
| Klinische Information Section | R2 | [0..1] | component/section
[/templateId/@root="1.3.6.1.4.1.19376.1.8.1.2.1"] |
PAT TF-3: 6.2.4.1 | 12 | OBR-13 | |
| Intraoperative Untersuchung
Section |
R2 | [0..1] | component/section
[/templateId/@root="1.3.6.1.4.1.19376.1.8.1.2.2"] |
PAT TF-3: 6.2.4.2 | 13 | ||
| Makroskopische Untersuchung
Section |
R2 | [0..1] | component/section
[/templateId/@root="1.3.6.1.4.1.19376.1.8.1.2.3"] |
PAT TF-3: 6.2.4.3 | 14 | ||
| Mikroskopische Untersuchung
Section |
R2 | [0..1] | component/section
[/templateId/@root="1.3.6.1.4.1.19376.1.8.1.2.4"] |
PAT TF-3: 6.2.4.4 | 15 | ||
| Diagnostische Schlussfolgerung Section | R | [1..1] | component/section
[/templateId/@root="1.3.6.1.4.1.19376.1.8.1.2.5"] |
PAT TF-3: 6.2.4.5 | 16 | ||
| Materialaufbereitung Section | R | [1..1] | component/section
[/templateId/@root="1.3.6.1.4.1.19376.1.8.1.2.6"] |
PAT TF-3: 6.2.4.6 | 17 | ||
| Schlusstext Section | O | [0..1] | component/section
templateId[@root=" ????? "] |
18 |
Bemerkungen:
(11): Die Anrede ist in DE im Pathologiebefund optional. Wenn vorhanden, wird sie wie im Arztbrief behandelt.
(12): Die Klinische Information Section enthält die Angaben des einsendenden Arztes: Anamnese, prä- und postoperative Diagnosen, Laborwerte, Probenbenennung, Probengewinnung, Fragestellung.
(13): Die Inraoperative Untersuchung Section enthält die Intraoperative (Gefrierschnitt-)Diagnose für jedes untersuchte Material, die Probenbeschreibung und die Benennung der intraoperativen Untersuchungsverfahren sowie für Spezialuntersuchungen abgezweigte Probenteile (z.B. Durchflusszytometrie, Zytogenetik, molekularpathologische Untersuchungen, Elektronenmikroskopie).
(14): Die Makroskopische Beschreibung Section enthält die Beschreibung aller erhaltenen Proben (Probentyp und -zustand), Links zu Makrofotos, die prozessurale Beschreibung und die Gewebsverwendung für zusätzliche Untersuchungsverfahren sowie die versendung in Biobanken.
(15): Die Mikroskopische Beschreibung Section enthält die histopathologischen Befunde zusammen mit den ergebnissen der zusätzlichen Untersuchungsverfahren (z.B. Histochemie, Immunhistochemie, Molekularpathologie).
(16): Die Diagnostische Schlussfolgerung Section enthält die Diagnosen aller eingesandten Proben eines Falles. Sie werden für jede Probe oder jede gruppe von Proben separat dargestellt. Die Section schließt zusätzliche Untersuchungsbefunde, Diagramme, statische bilder und Virtual Slides ein. Sie sammelt den minimalen Satz für die Diagnose relevanter Informationen.
(17): Die Materialaufbereitung (Procedure Steps) Section enthält die Beschreibung des eingesandten Materials, die Auflistung der zu beantwortenden Probleme, sowie der Gewebsaufarbeitung hinsichtlich repräsentativer Proben und davon abgeleitete Proben für zusätzliche Untersuchungsmethoden oder Biobanken. Eine Problemauflistung wird in deutschen Pathologiebefunden textlich nicht vorgenommen, sie bestimmt aber weitgehend die Struktur und den Umfang des Befundtextes. Sie muss in dieser Section auf Entry-Level vorhanden sein. Die Gewebsaufarbeitung ist in deutschen Pathologiebefunden optional.
(18): Der Schlusstext ist in DE im Pathologiebefund optional. Wenn vorhanden, wird er wie im Arztbrief behandelt. cdapath:Modell
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Section: Anrede
| Id | 1.2.276.0.76.10.3001 | Gültigkeit | 2013‑01‑10 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Status | Versions-Label | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Name | Salutation | Bezeichnung | Anrede | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Kontext | Elternknoten des Template-Element mit Id 1.2.276.0.76.10.3001 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Klassifikation | CDA Section level template | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Offen/Geschlossen | Offen (auch andere als die definierten Elemente sind erlaubt) | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Beziehung | Spezialisierung: Template 2.16.840.1.113883.10.12.201 CDA Section (2005‑09‑07) ref ad1bbr- | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Beispiel |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Section: Vorbefund
|
|
Diese Section wird unter cdapath nicht mehr benötigt. Die Seite kann wohl für andere Template durch verschieben recycelt werden. |
| Template ID | <OID für das Template> | |
| General Description | In diesem Abschnitt werden Vorbefunde aufgelistet. | |
| Status | genuin | |
| LOINC Code | Opt. | Description |
| ???? | O | |
Vorbefunde werden i.d.R. vom Pathologie-Management-System (PMS) bereit gestellt. Im Strukturierten Befund sollten sie allenfalls mit der jeweiligen Fall-Nr. aufgeführt werden, sofern sie Relevanz zum aktuellen Befund besitzen.
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Section: Klinische Information
| Template-Metadaten | |
| Template-Typ | Section |
| Template ID | 1.3.6.1.4.1.19376.1.8.1.2.1 (APSR 6.2.4.1) |
| generischeres Template | <Verweis auf das Template> |
| genutztes Templates | <Verweis auf das Template> |
| nutzende Templates | <Verweis auf das Template> |
| abgeleitete Templates | <Verweis auf das Template> |
| Schwester-Templates | <Verweis auf das Template> |
| Generelle Beschreibung | In diesem Abschnitt werden die Daten zu Klinischen Angeben übermittelt. |
| allg. Erläuterung | vom Sektorkommitee Pathologie/Neuropathologie nicht gefordert |
| Verhältnis zu IHE | identisch zu IHE (Übersetzung) |
| Ballotierungsstatus | in Arbeit |
| Erweiterbarkeit | geschlossen |
Definition und Zweck
Die klinische Information Section enthält diejenigen informationen, die der Einsender bereitstellt: Anamnese, prä- u./o. postoperative (Verdachts-)Diagnose, Anlass der Untersuchung, Laborwerte, Entnahmestelle, Materialtyp, klinische Materialbeschreibung, Tumorlokalisation.
|
|
Hier sollte noch ein Modell hin! Das erleichtert das Verständnis des Beispiels ungemein. |
Beispiel
<section>
<templateId root="1.3.6.1.4.1.19376.1.8.1.2.1"/>
<code code="22636-5" displayName="Pathology report relevant history" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC"/>
<title>CLINICAL INFORMATION</title>
<text> Tissue submitted: left breast biopsy and apical axillary tissue </text>
<entry typeCode="COMP">
<!-- Specimen Information Organizer -->
<organizer classCode="CLUSTER" moodCode="EVN">
<templateId root="1.3.6.1.4.1.19376.1.8.1.4.4"/>
<statusCode code="completed"/>
<component>
<!-- Specimen collection procedure -->
<procedure classCode="PROC" moodCode="EVN">
<templateId root="1.3.6.1.4.1.19376.1.3.1.2"/>
<code code="2257" codeSystem="1.3.6.1.4.1.19376.1.8.2.1" displayName="Breast-Specimen collection procedure-Excision with wire-guided localization" codeSystemName="PATHLEX"/>
<effectiveTime>
<low value="201012150905"/>
<high value="201012150935"/>
</effectiveTime>
<targetSiteCode code="76752008" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED-CT" displayName="Breast structure">
<qualifier>
<name code="272741003" displayName="laterality" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED-CT"/>
<value code="7771000" displayName="left" codeSystem="2.16.840.1.113883.6.96" codeSystemName="SNOMED-CT"/>
</qualifier>
</targetSiteCode>
<performer>
<!--Specimen collector: the surgeon in this case -->
<assignedEntity>
<id/>
<addr nullFlavor="NASK"/>
<telecom value="tel:0147150000" use="EC"/>
<assignedPerson>
<name>
<prefix>Doctor</prefix>
<given>Eva</given>
<family>Surgeon</family>
</name>
</assignedPerson>
<representedOrganization>
<name>Hospital Lullaby</name>
</representedOrganization>
</assignedEntity>
</performer>
<participant typeCode="PRD">
<!--the specimen collected -->
<participantRole classCode="SPEC">
<!--specimen ids -->
<id root="1.3.6.1.4.1.19376.1.8.9.7" extension="0102030405"/>
<playingEntity>
<!--Specimen type -->
<code code="663" codeSystem="1.3.6.1.4.1.19376.1.8.2.1" displayName="Breast-Specimen-Target site- Upper outer quadrant" codeSystemName="PATHLEX"/>
</playingEntity>
</participantRole>
</participant>
</procedure>
</component>
<component>
<!-- Problem organizer -->
<organizer classCode="BATTERY" moodCode="EVN">
<templateId root="1.3.6.1.4.1.19376.1.8.1.4.8"/>
<statusCode code="completed"/>
<component>
<observation classCode="OBS" moodCode="EVN">
<code code=" 33725-3" codeSystemName="LOINC" codeSystem=" 2.16.840.1.113883.6.1" displayName="tumor site"/>
<statusCode code="completed"/>
<value xsi:type="CD" code="663" codeSystem="1.3.6.1.4.1.19376.1.8.2.1" displayName="upper outer quadrant" codeSystemName="PATHLEX"/>
</observation>
</component>
</organizer>
</component>
</organizer>
</entry>
<!-- Sub-sections -->
<component>
<section>
<templateId root="1.3.6.1.4.1.19376.1.5.3.1.3.1"/>
<code code="42349-1" displayName="Reason for referral" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC"/>
<title>Reason for anatomic pathology procedure</title>
<text>Breast mass - left breast</text>
</section>
</component>
<component>
<section>
<templateId root="1.3.6.1.4.1.19376.1.5.3.1.3.4"/>
<code code="10164-2" displayName="History of present illness" codeSystem="2.16.840.1.113883.6.1" codeSystemName="LOINC"/>
<title> History of present illness </title>
<text>Carcinoma of breast. Post operative diagnosis: same. Left UOQ breast mass. </text>
</section>
</component>
<!-- .. -->
</section>
Spezifikation
Die Section MUSS ein Code Element mit folgenden Attributen enthalten:
@code="22636-5" @codeSystem="2.16.840.1.113883.6.1" @displayName="Pathologiebefund Klinische Information"
Die Section MUSS einen narrativen Block enthalten, repräsentiert durch ein Textelement, welches die lesbare Information trägt.
Die Section SOLLTE so viele Specimen Information Organizer Entry Elemente enthalten wie es Specimens oder Gruppen von Specimens gibt. Diese Entries enthalten die maschinenlesbaren Daten korrespondierend zum narrativen Block.
Die Section KANN eine Anzahl von Elementen Component/Section enthalten, welche Subsections mit jeweils besonderen Typen klinischer Informationen einführen.
Die Section SOLLTE ein Autor Element für den Fall enthalten, dass der/die Autoren von denen different sind, die in einem höheren Level im Dokument deklariert sind.
Content Module, die in die Section eingebunden werden sollten
| Name | Typ | Opt. | Card. | Template ID | Quelle | LOINC code |
| Fragestellung | Section | R2 | [0..*] | 1.3.6.1.4.1.19376.1.5.3.1.3.1 | PCC TF-2: 6.3.3.1.1 | 42349-1 |
| Anamnese | Section | R2 | [0..*] | 1.3.6.1.4.1.19376.1.5.3.1.3.4 | PCC TF-2: 6.3.3.2.1 | 10164-2 |
| Aktives Problem | Section | R2 | [0..*] | 1.3.6.1.4.1.19376.1.5.3.1.3.6 | PCC TF-2: 6.3.3.2.3 | 11450-4 |
| Specimen Information Organizer Entry | Entry | R2 | [0..*] | 1.3.6.1.4.1.19376.1.8.1.4.4 | PAT TF-3: 6.2.6.4 | |
| Author of the Section | Child | C | [0..*] | 1.3.6.1.4.1.19376.1.8.1.4.2 | PAT TF-3:6.2.6.2 |
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
SubSection: Fragestellung
Definition und Zweck
Diese optionale Subsection der Klinischen Information enthält eine narrative Beschreibung der Klinischen Fragestellung (des Grundes, weshalb das Material eingesandt wurde).
| Id | 1.2.276.0.76.10.3002 | Gültigkeit | 2017‑02‑01 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Status | Versions-Label | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Name | ReasonForReferral | Bezeichnung | Grund der Überweisung Section | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Kontext | Elternknoten des Template-Element mit Id 1.2.276.0.76.10.3002 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Klassifikation | CDA Section level template | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Offen/Geschlossen | Offen (auch andere als die definierten Elemente sind erlaubt) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Benutzt |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Beziehung | Adaptation: Template 1.3.6.1.4.1.19376.1.5.3.1.3.1 IHE Reason for Referral Section (DYNAMIC) ref IHE-PCC- Adaptation: Template 1.2.40.0.34.11.2.2.1 Aufnahmegrund (DYNAMIC) ref elgabbr- Spezialisierung: Template 2.16.840.1.113883.10.12.201 CDA Section (2005‑09‑07) ref ad1bbr- | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Beispiel |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Beispiel
<!-- Sub-section -->
<component>
<section>
<templateId root= '1.3.6.1.4.1.19376.1.5.3.1.3.1'/>
<code code='42349-1' displayName= ‘Fragestellung’
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title>Fragestellung</title>
<text>Brusttumor- Dignität?</text>
</section>
</component>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Material
Synonyme: Untersuchungsmaterial, Probe, Probenmaterial, Specimen
Definition
Das Konzept lautet: "Untersuchungsmaterial, d. h. bioptisch, operativ oder durch Punktion oder durch Sammeln gewonnenes Gewebs- oder Zellmaterial sowie Körperflüssigkeiten, das zur pathologisch-anatomischen Begutachtung eingesandt wurde. Jedes vom Einsender in separatem Gefäß ("container") übersandtes oder auf dem Untersuchungsantrag separat bezeichnetes Untersuchungsmaterial ("part") ist innerhalb einer Begutachtung auch separat zu behandeln. Die Materialbezeichnung richtet sich nach der des Einsenders.
Das Material spielt eine zentrale Rolle bei der Organisation des gesamten Befundberichtes. Jedes Material ist makroskopisch und mikroskopisch zu beschreiben. Die diagnostische Bewertung kann mehrere Materialien (Gruppen von Specimens) synoptisch behandeln. Zur vertieften Erläuterung dieser Problematik siehe auch cdapath:Child Element Content Module (Template): Specimen Collection Procedure Entry Modul
Der Terminus "Specimen" ist mehrdeutig: Ein physikalisches Objekt (oder eine Ansammlung von Objekten, ist dann ein Specimen, wenn das Labor es als eine diskrete, eindeutig identifizierte Einheit betrachtet, die das Subjekt eines oder mehrerer Schritte im diagnostischen Workflow ist. Damit ist "Specimen" definiert als eine Rolle, die durch ein physikalisches Objekt (oder eine Ansammlung solcher Objekte, die als Einheit betrachtet werden) gespielt wird, wenn das Objekt durch das Labor eindutig identifiziert wird und direkt das Subjekt eines oder mehrerer Schritte im diagnostischen Workflow des Labors ist.
Sowohl ein eingesandtes Resektat, als auch eine Gewebsscheibe daraus, als auch ein Gewebsstück in einem Block, als auch ein Gewebsschnitt ist ein Specimen. Im Laborprozess werden dabei Eigenschaften "vererbt", ein "Child" entsteht aus einem "Parent" durch einen Prozess, das Child ist Produkt des Prozesses.
Specimen Container (oder einfach Container) spielen eine wichtige Rolle in diesem Workflow: In den meisten Prozessschritten werden die Specimen in Containern bearbeitet, teilweise gehen Specimen und Container eine sehr enge Verbindung ein (z.B. Paraffin und Gewebe im Block), manchmal wird der Container Teil des diagnostischen Prozesses (Objektträger, Gewebsschnitt, Eindeckmedium und Deckglas im optischen Strahlengang beim bildgebenden Prozess).
Praktisch haben nur Container Identifikatoren, die für die Prozesssteuerung und teilweise auch beim bildgebenden Prozess (z.B. WSI) unerlässlich sind. Die Identifikatoren der Specimen bleiben meist virtuell, physikalisch nicht zu erfassen. In der Mehrzahl aller Fälle besteht zwischen Specimen und Container eine 1:1 Beziehung; die ID von Specimen und Container sind dann identisch. Es gibt allerdings auch Anwendungsfälle, in denen mehrere Specimen in Beziehung zu einem Container stehen. In diesen Situationen müssen die IDs verschieden sein!
([1]S.40-45) bzw. DICOM Suppl. 122 ([2], S.17 ff)
Organisation nach IHE
Textinformationen zum Material können sich in den Sections "Klinische Information", "Materialaufbereitung"und evtl. "Makroskopische Beschreibung" finden. Die Steuerungsfunktion wird durch die Materialaufbereitungs Section (Procedure Steps section) gewährleistet. In dieser Section werden über den "Specimen Information Organizer" und das "Specimen Collection Procedure template", beides Child Element Content Modules, die hierarchischen Beziehungen zwischen den Specimen auf Case- Part- Block- und Slide-Ebene gemäß CDA-RIM abgebildet.
Das IHE Diagramm veranschaulicht diese Zusammenhänge.
TBD: warten auf aktualisiertes Schema
Organisation nach HL7-D
Die Informationen zum Material werden in der Materialaufbereitungs-Section gesammelt und nicht über den gesamten Befund verstreut. In dieser Section werden auch die zu beschreibenden Probleme aufgeführt und identifiziert. Specimen- und Problembeschreibung könnten durch einen Specimen Information Organizer gebündelt werden
Alle Beziehungen zwischen Beobachtung und Material bzw. Problem werden durch Referenzierung auf die Material-und Problem-ID hergestellt.
Für die Organisationsfunktion des Materials böten sich auch die drei "klassischen" Materialebenen an: Part (Teil), Block, Schnitt, von denen bisher nur der Part als Organisationseinheit für die Befunde genutzt wird. Eine exakte Unterscheidung zwischen Part und Container erfolgt bisher nicht.
Für eine perspektivische Berücksichtigung von Digitalen Bildern (bis zum Virtual Slide) sollte die in IHE-PAT_TF-1 ([3]S.40-45) bzw. DICOM Suppl. 122 ([4], S.17 ff) vorgenommene Analyse der Beziehungen von Specimen und Container unbedingt im Auge behalten und in diesem Leitfaden berücksichtigt werden.
Die dafür notwendigen gestaffelten ID´s von Container und Specimen (Part, Block, Slide) sowie die Specimen Preparation Sequence und Specimen Preparation Step Content Item Sequence müssen vom Pathologiesystem gemäß DICOM Suppl. 122 bereitgestellt werden.
Auf der "Part"-Ebene kann die von ELGA (ELGA_CDA_Laborbefund_2.00_FWGD) vorgeschlagene Strukturierung der Spezimeninformation für Laborbefunde wahrscheinlich zum großen Teil durch das IHE-Konstrukt von Specimen Information Organizer mit dem Specimen Collection Procedure Template realisiert werden:
| Feld | Beschreibung |
|---|---|
| Zeitpunkt des Eintreffens | Zeitpunkt der Materialannahme |
| Specimen Type | Art der Probe (=Materialart) |
| Entnahmeort | Körperstelle / Organ der Materialentnahme |
| Entnahmeart (SpecimenCollectionProcedure - ActCode) | Art der Gewinnung |
| Specimen ID | Material (Part-, Block-, Slide)-ID |
| Entnehmende Person (Performer) | Person, die Materialentnahme durchgeführt hat |
| Kommentar | Kommentar |
| Qualität | Kommentar zur Materialqualität |
Für "Specimen Type", "Entnahmeort" und "Entnahmeart" müssen müssen SNOMED CT Codes aus der "Specimen", "Body Structure" und "Procedure" Achse übernommen werden. Die Unterscheidung zwischen Specimen Type und Entnahmeart ist evtl. tautologisch!! und kann durch einen SNOMED CT Code abgedeckt werden.
Der in Abstimmung befindliche ELGA-Vorschlag zu "Entnahmeort" (procedure target site) ist in folgender Tabelle dargestellt, ergänzt durch SNOMED CT Codes. Diese Darstellung reicht jedoch möglicherweise nicht in jedem Fall aus:
Bei Geweben/Organen, die eine große Ausdehnung haben, z.B. Haut, oder bei denen eine genaue Lokalisation therapieweisend sein könnte, z.B. Brustquadranten, Lebersegmente, etc., muss noch eine genaue Lokalisation hinzugegeben werden, die auf Freitext beschränkt bleiben sollte. Außerdem gehört hier auch bei paarigen Organen die Lateralitätsangabe hinzu. Die Darstellung des "Entnahmeortes" muss also durch weitere Qualifier der Body Structure behandelt werden.
| Material-Hauptkategorie | SNOMED CT | Material-Subkategorie | SNOMED CT |
|---|---|---|---|
| Bewegungsapparat / Weichgewebe | 309072003 | Bewegungsapparat, allgemein | 309104000 |
| Muskel | 119331003 | ||
| Knochen | 430268003 | ||
| Sehnen, Bänder, Gelenke | 309107007 | ||
| Weichgewebe allgemein | 309072003 | ||
| Brustdrüse / Axilla | Brustdrüse / Axilla | ||
| Haut / Subcutis | Haut / Subcutis, allgemein | ||
| Hämatopoetisches+Lymphatisches System | Hämatopoetisches System, allgemein | ||
| Blut | |||
| Thymus | |||
| Tonsille/Adenoide | |||
| Knochenmark | |||
| Lymphatisches System, allgemein | |||
| Lymphknoten | |||
| Milz | |||
| etc. |
|
|
GH:in HL7 v26_appendix_a.pdf (Data definition tables) werden Kapitel 17.4.2 und 17.4.7 referenziert, die ich bisher nicht finden konnte! |
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Section: Material und Materialaufbereitung (Procedure Steps)
| Template-Metadaten | |
| Template-Typ | Section |
| Template ID | 1.3.6.1.4.1.19376.1.8.1.2.6 |
| generischeres Template | |
| genutztes Templates | keine |
| nutzende Templates | |
| abgeleitete Templates | |
| Schwester-Templates | |
| Generelle Beschreibung | In diesem Abschnitt werden die Informationen zur Materialaufbereitung übermittelt. |
| allg. Erläuterung | vom Sektorkommitee Pathologie/Neuropathologie gefordert |
| Verhältnis zu IHE | dt.Übersetzung oder Ergänzung oder neu |
| Ballotierungsstatus | in Arbeit |
| Erweiterbarkeit | geschlossen |
Definition und Zweck
Diese Section beschreibt die Proben (Material, Specimens), die übersandt wurden, die Methode ihrer Gewinnung und ihre Herkunft (Specimen collection) und die Materialaufbereitung (Specimen processing): Repräsentative Proben, Schritte des Workflow (Trimming, Embedding, Cutting, Staining) und davon abgeleitete Gewebsproben für weitere Untersuchungstechniken (Flowzytometrie, zytogenetische und molekularpathologische Untersuchungen, Elektronenmikroskopie etc.) oder Biobanken. Diese Informationen werden in einem Specimen Information Organizer Entry organisiert (IHE APSR evolution 2014). Alternativ werden alle Informationen der Specimen collection procedure und Specimen processing procedures in einem Specimen Collection Procedure Entry Modul sowie alle diagnostischen Probleme, die während der Untersuchung der Specimens oder Gruppen von Specimens relevant geworden sind,in einem Problem organizer Entry Modul aufgelistet.
Erläuterungen
Synonyme: Untersuchungsmaterial, Probe, Probenmaterial, Specimen
Das zugrundeliegende Konzept lautet: "Untersuchungsmaterial, d. h. bioptisch, operativ oder durch Punktion oder durch Sammeln gewonnenes Gewebs- oder Zellmaterial sowie Körperflüssigkeiten, das zur pathologisch-anatomischen Begutachtung eingesandt wurde. Jedes vom Einsender in separatem Gefäß ("container") übersandtes oder auf dem Untersuchungsantrag separat bezeichnetes Untersuchungsmaterial ("part") ist innerhalb einer Begutachtung auch separat zu behandeln. Die Materialbezeichnung richtet sich nach der des Einsenders.
Das Material spielt eine zentrale Rolle bei der Organisation des gesamten Befundberichtes. Jedes Material ist makroskopisch und mikroskopisch zu beschreiben. Die diagnostische Bewertung kann mehrere Materialien (Gruppen von Specimens) synoptisch behandeln. Zur vertieften Erläuterung dieser Problematik siehe auch cdapath:Child Element Content Module (Template): Specimen Collection Procedure Entry Modul
Der Terminus "Specimen" ist mehrdeutig: Ein physikalisches Objekt (oder eine Ansammlung von Objekten, ist dann ein Specimen, wenn das Labor es als eine diskrete, eindeutig identifizierte Einheit betrachtet, die das Subjekt eines oder mehrerer Schritte im diagnostischen Workflow ist. Damit ist "Specimen" definiert als eine Rolle, die durch ein physikalisches Objekt (oder eine Ansammlung solcher Objekte, die als Einheit betrachtet werden) gespielt wird, wenn das Objekt durch das Labor eindutig identifiziert wird und direkt das Subjekt eines oder mehrerer Schritte im diagnostischen Workflow des Labors ist.
Sowohl ein eingesandtes Resektat, als auch eine Gewebsscheibe daraus, als auch ein Gewebsstück in einem Block, als auch ein Gewebsschnitt ist ein Specimen. Im Laborprozess werden dabei Eigenschaften "vererbt", ein "Child" entsteht aus einem "Parent" durch einen Prozess, das Child ist Produkt des Prozesses.
Specimen Container (oder einfach Container) spielen eine wichtige Rolle in diesem Workflow: In den meisten Prozessschritten werden die Specimen in Containern bearbeitet, teilweise gehen Specimen und Container eine sehr enge Verbindung ein (z.B. Paraffin und Gewebe im Block), manchmal wird der Container Teil des diagnostischen Prozesses (Objektträger, Gewebsschnitt, Eindeckmedium und Deckglas im optischen Strahlengang beim bildgebenden Prozess).
Praktisch haben nur Container Identifikatoren, die für die Prozesssteuerung und teilweise auch beim bildgebenden Prozess (z.B. WSI) unerlässlich sind. Die Identifikatoren der Specimen bleiben meist virtuell, physikalisch nicht zu erfassen. In der Mehrzahl aller Fälle besteht zwischen Specimen und Container eine 1:1 Beziehung; die ID von Specimen und Container sind dann identisch. Es gibt allerdings auch Anwendungsfälle, in denen mehrere Specimen in Beziehung zu einem Container stehen. In diesen Situationen müssen die IDs verschieden sein!
([5]S.40-45) bzw. DICOM Suppl. 122 ([6], S.17 ff)
Organisation nach IHE APSR
Textinformationen zum Material können sich in den Sections "Klinische Information", "Materialaufbereitung"und evtl. "Makroskopische Beschreibung" finden.
Die Steuerungsfunktion wird durch die Materialaufbereitungs Section (Procedure Steps section) gewährleistet. In dieser Section werden unter dem "Specimen procedure step entry", ein Child Element Content Module, die hierarchischen Beziehungen zwischen den Specimen auf Case- Part- Block- und Slide-Ebene gemäß CDA-RIM über entryRelationships abgebildet, indem die Specimens einer Hierarchie-Ebene jeweils als Produkt der prozedur der übergeordneten Hierarchie-Ebene beschrieben werden.
Das IHE Diagramm veranschaulicht diese Zusammenhänge.
Abb.: Entries der Specimen Procedure Step Section
Alternativ könnte auf das entryRelationship-Konstrukt verzichtet werden, indem für jedes Specimen neben seiner eineindeutigen ID noch die ID seines Ausgangsproduktes (parentID) angegeben wird (gemäß V3_DAM_SPECIMEN_R1_I1_2014MAY).
Für die Organisationsfunktion des Materials bieten sich die drei "klassischen" Materialebenen an: Part (Teil), Block, Schnitt, von denen bisher nur der Part als Organisationseinheit für konventionelle Befunde genutzt wird. Eine exakte Unterscheidung zwischen Specimen und Container erfolgt bisher nicht, ist aber für Spezialfälle (Tissue Micro Arrays - TMA, on-slide-Kontrollen, Serienschnitte) unabdingbar.
Für eine perspektivische Berücksichtigung von Digitalen Bildern (bis zum Virtual Slide) sollte die in IHE-PAT_TF-1 ([7]S.40-45) bzw. DICOM Suppl. 122 ([8], S.17 ff) vorgenommene Analyse der Beziehungen von Specimen und Container unbedingt in diesem Leitfaden berücksichtigt werden.
Die dafür notwendigen gestaffelten ID´s von Container und Specimen (Part, Block, Slide) sowie die Hierarchie der Specimens und Container einschl. der jeweiligen Herkunft und der Prozeduren, die zum Specimen geführt haben (im DICOM Modell die Specimen Preparation Sequence und Specimen Preparation Step Content Item Sequence) müssen vom Pathologiesystem gemäß DICOM Suppl. 122 bereitgestellt werden.
Alle Beziehungen zwischen Beobachtung und Specimen werden durch Referenzierung auf die Specimen-ID hergestellt.
Die auf der "Part"-Ebene von ELGA (ELGA_CDA_Laborbefund_2.00_FWGD) vorgeschlagene Strukturierung der Spezimeninformation für Laborbefunde ist nicht zum IHE-Konstrukt der Procedure Steps Section kompatibel:
| Feld | Beschreibung |
|---|---|
| Zeitpunkt des Eintreffens | Zeitpunkt der Materialannahme |
| Specimen Type | Art der Probe (=Materialart) |
| Entnahmeort | Körperstelle / Organ der Materialentnahme |
| Entnahmeart (SpecimenCollectionProcedure - ActCode) | Art der Gewinnung |
| Specimen ID | Material (Part-, Block-, Slide)-ID |
| Entnehmende Person (Performer) | Person, die Materialentnahme durchgeführt hat |
| Kommentar | Kommentar |
| Qualität | Kommentar zur Materialqualität |
Der in Abstimmung befindliche ELGA-Vorschlag zu "Entnahmeort" (procedure target site) ist in folgender Tabelle dargestellt, ergänzt durch SNOMED CT Codes. Diese Darstellung reicht jedoch möglicherweise nicht in jedem Fall aus:
Bei Geweben/Organen, die eine große Ausdehnung haben, z.B. Haut, oder bei denen eine genaue Lokalisation therapieweisend sein könnte, z.B. Brustquadranten, Lebersegmente, etc., muss noch eine genaue Lokalisation hinzugegeben werden, die auf Freitext beschränkt bleiben sollte. Außerdem gehört hier auch bei paarigen Organen die Lateralitätsangabe hinzu. Die Darstellung des "Entnahmeortes" muss also durch weitere Qualifier der Body Structure behandelt werden.
| Material-Hauptkategorie | SNOMED CT | Material-Subkategorie | SNOMED CT |
|---|---|---|---|
| Bewegungsapparat / Weichgewebe | 309072003 | Bewegungsapparat, allgemein | 309104000 |
| Muskel | 119331003 | ||
| Knochen | 430268003 | ||
| Sehnen, Bänder, Gelenke | 309107007 | ||
| Weichgewebe allgemein | 309072003 | ||
| Brustdrüse / Axilla | Brustdrüse / Axilla | ||
| Haut / Subcutis | Haut / Subcutis, allgemein | ||
| Hämatopoetisches+Lymphatisches System | Hämatopoetisches System, allgemein | ||
| Blut | |||
| Thymus | |||
| Tonsille/Adenoide | |||
| Knochenmark | |||
| Lymphatisches System, allgemein | |||
| Lymphknoten | |||
| Milz | |||
| etc. |
|
|
GH:in HL7 v26_appendix_a.pdf (Data definition tables) werden Kapitel 17.4.2 und 17.4.7 referenziert, die ich bisher nicht finden konnte! |
Attribute
|
|
GH 20141227:Abstimmung mit aktuellen Entwicklungen auf ArtDecor notwendig! |
Diese Section muss ein Code Element enthalten, das mit folgenden Attributen gefüllt ist:
| Lvl | RIM | Name | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|
| 1 | act | section | 1..1 | M | ||
| 2 | act | templateId | 1..1 | F | @root= 1.3.6.1.4.1.19376.1.8.1.2.xxx | |
| 2 | act | code | CD | 1..1 | F | @code= "68992-7" @codeSystem= "2.16.840.1.113883.6.1" |
| 2 | act | title | 1..1 | R | ||
| 2 | act | text | 1..1 | R | Diese Section soll ein Text Element enthalten, welches die Information zur Materialaufbereitung lesbar enthält. | |
| 2 | act | entry | 1..1 | M | ||
| 3 | act | templateId | 1..1 | F | @root= 1.3.6.1.4.1.19376.1.8.1.4.4 | |
| 3 | act | title | 1..1 | R | Specimen Information Organizer | |
| 2 | act | entry | 1..1 | M | (alternativ zu Specimen Information Organizer) | |
| 3 | act | templateId | 1..1 | F | @root= 1.3.6.1.4.1.19376.1.3.1.2 | |
| 3 | act | title | 1..1 | R | Specimen collection procedure | |
| 2 | act | entry | 1..1 | M | (alternativ zu Specimen Information Organizer) | |
| 3 | act | templateId | 1..1 | F | @root= 1.3.6.1.4.1.19376.1.8.1.4.8 | |
| 3 | act | title | 1..1 | R | Problem organizer | |
| 2 | part | 0..1 | O | |||
| 3 | role | author | 1..1 | R | Diese Section sollte ein Autor Element enthalten, wenn der Autor dieser Section von dem in höheren Ebenen des Dokuments verschieden ist. | |
| 4 | ent | Person | 0..1 | R | Arzt |
Beispiel:
Die Informationsstruktur kann infolge der Hierarchie von Einsendung, Parts, Blocks und Slides sehr komplex werden. Als ein konkretes Beispiel soll das der xml-Instanz im Anhang A dienen:
Einsendung (Specimen): Zystoprostatektomiepräparat
a) specimen collection method:
a1) radical cystoprostatectomy (procedure), for all
b) specimen type:
b1) specimen from urinary bladder obtained by radical cystectomy (specimen)
b2) specimen from prostate obtained by radical prostatectomy (specimen)
b3) Tissue specimen from urethra (specimen)
b4) specimen obtained by marginal resection (specimen)
c) specimen collection source site:
c1) prostate and urinary bladder combinated site (body structure), for b1), b2)
c2) Urethra part (body structure), for b3), b4)
d) specimen treatmentProcedure:
d1) Tissue processing technique, routine, embed, cut and stain, per surgical specimen (procedure), for b1) through b3)
d2) Tissue frozen section technique, complete (procedure), for b4)
Vom Pathologiesystem werden für jedes dieser Items separate IDs vergeben, die diese Hierarchie wiedergeben können, aber nicht müssen. Im APSR finden diese IDs Verwendung bei der Referenzierung der AP observations. Jede ID muss eineindeutig die Charakteristika des jeweiligen Specimens rückverfolgbar machen.
Das nachfolgende XML-Beispiel drückt das noch nicht aus!
<section>
<templateId root='1.3.6.1.4.1.19376.1.8.1.2.99'/>
<code code='46059-2' displayName='Special treatments and procedures section'
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title>PROCEDURE STEPS</title>
<text>
Probe 1 (bezeichnet mit “Mammabiopsie links”) wird für Gefrierschnitt aufgearbeitet.
Restgewebe für Paraffinhistologie.
Probe 2 (bezeichnet als “ax.Fettgewebe apikal”), in zwei Teilen übersandt, wird vollständig gebettet und mit zahlreichen Schnittstufen untersucht.
</text>
<!-- Procedure steps entry -->
<entry typeCode="" contextConductionInd="false">
<procedure negationInd="false" classCode="PROC" moodCode="EVN">
<templateId root="2.16.840.1.113883.10.12.306"/>
<templateId root="2.16.840.1.113883.3.1937.99.61.5.10.900283"/>
<id root="1.2.3.999" extension="--example only--"/>
<code code="17636008" codeSystem="2.16.840.1.113883.6.96" displayName="Specimen collection (procedure)"/>
<text/>
<statusCode code="normal"/>
<effectiveTime><low value="20141226122646"/></effectiveTime>
<methodCode/>
<targetSiteCode/>
<specimen><!-- template 2.16.840.1.113883.10.12.322 (dynamic) --></specimen>
<performer><!-- template CDAPerformerBody (dynamic) --></performer>
<author><!-- template CDAAuthorBody (dynamic) --></author>
<participant><!-- include template 2.16.840.1.113883.3.1937.99.61.5.10.900284 (dynamic) 1..1 M--></participant>
</procedure>
</entry>
</section>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Section:Färbungen
| Template-Metadaten | |
| Template-Typ | Section |
| Template ID | |
| generischeres Template | |
| genutztes Templates | |
| nutzende Templates | |
| abgeleitete Templates | |
| Schwester-Templates | |
| generelle Beschreibung | |
| allg. Erläuterung | |
| Verhältnis zu IHE | neu |
| Ballotierungsstatus | in Arbeit |
| Erweiterbarkeit | offen |
|
|
falsche Überschrift???? Vgl. nachfolgenden Text ... |
Immunhistochemische Färbungen
Die Informationen zu immunhistochemischen Färbungen bestehen aus folgenden Informationen:
| Bedeutung | Datentyp | OID |
|---|---|---|
| Antikörper (Kurzbezeichnung) | Code oder String?? | 2.16.840.1.113883.6.1 |
| Klon | String | 1.2.276.0.76.5.???? |
| Hersteller | Code oder String?? | 1.2.276.0.76.5.???? |
| Antikörperklasse | CD | ???? |
| Protokoll-ID | II | 1.2.276.0.76.5.???? |
| Färbeintensität | CD | 1.2.276.0.76.5.???? |
| Färbemuster | CD | 1.2.276.0.76.5.???? |
| Verteilungsmuster | CD | 1.2.276.0.76.5.???? |
| Anteil positiver Zellen | PQ | ?? |
| Gewebetyp | CD | 1.2.276.0.76.5.???? |
| Färbeergebnis | CD | 1.2.276.0.76.5.???? |
| Fixierung | CD | 1.2.840.10008.???? |
| Bildanalyseprogramm | String | 1.2.276.0.76.5.???? |
| Score-Typ | vgl. Scores & Assessment DSTU | |
| Score-Ergebnis | vgl. Scores & Assessment DSTU |
Tabelle 4: Färbungen
Entry:Immunhistologie Mikroskopie
| Template-Metadaten | |
| Template-Typ | Entry |
| Template ID | tbd |
| genutztes Template | |
| abgeleitete Templates | |
| Generelle Beschreibung | Hiermit wird auf Entry-Level die Übermittlung von Ergebnissen und Prozessdaten der Immunhistochemie definiert. |
| allg. Erläuterung | < Verweis auf eine allgemeine Erläuterungsseite > |
| Ballotierungsstatus | in Ballotierung |
| Erweiterbarkeit | geschlossen |
Code: @code="55229-9" @codeSystem="2.16.840.1.113883.6.1"
Value Set (OID TODO - Code System ?):
Definition und Zweck
Das Immunhistologie Mikroskopie <entry>Content Modul ist ein generisches Template für den strukturierten Pathologiebericht. Es enthält in maschinenlesbarer Form die Informationen bezüglich der Prozess- und Ergebnisdaten immunhistochemischer Färbungen eines Materials (Specimen) oder Gruppe von Materialien. Es ist in das Mikroskopie <section> Content Modul verschachtelt. Es bündelt eine Reihe von AP Observations und Prozedurelemente.
Die Informationen zu immunhistochemischen Färbungen bestehen aus folgenden Informationen:
| Bedeutung | Verw. | Kardin. | HL7 | Datentyp | OID |
|---|---|---|---|---|---|
| Antikörper (Kurzbezeichnung) | R | [1..1] | method / substance? | CD | 2.16.840.1.113883.6.1 ???? /http://www.antibodies-online.com/antibody/ |
| Klon | R | [1..1] | method / substance? | CD | 1.2.276.0.76.5.???? / http://www.antibodies-online.com/antibody/ |
| Hersteller | R | [1..1] | @id Entity | ?? | 1.2.276.0.76.5.???? |
| Antikörperklasse | R | [1..1] | method / substance? | CD | ???? |
| Protokoll-ID | R2 | [0..1] | Instanz ID | II | 1.2.276.0.76.5.???? |
| Färbeintensität | R | [1..1] | AP observation | CD | evtl. Scores & Assessment DSTU, SNOMED-CT 444775005 |
| Färbemuster | O | [0..1] | AP observation | CD | evtl.. Scores & Assessment DSTU |
| Verteilungsmuster | O | [0..1] | AP observation | CD | evtl. Scores & Assessment DSTU |
| Anteil positiver Zellen | O | [0..1] | AP observation | PQ | |
| Gewebetyp | R | [1..1] | AP observation | CD | 1.2.276.0.76.5.???? |
| Färbeergebnis | O | [0..1] | AP observation | CD | 1.2.276.0.76.5.???? |
| Fixierung | O | [0..1] | method / substance? | CD | 1.2.840.10008.???? |
| Bildanalyseprogramm | O | [0..1] | Instanz ID | II | 1.2.276.0.76.5.???? |
| Score-Typ | O | [0..1] | AP observation | CD | vgl. Scores & Assessment DSTU, SNOMED-CT 278061009 oder 246262008 |
| Score-Ergebnis | O | [0..1] | AP observation | CD | vgl. Scores & Assessment DSTU |
Tabelle 4: Informationen zu immunhistochemischen Färbungen
Text-Beispiel
Nachfolgend ein Beispiel für eine Reihe immunhistochemischer Reaktionen (einschl. von Positiv- und Negativkontrollen) in der Text-Darstellung:
| Anti- körper | Klon | Her- steller | AK- Klasse | Proto- koll- ID | Reak- tions-stärke | Färbe- muster | Vertei- lungs- muster | %pos. Zellen | Gewebe- typ | Färbe- er- gebnis | Fixie- rung | Bild- analyse | Score- Typ | Score-Ergebnis |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Ki67 | 30-9 | Ventana | 2 | xyz | stark | nukleär | diffus | 9 | Tumor | positiv | FFPE | Immuno- Ratio | ||
| Ki67 | 30-9 | Ventana | 2 | xyz | keine | keine | keine | 0 | neg.Färbe- kontrolle | negativ | FFPE | |||
| CK5/6 | D5/16B4 | DAKO | 1 | uvw | mittel | membran- st. komplett | basal | Tumor | positiv | FFPE | ||||
| ER/PR pharmDX | 1D5 / ER 2-123 | DAKO | 3 | abc | stark | nukleär | diffus | 87 | Tumor | positiv | FFPE | Immuno- Ratio | Rem- mele | endokrin responsiv |
| ER/PR pharmDX | 1D5 / ER 2-123 | DAKO | 3 | abc | stark | nukleär | diffus | 95 | ext. Positiv on-slide-kontrolle |
positiv | FFPE | Immuno- Ratio | ||
| ER/PR pharmDX | 1D5 / ER 2-123 | DAKO | 3 | abc | mittel | nukleär | fokal | 30 | int. Positiv kontrolle |
positiv | FFPE | Immuno- Ratio | ||
| ER/PR pharmDX | 1D5 / ER 2-123 | DAKO | 3 | abc | keine | keine | keine | 0 | neg.Färbe kontrolle |
negativ | FFPE | |||
| ER/PR pharmDX | PgR 1294 | DAKO | 3 | def | mittel | nukleär | diffus | 38 | Tumor | positiv | FFPE | Immuno- Ratio | Rem- mele | endokrin responsiv |
| ER/PR pharmDX | PgR 1294 | DAKO | 3 | def | keine | keine | keine | 0 | neg.Färbe kontrolle |
negativ | FFPE |
Abbildung in CDA
Kommentar: muss noch an Textbeispiel angepasst werden.
<section>
<!-- Darstellung als Tabelle -->
<text>
<tbody>
<tr>
<th>Antikörper</th>
<th>Klon</th>
<th>Hersteller</th>
<th>AK-Klasse</th>
<th>Protokoll-ID</th>
<th>Färbeintensität</th>
<th>Färbemuster</th>
<th>Verteilungsmuster</th>
<th>% pos. Zellen</th>
<th>Gewebetyp</th>
<th>Färbeergebnis</th>
<th>Fixierung</th>
<th>Score-Typ</th>
<th>Score-Ergebnis</th>
</tr>
<tr>
<td><content ID="d1">CK5/14</content></td>
<td><content ID="d2">D5/16B4</content></td>
<td><content ID="d3">DAKO</content></td>
<td><content ID="d4"> 1</content></td>
<td><content ID="d5">mittel</content></td>
<td><content ID="d6">membranständig-komplett</content></td>
<td><content ID="d7">basal</content></td>
<td><content ID="d8"></content></td>
<td><content ID="d9">Tumor</content></td>
<td><content ID="d10">positiv</content></td>
<td><content ID="d11">FFPE</content></td>
</tr>
...
</tbody>
</text>
<!-— erste Information -->
<entry typeCode="DRIV">
<observation classCode="OBS" moodCode="EVN">
<code code="68498002"
codeSystem="2.16.840.1.113883.6.96"
displayName="Antikörper(kurz)" />
<value xsi:type="CD" code="50315-1" codeSystem="2.16.840.1.113883.6.1">
<originalText><reference value="#d1"/></originalText>
</value>
</observation>
</entry>
<!-— zweite Information -->
<entry typeCode="DRIV">
<observation>
<code code="47308002"
codeSystem="2.16.840.1.113883.6.96"
displayName="Antikörper Klon" />
<value xsi:type="CD" code="??" codeSystem="????">
<originalText><reference value="#d2"/></originalText>
</value>
</observation>
</entry>
<!-— dritte Information -->
<entry typeCode="DRIV">
<observation classCode="OBS" moodCode="EVN">
<code code="xxxx"
codeSystem="a.b.c.dx.y.z"
displayName="Antikörper Hersteller" />
<value xsi:type="CD" code="?" codeSystem="????">
<originalText><reference value="#d3"/></originalText>
</value>
</observation>
</entry>
<!-— vierte Information -->
<entry typeCode="DRIV">
<observation classCode="OBS" moodCode="EVN">
<code code="xxxx"
codeSystem="a.b.c.dx.y.z"
displayName="Antikörper Klasse" />
<value xsi:type="CD" code="1" codeSystem="????">
<originalText><reference value="#d4"/></originalText>
</value>
</observation>
</entry>
<!-— fünfte Information -->
<entry typeCode="DRIV">
<observation classCode="OBS" moodCode="EVN">
<code code="444775005"
codeSystem="2.16.840.1.113883.6.96"
displayName="Färbeintensität" />
<value xsi:type="CD" code="2" codeSystem="????">
<originalText><reference value="#d5"/></originalText>
</value>
</observation>
</entry>
<!-— sechste Information -->
<entry typeCode="DRIV">
<observation classCode="OBS" moodCode="EVN">
<code code="246461003"
codeSystem="2.16.840.1.113883.6.96"
displayName="Färbemuster" />
<value xsi:type="CD" code="3" codeSystem="????">
<originalText><reference value="#d6"/></originalText>
</value>
</observation>
</entry>
<!-— siebte Information -->
<entry typeCode="DRIV">
<observation classCode="OBS" moodCode="EVN">
<code code="xxxx"
codeSystem="a.b.c.dx.y.z"
displayName="Verteilungsmuster" />
<value xsi:type="CD" code="3" codeSystem="????">
<originalText><reference value="#d7"/></originalText>
</value>
</observation>
</entry>
...
</section>
Value sets für Immunhistologie
Antikörper
Bisher keine geeignete Klassifikation gefunden. Neutrale Übersicht in http://www.antibodies-online.com/antibody/
Die LOINC Codes in untenstehender Tabelle beschreiben das Färbeergebnis im Gewebe, nicht den Antikörper!!
| Code | Antikörper | Bedeutung |
|---|---|---|
| 40563-9 | Aktin(glattmuskulär)) | reagiert mit Aktin in glatter Muskulatur |
| 10463-8 | Amyloid A | reagiert mit Amyloid vom Typ AA |
| ???? | CD45 | reagiert mit LCA |
| u.s.w. |
Tabelle 6: Antikörper (OID: 2.16.840.1.113883.6.1)
Antikörperklone
Bisher keine geeignete Klassifikation gefunden. Neutrale Übersicht in http://www.antibodies-online.com/antibody/
| Klon (Code) | Bedeutung |
|---|---|
| 1A4 | monoklonaler Maus-AK gegen glattmuskuläres Aktin |
| mc1 | monoklonaler Maus-AK gegen Amyloid vom Typ AA |
| 2B11 | monoklonaler Maus-AK gegen LCA |
| PD7/26 | monoklonaler Maus-AK gegen LCA |
| u.s.w. |
Tabelle 7: Klone monoklonaler Antikörper (OID: 1.2.276.0.76.5.????)
Antikörperhersteller
| OID | Hersteller |
|---|---|
| 1.3.6.1.4.1.11987 | DAKO |
| 2.16.840.1.113995 | Roche |
| ???? | Zytomed |
| 1.3.6.1.4.1.22868 | Ventana |
| 1.3.6.1.4.1.492 | DCS |
| u.s.w. |
Tabelle 8: Hersteller von Antikörpern (OID: 1.2.276.0.76.5.????)
Die Hersteller werden über die Klasse Entity mit @id identifiziert. Insofern wird diese Tabelle so nicht benötigt.
Antikörperklasse
Bisher keine geeignete Klassifikation gefunden. Neutrale Übersicht in Sektorkomitee Pathologie
| Code | Antikörperklasse | Bedeutung |
|---|---|---|
| 1 | Klasse I | AK für diagnostische Immunhisto-/-zytochemie,
die im Kontext mit Morphologie und klinischen Daten interpretiert werden. Sie dienen der Zelldifferenzierung. |
| 2 | Klasse II | AK für diagnostische Immunhisto-/-zytochemie mit
direkter Therapierelevanz, die als Einzelergebnis semiquantitativ bewertet werden. |
| 3 | CE | AK mit CE-Kennzeichnung.
Die Validierungsdaten wurden vom Hersteller erhoben. |
Tabelle 9: Antikörperklasse (OID: ????)
Protokoll der Färbung
| Protokoll-ID | |
|---|---|
| xyz | |
| abc | |
| u.s.w. |
Tabelle 10: ID des verwendeten Protokolls im Immunfärbeautomaten (OID: 1.2.276.0.76.5.????)
Woraus ergibt sich diese Information? Wird die generiert? Dann ist es eine Instanz-ID!
GH: Wird vom Färbeautomaten via Pathologiesystem geliefert (wenn vorhanden)
Färbeintensität
| Code | Färbeintensität | Bedeutung |
|---|---|---|
| 0 | keine | keine Reaktion |
| 1 | schwach | schwache Reaktion (im Vergleich zur Positivkontrolle) |
| 2 | mittel | mäßig starke Reaktion (im Vergleich zur Positivkontrolle) |
| 3 | stark | starke Reaktion (im Vergleich zur Positivkontrolle) |
Tabelle 11: Färbeintensität (OID: 1.2.276.0.76.5.????)
Färbemuster
| Code | Färbemuster | Bedeutung |
|---|---|---|
| 0 | keine Färbung | keine Reaktion |
| 1 | nukleär | Kerne gefärbt |
| 2 | zytoplasmatisch | Zytoplasma gefärbt |
| 3 | membranständig_komplett | Zellmembran vollständig gefärbt |
| 4 | membranständig_partiell | Zellmembran teilweise gefärbt |
| 5 | Kombination aus 1-3 oder 4 | mehrere Zellstrukturen gefärbt |
Tabelle 12: Färbemuster(OID: 1.2.276.0.76.5.????)
Verteilungsmuster der gefärbten Objekte
| Code | Verteilungsmuster | Bedeutung |
|---|---|---|
| 0 | keine Färbung | keine Objekte gefärbt |
| 1 | diffus | homogene Verteilung der gefärbten Objekte |
| 2 | fokal | Objekte nur herdförmig gefärbt |
| 3 | basal | Basalzellschicht gefärbt |
| 4 | luminal | lumenseitige Zellschicht gefärbt |
| 5 | mosaik | Mosaikmuster gefärbter Objekte im Verband |
Tabelle 13: Verteilungsmuster der gefärbten Objekte (OID: 1.2.276.0.76.5.????)
Anteil gefärbter Objekte
| Anteil positiver Objekte | Bedeutung |
|---|---|
| Anteil gefärbter Objekte an allen Objekten gleicher Art in % |
Tabelle 14: Anteil positiver Zellen (OID: 1.2.276.0.76.5.????)
Sollte als physical quantity übermittelt werden.
Gewebetyp
| Code | Gewebetyp | Bedeutung |
|---|---|---|
| 1 | Normalgewebe | nichtpathologisch verändertes Gewebe (in der Umgebung der Läsion) |
| 2 | Tumorgewebe | Tumorgewebe (Läsion) |
| 3 | Zellinie | Zelllinie mit definiertem Reaktionsausfall |
| 4 | Positive Gewebskontrolle (extern) | Als geeignet bekanntes Läsionsgewebe, nichtdiagnostisch, mit definiertem positiven Reaktionsausfall |
| 5 | Positive Gewebskontrolle (intern) | Läsionsgewebe, diagnostisch, mit erwartetem definierten positiven Reaktionsausfall |
| 6 | Negative Gewebskontrolle (intern) | Läsionsgewebe, diagnostisch, mit erwartetem definierten negativen Reaktionsausfall |
| 7 | Negative Färbekontrolle | Läsionsgewebe, diagnostisch, mit definiertem positiven Reaktionsausfall, ohne Primärantikörper |
| 8 | Externe Gewebskontrolle (separat) | Als geeignet bekanntes Läsionsgewebe, nichtdiagnostisch, auf separatem Objektträger, mit definiertem positiven und/oder negativen Reaktionsausfall |
| 9 | Externe Gewebskontrolle (on-slide) | Als geeignet bekanntes Läsionsgewebe, nichtdiagnostisch, auf diagnostischem Objektträger, mit definiertem positiven und/oder negativen Reaktionsausfall |
Tabelle 15: Gewebetypen (OID: 1.2.276.0.76.5.????)
Färbeergebnis
| Code | Färbeergebnis | Bedeutung |
|---|---|---|
| 0 | negativ | keine diagnostische Färbung |
| 1 | fraglich positiv | unsichere diagnostische Färbung |
| 2 | positiv | diagnostische Färbung |
| 9 | nicht auswertbar | diagnostisch nicht verwertbar |
Tabelle 16: Färbeergebnis (OID: 1.2.276.0.76.5.????)
Fixierung, Vorbehandlung und Einbettung
Hierzu gibt es drei Codesysteme von DICOM, suppl. 122
| CodingSchemeDesignator | Wert | Bedeutung |
|---|---|---|
| SRT | ?? | frisches oder gefrorenes Gewebe |
| SRT | C-2141C | neutral gepuffertes Formalin |
| SRT | ?? | andere Fixations- und/oder Einbettungsverfahren |
Tabelle 17a: Fixierung (OID: 1.2.840.10008.2.16.4.?? oder 1.2.840.10008.??, Context ID 8114)
| CodingSchemeDesignator | Wert | Bedeutung |
|---|---|---|
| SRT | P3-40005 | Mikrowelle |
| SRT | P3-40009 | Dampfkocher |
| SRT | P3-40006 | Proteaseverdauung |
| SRT | P3-4000B | Dehydrierung |
| SRT | P3-05050 | Frosten |
| SRT | P3-40003 | Clearing |
Tabelle 17b: Vorbehandlung(OID: 1.2.840.10008.2.16.4.?? oder 1.2.840.10008.??, Context ID 8113)
| CodingSchemeDesignator | Wert | Bedeutung |
|---|---|---|
| SRT | F-616D8 | Paraffin |
| SRT | F-62232 | Gefriermedium |
| SRT | C-2A000 | Plastic |
| SRT | C-84085 | Agar |
| SRT | C-2A400 | Epoxid |
| SRT | C-100EA | Acrylat |
Tabelle 17c: Einbettung(OID: 1.2.840.10008.2.16.4.?? oder 1.2.840.10008.??, Context ID 8115)
Bildanalyseprogramm
| Programmname | Hersteller | Verwendungszweck |
|---|---|---|
| ImmunoRation | http://imtmicroscope.uta.fi/immunoratio | Quantifizierung von Hormonrezeptorexpression und Ki-67-Expression |
| ImmunoMembrane | http://imtmicroscope.uta.fi/immunomembrane | Quantifizierung der Her-2-neu-Rezeptorexpression |
| ACIS III | DAKO | Antigenquantifizierung für pharmDX Kits |
| VIAS | Ventana/Roche | Quantifizierung des Ventana/Roche Breast Panel |
| u.s.w. |
Tabelle 18: Bildanalyseprogramme (OID: 1.2.276.0.76.5.????)
Score-Typ
| Code | Name | Bedeutung |
|---|---|---|
| 1 | Remmele-Stegner | Hormonrezeptorexpression |
| 2 | Allred | Hormonrezeptorexpression |
| 3 | Her-2-neu-Brust | Her-2-neu-Expression bei Mammakarzinom |
| 4 | Her-2-neu-Magen | Her-2-neu-Expression bei Magenkarzinom |
| u.s.w. |
Tabelle 19: Score-Typ (OID: 1.2.276.0.76.7.2.????)
FO: Scores und die Ergebnisse sollten gemäß Scores und Assessment DSTU übermittelt werden!
Score-Ergebnis
| Score-Code | Ergebnis | Bedeutung |
|---|---|---|
| 1 | 0 | endokrin nicht responsiv |
| 1 | 1 | endokrin fraglich responsiv |
| 1 | 2-12 | endokrin responsiv |
| 3 | 0 und 1+ | keine Überexpression |
| 3 | 2+ | fragliche Überexpression |
| 3 | 3+ | Überexpression |
| u.s.w. |
Tabelle 20: Score-Ergebnis (OID: 1.2.276.0.76.5.????)
Kommentar: Die mit ???? endenden OID müssen alle noch endgültig definiert werden. Können wir das tun?
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Section: Makroskopische Beschreibung
| Template ID | 1.3.6.1.4.1.19376.1.8.1.2.3 (AP APSR 6.2.4.3) | |
| General Description | In diesem Abschnitt werden die Daten zur Makroskopischen Beschreibung übermittelt. | |
| Status | identisch zu IHE | |
| LOINC Code | Opt. | Description |
| 22634-0 | R | Makroskopische Beschreibung |
| Template-Metadaten | |
| Template-Typ | Section |
| Template ID | 1.3.6.1.4.1.19376.1.8.1.2.3 (AP APSR 6.2.4.3) |
| generischeres Template | <Verweis auf das Template> |
| genutztes Templates | <Verweis auf das Template> |
| nutzende Templates | <Verweis auf das Template> |
| abgeleitete Templates | <Verweis auf das Template> |
| Schwester-Templates | <Verweis auf das Template> |
| Generelle Beschreibung | In diesem Abschnitt werden die Daten zur Makroskopischen Beschreibung übermittelt. |
| allg. Erläuterung | |
| Verhältnis zu IHE | identisch zu IHE (Übersetzung) |
| Ballotierungsstatus | in Arbeit |
| Erweiterbarkeit | geschlossen |
Definition und Zweck
Die makroskopische Beschreibung erfolgt als Freitext. Sie beinhaltet die Beschreibung des eingesandten Materials, seine makroskopischen Eigenschaften und Besonderheiten, Verarbeitungsinformationen des Materials (??), und die Zuschnittsinformationen (repräsentatives sampling, Gewebe für Spezialuntersuchungen)
Beispiel
<section>
<templateId root='1.3.6.1.4.1.19376.1.8.1.2.3'/>
<code code='22634-0'displayName='Pathologiebefund Makroskopische Beschreibung'
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title>MAKROSKOPIE</title>
<text>
1. Bezeichnet als Brustbiopsie links wurde das 3x3x3 cm messende, 20g schwere
rötlich-weiße weiche gewebsstück nach Entnahme einer mittigen Scheibe für die
Schnellschnittuntersuchung in 5 jeweils 3 mm dicke Scheiben von mamillennah nach mamillenfern
quer zur Milchgangsachse lamelliert. Zentral, nach allen Seiten mehr als 10 mm von den
Resektionsrändern entfernt, stellt sich ein grauweißlicher, leicht strahlig begrenzter derber
Knoten von 9 mm Durchmesser ab, an dessen lateralem Rand ein Stichkanal verläuft. Die
Gewebsscheiben I, III und V werden jeweils komplett unter Tuschemarkierung der
Resektionsränder gebettet. Die mediale Hälfte der Gewebsscheibe des Gefrierschnittes wird
schockgefrostet für die uPA/PAI-1-Untersuchung asserviert.
2. Bezeichnet als SLN Biopsie links finden sich zwei, jeweils 7 mm durchmessende
graurötliche, weiche Gewebsknoten mit wenig anhängendem Fettgewebe. Dies werden nach
Halbierung jeweils vollständig gebettet.
</text>
<entry> <-- Specimen Information entry, not shown here --> </entry>
</section>
Spezifikation
Die Section MUSS ein Code Element mit folgenden Attributen enthalten:
@code="22634-0" @codeSystem="2.16.840.1.113883.6.1" @displayName="Pathologiebefund Makroskopische Beschreibung"
Die Section MUSS einen narrativen Block enthalten, repräsentiert durch ein Textelement, welches die lesbare Information trägt.
Die Section SOLLTE so viele Specimen Information Organizer entry Elemente enthalten wie es Specimens oder Gruppen von Specimens gibt. Diese Entries enthalten die maschinenlesbaren Daten korrespondierend zum narrativen Block.
Die Section enthält keine Subsections.
Die Section SOLLTE ein Autor Element für den Fall enthalten, dass der/die Autoren von denen different sind, die in einem höheren Level im Dokument deklariert sind.
Content Module, die in die Section eingebunden werden sollten
| Name | Typ | Opt. | Card. | Template ID | Quelle |
| Specimen Information Organizer Entry | Entry | R2 | [0..*] | 1.3.6.1.4.1.19376.1.8.1.4.4 | PAT TF-3:6.2.6.4 |
| Author of the Section | Child | C | [0..*] | 1.3.6.1.4.1.19376.1.8.1.4.2 | PAT TF-3:6.2.6.2 |
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Section: Mikroskopische Beschreibung
| Template-Metadaten | |
| Template-Typ | Section |
| Template ID | 1.3.6.1.4.1.19376.1.8.1.2.4 (APSR 6.2.4.4) |
| generischeres Template | <Verweis auf das Template> |
| genutztes Templates | <Verweis auf das Template> |
| nutzende Templates | <Verweis auf das Template> |
| abgeleitete Templates | <Verweis auf das Template> |
| Schwester-Templates | <Verweis auf das Template> |
| Generelle Beschreibung | In diesem Abschnitt werden die Daten zur Mikroskopischen Beschreibung übermittelt. |
| allg. Erläuterung | vom Sektorkommitee Pathologie/Neuropathologie gefordert |
| Verhältnis zu IHE | identisch zu IHE (Übersetzung) |
| Ballotierungsstatus | in Arbeit |
| Erweiterbarkeit | geschlossen |
Definition und Zweck
Die mikroskopische Beschreibung erfolgt als Freitext. Je Material (Specimen) ist eine Section Mikroskopische Beschreibung notwendig. Optional können mehrere Materialien (Specimens) zusammengefasst werden. Ergebnisse von Spezialfärbungen, histochemischen, immunhistochemischen, molekularpathologischen und elektronenmikroskopischen Untersuchungen werden freitextlich in die beschreibung eingebunden und optional über Entries im CDA-Level 3 kodiert.
Beispiel
<section>
<templateId root='1.3.6.1.4.1.19376.1.8.1.2.4'/>
<code code='22635-7'displayName='Pathologiebefund Mikroskopische Beschreibung'
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title>MIKROSKOPIE</title>
<text>
Histologisch erweist sich der makroskopisch beschriebene Knoten als ein überwiegend
trabekulär wachsender, zu weniger als 10% tubuläre Formationen ausbildender adenomatöser
Tumor mit mäßiggradiger Zell- und Kernpolymorphie und einer durchschnittlichen Kerngröße
von mehr als 3 Erythrozytendurchmessern. Pro 10 HPF finden sich 8 Mitosefiguren.
Einbrüche in Lymph- oder Blutgefäße sind nicht nachweisbar. Geringgradige peritumorale
lymphoidzellige Stromareaktion. Kein Mikrokalk. Am Rande des Tumors zeigt sich ein
strichförmiges Gebiet von Blutungsresiduen und einem frischeren Granulationsgewebe
(Stichkanal der Stanzbiopsie).
</text>
<entry> <-- Specimen Information Organizer Entry, not shown here --> </entry>
</section>
Spezifikation
Die Section MUSS ein Code Element mit folgenden Attributen enthalten:
@code="22635-7" @codeSystem="2.16.840.1.113883.6.1" @displayName="Pathologiebefund Mikroskopische Beschreibung"
Die Section MUSS einen narrativen Block enthalten, repräsentiert durch ein Textelement, welches die lesbare Information trägt.
Die Section SOLLTE so viele Specimen Information Organizer Entry Elemente enthalten wie es Specimens oder Gruppen von Specimens gibt. Diese Entries enthalten die maschinenlesbaren Daten korrespondierend zum narrativen Block.
In den Specimen Information Organizer Entry Elements SOLLTEN so viele Immunhistologie Mikroskopie Entry Elemente enthalten sein wie es derartige Untersuchungen gibt. Diese Entries enthalten die maschinenlesbaren Daten korrespondierend zum narrativen Block.
Die Section SOLLTE ein Autor Element für den Fall enthalten, dass der/die Autoren von denen different sind, die in einem höheren Level im Dokument deklariert sind.
Die Section enthält keine Subsections.
Content Module, die in die Section eingebunden werden sollten
| Name | Typ | Opt. | Card. | Template ID | Quelle |
| Specimen Information Organizer Entry | Entry | R2 | [0..*] | 1.3.6.1.4.1.19376.1.8.1.4.4 | PAT TF-3:6.2.6.4 |
| child element: Immunhistologie Microskopie Entry-Template | Entry | R2 | [0..*] | tbd | |
| Author of the Section | Child | C | [0..*] | 1.3.6.1.4.1.19376.1.8.1.4.2 | PAT TF-3:6.2.6.2 |
Ergebnisse spezieller Untersuchungsmethoden
Immunhistologie (siehe Entry cdapath:Färbung-Section (Template) )
| Template ID | ?? | |
| General Description | In diesem Abschnitt werden die Daten zur Immunhistologie übermittelt. | |
| Status | genuin | |
| LOINC Code | Opt. | Description |
| 55229-9 | O | Immune stain study |
I.d.R. werden diagnoserelevante immunhistologische Befunde in der mikroskopischen Beschreibung erwähnt. Im Entry ist ein Vorschlag für eine sowohl diagnostisch als auch methodisch wichtige detaillierte Einzelbeschreibung zahlreicher Aspekte der durchgeführten Untersuchungen aufgeführt. Diese sollten zum großen Teil aus Prozessdaten der Laborautomaten / des Pathologiesystems beritgestellt werden.
Elektronenmikroskopie (z.Zt. kein strukturierter Befundvorschlag, d.h. kein Entry??)
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.7 ???? | |
| General Description | In diesem Abschnitt werden die Daten zur Elektronenmikroskopie übermittelt. | |
| Status | genuin | |
| LOINC Code | Opt. | Description |
| ???? | O | |
I.d.R. werden diagnoserelevante elektronenmikroskopische Befunde in der mikroskopischen Beschreibung erwähnt.
Molekularpathologie(z.Zt. kein strukturierter Befundvorschlag, d.h. kein Entry??)
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.7 ???? | |
| General Description | In diesem Abschnitt werden die Daten zur Molekularpathologie übermittelt. | |
| Status | genuin | |
| LOINC Code | Opt. | Description |
| ???? | O | |
I.d.R. werden diagnoserelevante molekularpathologische Befunde in der mikroskopischen Beschreibung erwähnt.
Bisher keine Vorarbeiten für entry bekannt, einzelne Ergebnisse (Befunde) in LOINC und IHE APSR als Observation zu kodieren.
Präparatradiographie(z.Zt. kein strukturierter Befundvorschlag, d.h. kein Entry)
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.7 ???? | |
| General Description | In diesem Abschnitt werden die Daten zur Präparatradiographie übermittelt. | |
| Status | identisch zu cdaab2??? | |
| LOINC Code | Opt. | Description |
| ???? | O | |
Diagnoserelevante Befunde werden in der mikroskopischen Beschreibung erwähnt. Vorschläge für kodierte Form liegen in den Checklisten für Mammakarzinome vor.
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Section: Unterbeauftragung
| Template ID | 1.2.276.0.76.3.1.131.1.10.2. ???? oder 1.3.6.1.4.1.19376.1.8.1.2.6 | |
| General Description | In diesem Abschnitt werden die Informationen über Unterbeauftragungen übermittelt. | |
| Status | genuin oder IHE | |
| LOINC Code | Opt. | Description |
| 46059-2 ???? | O | |
Untersuchungen, die als Unterauftrag weitergegeben werden, müssen hier gekennzeichnet werden:
- welches Material
- welche Untersuchung
- an wen gesandt
 |
Mgl.weise können diese Informationen auch in der Materialaufbereitungs-Section untergebracht werden. Sie betreffen aber nur ausnahmsweise das frische, eingesandte material, sondern viel häufiger einzelne Blöcke (Specimen Hierarchie!) |
Die Ergebnisse derartiger Untersuchungen werden in der Regel als Nachbericht mitgeteilt.
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Section: Diagnostische Schlussfolgerung
| Template-Metadaten | |
| Template-Typ | Section |
| Template ID | 1.3.6.1.4.1.19376.1.8.1.2.5 (APSR 6.2.4.5) |
| generischeres Template | <Verweis auf das Template> |
| genutztes Templates | <Verweis auf das Template> |
| nutzende Templates | <Verweis auf das Template> |
| abgeleitete Templates | <Verweis auf das Template> |
| Schwester-Templates | <Verweis auf das Template> |
| Generelle Beschreibung | In diesem Abschnitt werden die diagnostischen Schlussfolgerungen übermittelt. |
| allg. Erläuterung | vom Sektorkommitee Pathologie/Neuropathologie gefordert |
| Verhältnis zu IHE | constraints zu IHE |
| Ballotierungsstatus | in Arbeit |
| Erweiterbarkeit | geschlossen |
|
|
Die Diagnosen sind gemäß Diagnoseleitfaden[9] zu übermitteln! Die Darstellung wird aus den codierten Informationen (sofern z.B. aus Cancer Check List vorhanden) abgeleitet. |
Definition und Zweck
Die Diagnostische Schlussfolgerung Section enthält die diagnostischen Schlussfolgerungen ("Diagnosen") aller untersuchten Specimens (Parts, Materialien), die zusammen zur Untersuchung eingesandt wurden. Die Diagnosen werden für jedes Material oder jede Gruppe von Materialien separat angegeben. Die Section schließt zusätzliche pathologische Befunde und die Ergebnisse zusätzlicher Untersuchungen ein und kann Diagramme und Bilder und Virtual Slides beinhalten.
Bei Tumordiagnosen enthält sie innerhalb des Specimen Information Organizer-Entry die TNM- und ICD-O-, und R-Klassifikation sowie, falls vorhanden, eine Cancer Checklist.
Beispiel
<component>
<section>
<templateId root='1.3.6.1.4.1.19376.1.8.1.2.5'/>
<code code='22637-3' displayName='Pathology report diagnosis'
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title>DIAGNOSTISCHE SCHLUSSFOLGERUNG SECTION</title>
<text> 1.Intaktes Resektat eines mäßig differenzierten invasiven Mammakarzinoms, NST,
Malignitätsgrad 2 nach Elston und Ellis, aus dem oberen äußeren Quadranten der
linken Brust, im Gesunden entfernt.
2. Mikrometastasen in einem von 18 untersuchten axillären Lymphknoten.
</text>
<entry> <--Specimen Information Organizer, not shown here --> </entry>
</section>
</component>
Spezifikation
Die Section MUSS ein Code Element mit folgenden Attributen enthalten:
@code="22637-3" @codeSystem="2.16.840.1.113883.6.1" @displayName="Pathology report diagnosis"
Die Section MUSS einen narrativen Block enthalten, repräsentiert durch ein Textelement, welches die lesbare Information trägt.
Die Section MUSS so viele Specimen Information Organizer Entry Elemente enthalten wie es Specimens (Materialien) oder Gruppen von Specimens gibt. Diese Entries enthalten die maschinenlesbaren Daten korrespondierend zum narrativen Block.
|
|
Constraint zu IHE: Die Section MUSS im Falle von Tumordiagnosen die ICD-O-, TNM- und R-Klassifikation als Child Elemente die ICD-O-Klassifikation Entry (Template), TNM-Diagnose Entry (Template) und R-Klassifikation-Entry (Template) sowie die Phänomendaten aus cdaonk gemäß Phänomendaten-Section (Template)als Entry enthalten. |
Die Section SOLLTE ein Autor Element für den Fall enthalten, dass der/die Autoren von denen different sind, die in einem höheren Level im Dokument deklariert sind.
Die Section KANN eine Subsection "Zusammenfassung: ausführl. kritische gutachterliche Stellungnahme/Epikrise/Kommentar" enthalten.
Content Module, die in die Section eingebunden werden sollten
| Name | Typ | Opt. | Card. | Template ID | Quelle |
| Specimen Information Organizer | Entry | R | [1..*] | 1.3.6.1.4.1.19376.1.8.1.4.4 | PAT TF-3: 6.2.6.4 |
| Child element:TNM-Diagnose Entry | Entry | C | [0..*] | 1.2.276.0.76.3.1.131.1.10.3.5 | TNM-Diagnose |
| Child element:ICD-O-Diagnose Entry | Entry | C | [0..*] | 1.2.276.0.76.3.1.131.1.10.3.3 | ICD-O-Diagnose |
| Child element:R-Klassifikation Entry | Entry | C | [0..*] | 1.2.276.0.76.3.1.131.1.10.3.7 | R-Klassifikation |
| Child Element:Zusammenfassung | Sub-Section | O | [0..*] | 1.3.6.1.4.1.19376.1.8.1.2.7 | PAT TF-3: 6.2.4.7 |
| Author of the Section | Child | C | [0..*] | 1.3.6.1.4.1.19376.1.8.1.4.2 | PAT TF-3: 6.2.6.2 |
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Allgemeine Spezifikation aller APSR Entry Content Module
Jedes <entry> Content Modul ist an das korrespondierende Section Content Modul gebunden und enthält die maschinenlesbaren Daten in Bezug auf ein Material (Specimen) oder Gruppe von Materialien dieser Section.
Jedes <entry> Content Modul ist in der Section wiederholbar, so dass es das Konzept von mehreren Materialien, die als ein Fall eingesandt wurden, unterstützt.
| Datenelement | Verw. | Card. | Path and Constraints (Xpath + indentation) | Vokabular Quelle |
| Entry | Entry | |||
| Content Module conformance |
R | [1..1] | templateId[@root] |
Beispiel
 |
probeweise TNM als Component entsprechend cdaab2-TNM-Klassifikation-Entry eingebaut. Bitte prüfen, ob die Verschachtelung stimmt!! |
<entry>
<templateId root="1.3.6.1.4.1.19376.1.8.1.3.5"/>
<!-- Specimen Information Organizer -->
<organizer classCode="CLUSTER" moodCode="EVN">
<templateId root="1.3.6.1.4.1.19376.1.8.1.4.4"/>
<statusCode code="completed"/>
<!-- Specimen collection procedure -->
<component>
<procedure classCode="PROC" moodCode="EVN">
<templateId root="1.3.6.1.4.1.19376.1.3.1.2"/>
<code code="277261002" codeSystem="2.16.840.1.113883.6.96"
displayName="Exzision"/>
<targetSiteCode code="76752008" codeSystem="2.16.840.1.113883.6.96"
displayName="Brust"/>
<participant typeCode="PRD">
<participantRole classCode="SPEC">
<playingEntity>
<code code="309546004"
codeSystem="2.16.840.1.113883.6.96"
codeSystemName="SNOMED-CT"
displayName="Lumpektomie Probe"/>
</playingEntity>
</participantRole>
</participant>
</procedure>
</component>
<!-- Problem organizer -->
<component>
<organizer classCode="BATTERY" moodCode="EVN">
<templateId root="1.3.6.1.4.1.19376.1.8.1.4.8"/>
<statusCode code="completed"/>
<component>
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.3.6.1.4.1.19376.1.8.1.4.9"/>
<code code="371441004" 1805
codeSystem="2.16.840.1.113883.6.96"
codeSystemName="SNOMED-CT"
displayName="Histologischer Typ"/>
<statusCode code="completed"/>
<effectiveTime value="20100321063000.0000-0500"/>
<value xsi:type="CD" code="408643008"
displayName="Invasives Mammakarzinom, NST (Krankheit)"
codeSystem="2.16.840.1.113883.6.96"/>
</observation>
</component>
<!-- Tumorformel nach TNM -->
<component>
<observation classCode="OBS" moodCode="EVN">
<templateId root='' />
<id root='' extension=''' />
<code code='' codeSystem='' />
...
<value xsi:type="CD"
codeSystem="2.16.840.1.113883.15.8"
code="IV">
<originalText>
Tumorformel: T1 N2 ......
</originalText>
<!-- Einzelteile der Tumorformel -->
<entryRelationship typeCode="SPRT">
<observation moodCode="EVN" classCode="OBS">
<!-- T-Code -->
<value xsi:type="CD" code="T1"
codeSystem="2.16.840.1.113883.15.8" codeSystemName=" tnm5"/>
<qualifier>
<name code="341"
codeSystem="2.16.840.1.113883.3.7.1.0"/>
<value code="C2" codeSystem="1.2.276.0.76.5.341"/>
</qualifier>
</value>
</observation>
</entryRelationship>
<entryRelationship typeCode="SPRT">
<observation moodCode="EVN" classCode="OBS">
<!-- N-Code -->
<value xsi:type="CD" code="N2"
codeSystem="2.16.840.1.113883.15.8" codeSystemName="tnm5"/>
</value>
</observation>
</entryRelationship>
<entryRelationship typeCode="SPRT">
<observation moodCode="EVN" classCode="OBS">
<!-- M-Code -->
<value xsi:type="CD" code="M0"
displayName="Fernmetastasen nicht vorhanden"
codeSystem="2.16.840.1.113883.15.8" codeSystemName="tnm5"/>
</value>
</observation>
</entryRelationship>
</observation>
</component>
</organizer>
</component>
</organizer>
</entry>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Section: Zusammenfassung: ausführl. kritische gutachterliche Stellungnahme/Epikrise/Kommentar
| Template-Metadaten | |
| Template-Typ | Section |
| Template ID | 1.3.6.1.4.1.19376.1.8.1.2.7 (APSR 6.2.4.7) |
| generischeres Template | <Verweis auf das Template> |
| genutztes Templates | <Verweis auf das Template> |
| nutzende Templates | <Verweis auf das Template> |
| abgeleitete Templates | <Verweis auf das Template> |
| Schwester-Templates | <Verweis auf das Template> |
| Generelle Beschreibung | In diesem Abschnitt wird eine ausführliche kritische Gutachterliche Stellungnahme/Epikrise/Kommentar übermittelt. |
| allg. Erläuterung | vom Sektorkommitee Pathologie/Neuropathologie gefordert |
| Verhältnis zu IHE | identisch zu IHE (Übersetzung) |
| Ballotierungsstatus | in Arbeit |
| Erweiterbarkeit | geschlossen |
Definition und Zweck
Diese Section ist eine optionale Subsection der Diagnosesection. Sie enthält eine textliche Zusammenfassung bzw. epikritische Bewertung bzw. Kommentar des Pathologiebefundes, die aus dem Befund extrahiert und in klinische Dokumente eingefügt werden kann, um dort ein Segment zu bilden "...der Pathologiebefund stellt fest, dass ...".T
Entspricht inhaltlich der Epikrise des bvitG-Arztbriefes.
Beispiel
<section>
<templateId root='1.3.6.1.4.1.19376.1.8.1.2.5'/>
<code code='22637-3' displayName='Pathology report diagnosis'
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title>DIAGNOSE SECTION</title>
<text>1. Komplette Exzision eines invasiven Karzinoms, NST, im oberen äußeren Quadranten der linken Brust.
Malignitätsgrad 2 nach Elston und Ellis (2-3-2). Minimaler Sicherheitsabstand 11 mm nach kranial.
2. Micrometastase in einem der axillären Lymphknoten. </text>
<entry> <!-- detail not shown --> </entry>
<component>
<section>
<templateId root='1.3.6.1.4.1.19376.1.8.1.2.7'/>
<code code='35660-0' displayName='Pathology report final diagnosis section - text'
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC'/>
<title>ZUSAMMENFASSUNG</title>
<text>Invasives Karzinom der linken Brust
pT2pN1a(1/18)cM0, G2, R0 </text>
</section>
<component>
</section>
Spezifikation
Die Section MUSS ein Code Element mit folgenden Attributen enthalten:
@code="35660-0" @codeSystem="2.16.840.1.113883.6.1" @displayName="Pathology report final diagnosis section - text"
Die Section MUSS einen narrativen Block enthalten, repräsentiert durch ein Textelement, welches die lesbare Information trägt.
Die Section SOLLTE ein Autor Element für den Fall enthalten, dass der/die Autoren von denen different sind, die in einem höheren Level im Dokument deklariert sind.
Die Section DARF KEINE Entries enthalten.
Content Module, die in die Section eingebunden werden sollten
| Name | Typ | Opt. | Card. | Template ID | Quelle |
| Author of the Section | Child | C | [0..*] | 1.3.6.1.4.1.19376.1.8.1.4.2 | PAT TF-3:6.2.6.2 |
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Section: Grundleiden/Todesursache (klininisch)
| Template-Metadaten | |
| Template-Typ | Section |
| Template ID | |
| generischeres Template | |
| genutztes Templates | |
| nutzende Templates | |
| abgeleitete Templates | |
| Schwester-Templates | |
| generelle Beschreibung | In diesem Abschnitt werden die Informationen zur klin. Todesursache übermittelt. |
| allg. Erläuterung | |
| Verhältnis zu IHE | dt.Übersetzung oder Ergänzung oder neu |
| Ballotierungsstatus | identisch zu cdaab2??? |
| Erweiterbarkeit | offen oder geschlossen |
Beschreibung
Dieser Abschnitt ist nur Bestandteil des Obduktionsbefundes / Sektionsberichtes. Im Einzelnen werden hier tabellarisch folgende Items aufgelistet:
| Bedeutung | Diagnosetext | Zeitdauer zwischen Krankheit und Tod |
ICD-10 Code |
|---|---|---|---|
| Unmittelbar zum Tode führende Krankheit (Todesursache) | Ia) 1..1 | 0..1 | 1..1 |
| Vorausgegangene Ursache | Ib) 0..1 | 0..1 | 0..1 |
| Vorausgegangene Ursache (Grundleiden) | Ic) 0..1 | 0..1 | 0..1 |
| Begleitkrankheit | II 0..1 | 0..1 | 0..1 |
| Begleitkrankheit | II 0..1 | 0..1 | 0..1 |
|
|
Begleitkrankheit ist doppelt. |
|
|
Was ist hier mit den Codes gemeint? FO: Code zur Identifkation der Section, ggf. auch codes für die Inhalte |
Für diese Tabellen wurden noch keine Codes vorgeschlagen
Beispiel
|
|
fehlt |
Section: Grundleiden/Todesursache (autoptisch)
| Template-Metadaten | |
| Template-Typ | Header oder Section oder Entry |
| Template ID | |
| generischeres Template | |
| genutztes Templates | |
| nutzende Templates | |
| abgeleitete Templates | |
| Schwester-Templates | |
| generelle Beschreibung | In diesem Abschnitt werden die Informationen zur autoptischen Todesursache übermittelt. |
| allg. Erläuterung | |
| Verhältnis zu IHE | dt.Übersetzung oder Ergänzung oder neu |
| Ballotierungsstatus | identisch zu cdaab2??? |
| Erweiterbarkeit | offen oder geschlossen |
Beschreibung
Dieser Abschnitt ist nur Bestandteil des Obduktionsbefundes / Sektionsberichtes. Im Einzelnen werden hier tabellarisch folgende Items aufgelistet:
| Bedeutung | Diagnosetext | ICD-10 Code |
|---|---|---|
| Unmittelbar zum Tode führende Krankheit (Todesursache) | Ia) 1..1 | 1..1 |
| Vorausgegangene Ursache | Ib) 0..1 | 0..1 |
| Vorausgegangene Ursache (Grundleiden) | Ic) 0..1 | 0..1 |
| Begleitkrankheit | II 0..1 | 0..1 |
| Begleitkrankheit | II 0..1 | 0..1 |
|
|
Begleitkrankheit ist doppelt. |
|
|
Was ist hier mit den Codes gemeint? FO: Code zur Identifkation der Section, ggf. auch codes für die Inhalte |
Für diese Tabellen wurden noch keine Codes vorgeschlagen
Beispiel
|
|
fehlt |
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Section: Äußere Leichenschau
| Template-Metadaten | |
| Template-Typ | Section |
| Template ID | |
| generischeres Template | |
| genutztes Templates | |
| nutzende Templates | |
| abgeleitete Templates | |
| Schwester-Templates | |
| generelle Beschreibung | In diesem Abschnitt werden die Informationen zur äußeren Leichenschau übermittelt. |
| allg. Erläuterung | |
| Verhältnis zu IHE | neu |
| Ballotierungsstatus | in Arbeit |
| Erweiterbarkeit | offen |
Beschreibung
|
|
tbd |
Attribute
| Lvl | RIM | Name | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|
| 1 | act | section | 1..1 | M | ||
| 2 | act | templateId | CD CNE | 1..1 | F | |
| 2 | act | code | CD CNE | 1..1 | F | |
| 2 | act | title | ST | 1..1 | F | "äußere Leichenschau" |
| 2 | act | text | ST | 1..1 | M | Beschreibung |
Beispiel
<section>
<templateId root="1.2.276.0.76.3.1.131.1.10.3.xxxx"/>
<code code="xxxx" codeSystem="" />
<title>äußere Leichenschau</title>
<text>
...
</text>
</section>
Section: Innere Leichenschau
| Template-Metadaten | |
| Template-Typ | Section |
| Template ID | |
| generischeres Template | |
| genutztes Templates | |
| nutzende Templates | |
| abgeleitete Templates | |
| Schwester-Templates | |
| generelle Beschreibung | In diesem Abschnitt werden die Informationen zur inneren Leichenschau übermittelt. |
| allg. Erläuterung | |
| Verhältnis zu IHE | neu |
| Ballotierungsstatus | in Arbeit |
| Erweiterbarkeit | offen |
Beschreibung
|
|
tbd |
Attribute
| Lvl | RIM | Name | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|
| 1 | act | section | 1..1 | M | ||
| 2 | act | templateId | CD CNE | 1..1 | F | |
| 2 | act | code | CD CNE | 1..1 | F | |
| 2 | act | title | ST | 1..1 | F | "innere Leichenschau" |
| 2 | act | text | ST | 1..1 | M | Beschreibung |
Beispiel
<section>
<templateId root="1.2.276.0.76.3.1.131.1.10.3.xxxx"/>
<code code="xxxx" codeSystem="" />
<title>innere Leichenschau</title>
<text>
...
</text>
</section>
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Section: Weitergabemodus
| Template ID | 1.2.276.0.76.3.1.131.1.10.2.7 ???? | |
| General Description | In diesem Abschnitt werden die Daten zum Weitergabemodus übermittelt. | |
| LOINC Code | Opt. | Description |
| ???? | O | |
tbd
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Section: Abschließende Bemerkungen (Schlusstext)
| Id | 1.2.276.0.76.10.3034 | Gültigkeit | 2013‑12‑30 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Status | Versions-Label | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Name | Finalremarkssection | Bezeichnung | Abschließende Bemerkungen | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Beschreibung | Ein am Ende des Briefes formulierter Freitext entsprechend einer Grußformel, z.B.: "mit kollegialen Grüßen | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Klassifikation | CDA Section level template | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Offen/Geschlossen | Offen (auch andere als die definierten Elemente sind erlaubt) | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Beziehung | Spezialisierung: Template 2.16.840.1.113883.10.12.201 CDA Section (2005‑09‑07) ref ad1bbr- | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
Anmerkung: LOINC Codes mit einem vorangestellten X, wie hier X-FINREM, werden kurzfristig durch tatsächliche numerische LOINC Codes ersetzt. |
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Vokabeldomänen
Einleitung
Dieser Abschnitt dient der Trennung von verwendeten Codes und der normativen Spezifikation. Damit lassen sich die Codes aktualisieren, ohne dass die Spezifikation überarbeitet werden muss.
Dieser Abschnitt ist deshalb nur informativ. Die jeweils aktuellen Codes sind deshalb zu erfragen.
Überblick über die Codierschemata
Die Liste der Kodesysteme wurde auf eine eigene Seite ausgelagert.
generische Codes für Attribut-Wert-Paare
Befundinterpretation im Kontext
| Code | Codename | Bedeutung |
|---|---|---|
| negativ | nicht zutreffend (z.B.für Malignität) | |
| positiv | zutreffend (z.B. für Malignität) | |
| zweifelhaft | nicht sicher zutreffend (z.B. für Malignität) |
Tabelle 19: Befundinterpretation im Kontext mit Fragestellung (OID 1.2.276.0.76.5.??????)
Hier stellt sich die Frage, ob bestimmte Attribute nicht auch über boolesche Werte abgebildet werden können?
z.B. in folgendem Beispiel
| Code | Codename | ja | nein | unsicher | nicht bestimmbar | keine Aussage |
|---|---|---|---|---|---|---|
| Übereinstimmung mit klinischer Fragestellung | x | x | x | x | ||
| Übereinstimmung mit Referenzdiagnose | x | x | x | x | ||
| etc. |
Tabelle 20: Befundinterpretation im Kontext mit Fragestellung (OID 1.2.276.0.76.5.??????)
Codes aus IHE Anatomy Pathology Report
Codes für Specimen Types
nach IHE (IHE APSR Trial Implementation, March 31, 2011) ist ein Specimen collection procedure generic template das Elterntemplate für jedes organspezifische Template. Jedes organspezifische Template hat Vokabularbegrenzungen, die für das jeweilige Organ spezifisch sind:
Ein Value Set ist gebunden an ein Prozedur Code-Element, das die verschiednen Prozeduren, die an diesem Organ möglich sind, auflistet.
Ein zweiter Value Set ist gebunden and das Prozedut-Target-Site Code-Element, das die möglichen präzisen Lokalisationen an diesem spezifischen Organ auflistet.
z.B.
| (PathLex)Code | Codename | Bedeutung |
|---|---|---|
| 2257 | Breast-Specimen-Specimen collection procedure | Excision with wire-guided localization |
| 2256 | Breast-Specimen-Specimen collection procedure | Excision without wire-guided localization |
| 662 | Breast-Specimen-Specimen collection procedure | Total mastectomy (including nipple and skin) |
| 666 | Breast-Specimen-Target site | Lower inner quadrant |
| 663 | Breast-Specimen-Target site | Upper outer quadrant |
Tabelle 24: HL7 Table 0487 resp. 0700 (OID 1.2.276.0.76.5.??????)
Codes für Specimen Reject Reason
| Code | Codename | Bedeutung |
|---|---|---|
| EX | expired | verfallen |
| QS | quantity not sufficient | Menge nicht ausreichend |
| RB | broken container | Einsendegefäß zerbrochen |
| RE | missing collection date | fehlende Angabe zum Entnahmedatum |
| R | missing patient ID | fehlender Patientencode |
| RE | missing patient name | fehlender Patientenname |
| etc. |
Tabelle 25: HL7 Table 0490 (OID 1.2.276.0.76.5.??????)
LOINC Codes
| LOINC Code | LOINC Code Name |
|---|---|
| 22637-3 | Path report.final diagnosis |
| 33746-9 | Pathologic findings |
| 22636-5 | Path report.relevant Hx |
| 22633-2 | Path report.site of origin |
| 22634-0 | Path report.gross description |
| 22635-7 | Path report.microscopic observation |
| 22638-1 | Path report.comments |
| 22639-9 | Path report.supplemental reports |
Tabelle 26: LOINC Codes (OID 2.16.840.1.113883.6.1)
Cancer Check Lists
| OID | Name |
|---|---|
| 1.3.6.1.4.1.19376.1.8.1.1.1 | Generic APSR |
| 1.3.6.1.4.1.19376.1.8.1.1.2.1 | Breast APSR |
| 1.3.6.1.4.1.19376.1.8.1.1.2.2 | Colonic APSR |
| 1.3.6.1.4.1.19376.1.8.1.1.2.3 | Prostate APSR |
| 1.3.6.1.4.1.19376.1.8.1.1.2.x | etc. |
Tabelle 27: Generische und Organspezifische Cancer Check Lists (OID 1.3.6.1.4.1.19376.1.8.1.1.2)
Generische Cancer Check List und Value set (ohne Organspezifität)
zum Teil werden die Sachverhalte auch vom Diagnoseleitfaden abgebildet (z.B. TNM). Doppelungen zulassen??
| Name | IHE_PAT_elementTemplates _Element_template_ID | IHE_PAT_Element_name | ValueSetID |
|---|---|---|---|
| Material | 1.3.6.1.4.1.19376.1.8.1.4.1 | Generic-Specimen Collection Procedure | 1.3.6.1.4.1.19376.1.8.5.371 |
| RR_Infiltration_INV | 1.3.6.1.4.1.19376.1.8.1.4.140 | Generic-Infiltrating malignant neoplasm-Margins involvement | BL |
| RR_Infiltration_CIS | 1.3.6.1.4.1.19376.1.8.1.4.141 | Generic-In situ neoplasm-Margins involvement | BL |
| RR_Infiltration | 1.3.6.1.4.1.19376.1.8.1.4.142 | Generic-Lesion-Margins involvement | BL |
| Fokalität_INV | 1.3.6.1.4.1.19376.1.8.1.4.143 | Generic-Infiltrating malignant neoplasm-Lesion focality | 1.3.6.1.4.1.19376.1.8.5.5 |
| TNM-Deskriptoren | 1.3.6.1.4.1.19376.1.8.1.4.144 | Generic-Infiltrating malignant neoplasm-TNM Descriptors | 1.3.6.1.4.1.19376.1.8.5.6 |
| Abstand_Nächster_RR_INV | 1.3.6.1.4.1.19376.1.8.1.4.145 | Generic-Infiltrating malignant neoplasm-Distance of lesion from closest uninvolved margin | PQ |
| Abstand_Nächster_RR_CIS | 1.3.6.1.4.1.19376.1.8.1.4.146 | Generic-In situ neoplasm-Distance of lesion from closest uninvolved margin | PQ |
| Lymphgefäßinvasion | 1.3.6.1.4.1.19376.1.8.1.4.147 | Generic-Infiltrating malignant neoplasm-Lymph-vascular invasion | BL |
| Lymphknotensampling | 1.3.6.1.4.1.19376.1.8.1.4.148 | Generic-Infiltrating malignant neoplasm-Lymph node sampling | BL |
| Ausdehnung | 1.3.6.1.4.1.19376.1.8.1.4.149 | Generic-Infiltrating malignant neoplasm-Extent | 1.3.6.1.4.1.19376.1.8.5.328 |
| Histol_Grad_2Stufig | 1.3.6.1.4.1.19376.1.8.1.4.150 | Generic-Infiltrating malignant neoplasm-Histologic grade (Two-Tier Grading System) | 1.3.6.1.4.1.19376.1.8.5.245 |
| Histol_Grad_WHO | 1.3.6.1.4.1.19376.1.8.1.4.151 | Generic-Infiltrating malignant neoplasm-Histologic grade (WHO) | 1.3.6.1.4.1.19376.1.8.5.246 |
| Perineuralscheideninvasion | 1.3.6.1.4.1.19376.1.8.1.4.152 | Generic-Infiltrating malignant neoplasm-Perineural invasion | BL |
| Lokalisation_RR | 1.3.6.1.4.1.19376.1.8.1.4.153 | Generic-Infiltrating malignant neoplasm-Margin site | 1.3.6.1.4.1.19376.1.8.5.192 |
| Lokalisation_Läsion_INV | 1.3.6.1.4.1.19376.1.8.1.4.154 | Generic-Infiltrating malignant neoplasm-Lesion site | 1.3.6.1.4.1.19376.1.8.5.329 |
| Lokalisation_Lymphknoten | 1.3.6.1.4.1.19376.1.8.1.4.155 | Generic-Infiltrating malignant neoplasm-Lymph node site | 1.3.6.1.4.1.19376.1.8.5.330 |
| Lymphknoten_befallen | 1.3.6.1.4.1.19376.1.8.1.4.156 | Generic-Infiltrating malignant neoplasm-Number of lymph nodes involved | INT |
| Lymphknoten_untersucht | 1.3.6.1.4.1.19376.1.8.1.4.157 | Generic-Infiltrating malignant neoplasm-Number of lymph nodes examined | INT |
| pM | 1.3.6.1.4.1.19376.1.8.1.4.158 | Generic-Infiltrating malignant neoplasm-pM | 1.3.6.1.4.1.19376.1.8.5.9 |
| pN | 1.3.6.1.4.1.19376.1.8.1.4.159 | Generic-Infiltrating malignant neoplasm-pN | 1.3.6.1.4.1.19376.1.8.5.8 |
| Probengewicht_INV | 1.3.6.1.4.1.19376.1.8.1.4.160 | Generic-Infiltrating malignant neoplasm-Specimen weight | PQ |
| pT | 1.3.6.1.4.1.19376.1.8.1.4.161 | Generic-Infiltrating malignant neoplasm-pT | 1.3.6.1.4.1.19376.1.8.5.7 |
| Behandlungseffekt | 1.3.6.1.4.1.19376.1.8.1.4.162 | Generic-Infiltrating malignant neoplasm-Treatment effect | 1.3.6.1.4.1.19376.1.8.5.10 |
| Läsionsgröße_Zusatzdimension | 1.3.6.1.4.1.19376.1.8.1.4.163 | Generic-Infiltrating malignant neoplasm-Lesion size, additional dimension | PQ |
| Läsionsgröße_größteDimension | 1.3.6.1.4.1.19376.1.8.1.4.164 | Generic-Infiltrating malignant neoplasm-Lesion size, largest dimension | PQ |
| Probengröße_Zusatzdimension_INV | 1.3.6.1.4.1.19376.1.8.1.4.165 | Generic-Infiltrating malignant neoplasm-Specimen size, additional dimension | PQ |
| Probengröße_größteDimension_INV | 1.3.6.1.4.1.19376.1.8.1.4.166 | Generic-Infiltrating malignant neoplasm-Specimen size, largest dimension | PQ |
| HistologischerTyp_INV | 1.3.6.1.4.1.19376.1.8.1.4.167 | Generic-Infiltrating malignant neoplasm-Histologic type | 1.3.6.1.4.1.19376.1.8.5.331 |
| MakroskopischerTyp_INV | 1.3.6.1.4.1.19376.1.8.1.4.168 | Generic-Infiltrating malignant neoplasm-Macroscopic type | 1.3.6.1.4.1.19376.1.8.5.187 |
| Lokalisation_Läsion_CIS | 1.3.6.1.4.1.19376.1.8.1.4.169 | Generic-In situ neoplasm-Lesion site | 1.3.6.1.4.1.19376.1.8.5.184 |
| HistologischerTyp_CIS | 1.3.6.1.4.1.19376.1.8.1.4.170 | Generic-In situ neoplasm-Histologic type | 1.3.6.1.4.1.19376.1.8.5.183 |
| Fokalität | 1.3.6.1.4.1.19376.1.8.1.4.171 | Generic-Lesion-Lesion focality | 1.3.6.1.4.1.19376.1.8.5.3 |
| Lokalisation | 1.3.6.1.4.1.19376.1.8.1.4.172 | Generic-Lesion-Lesion site | 1.3.6.1.4.1.19376.1.8.5.332 |
| HistologischerTyp | 1.3.6.1.4.1.19376.1.8.1.4.173 | Generic-Lesion-Histologic type | 1.3.6.1.4.1.19376.1.8.5.333 |
| Probenintegrität | 1.3.6.1.4.1.19376.1.8.1.4.174 | Generic-Specimen-Specimen integrity | 1.3.6.1.4.1.19376.1.8.5.2 |
Tabelle 28: Codeliste für Generische Cancer Check List (ohne Organspezifität)(OID 1.3.6.1.4.1.19376.1.8.1.1.1)
| ValueSetID | PathLex_Code | PathLex_Value |
|---|---|---|
| 1.3.6.1.4.1.19376.1.8.5.371 | ?? | ?? |
| 1.3.6.1.4.1.19376.1.8.5.5 | 801 | Single focus (Solitary - Unifocal) |
| 1.3.6.1.4.1.19376.1.8.5.5 | 802 | Multiple foci (Multifocal - (Ipsilateral – Bilateral))(specify location) |
| 1.3.6.1.4.1.19376.1.8.5.6 | 1597 | Multiple primary tumors |
| 1.3.6.1.4.1.19376.1.8.5.6 | 1598 | Recurrent |
| 1.3.6.1.4.1.19376.1.8.5.6 | 1599 | Post-treatment |
| 1.3.6.1.4.1.19376.1.8.5.328 | ?? | ?? |
| 1.3.6.1.4.1.19376.1.8.5.245 | 623 | High grade |
| 1.3.6.1.4.1.19376.1.8.5.245 | 624 | Low grade |
| 1.3.6.1.4.1.19376.1.8.5.246 | 569 | G1: Well differentiated |
| 1.3.6.1.4.1.19376.1.8.5.246 | 570 | G2: Moderately differentiated |
| 1.3.6.1.4.1.19376.1.8.5.246 | 571 | G3: Poorly differentiated" |
| 1.3.6.1.4.1.19376.1.8.5.192 | ?? | ?? |
| 1.3.6.1.4.1.19376.1.8.5.329 | ?? | ?? |
| 1.3.6.1.4.1.19376.1.8.5.330 | ?? | ?? |
| 1.3.6.1.4.1.19376.1.8.5.9 | 1433 | Concept pM UICC TNM 7ème édition |
| 1.3.6.1.4.1.19376.1.8.5.8 | 1432 | Concept pN UICC TNM 7ème édition |
| 1.3.6.1.4.1.19376.1.8.5.7 | 1431 | Concept pT UICC TNM 7ème édition |
| 1.3.6.1.4.1.19376.1.8.5.10 | 734 | No residual tumor (complete response, grade 0) |
| 1.3.6.1.4.1.19376.1.8.5.10 | 735 | Moderate response (grade 2) |
| 1.3.6.1.4.1.19376.1.8.5.10 | 736 | No definite response identified (grade 3, poor or no response) |
| 1.3.6.1.4.1.19376.1.8.5.10 | 2271 | Marked response (grade 1, minimal residual cancer) |
| 1.3.6.1.4.1.19376.1.8.5.331 | ?? | ?? |
| 1.3.6.1.4.1.19376.1.8.5.187 | ?? | ?? |
| 1.3.6.1.4.1.19376.1.8.5.184 | ?? | ?? |
| 1.3.6.1.4.1.19376.1.8.5.183 | ?? | ?? |
| 1.3.6.1.4.1.19376.1.8.5.3 | 799 | Single focus (Solitary - Unifocal) |
| 1.3.6.1.4.1.19376.1.8.5.3 | 800 | Multiple foci (Multifocal - (Ipsilateral – Bilateral))(specify location) |
| 1.3.6.1.4.1.19376.1.8.5.332 | ?? | ?? |
| 1.3.6.1.4.1.19376.1.8.5.333 | ?? | ?? |
| 1.3.6.1.4.1.19376.1.8.5.2 | 797 | Intact specimen(s) (Unopened, Capsule intact, Single intact specimen, Multiple intact, designated specimens (margins can be evaluated)) |
| 1.3.6.1.4.1.19376.1.8.5.2 | 798 | Non intact specimen (Open, Capsule ruptured specimen, Multiple non designated specimens (Fragmented, Morcellated)(margins cannot be evaluated with certainty) |
Tabelle 29: Value sets für Generische Cancer Check List (ohne Organspezifität)(OID 1.3.6.1.4.1.19376.1.8.2.1)
Cancer Check List und Value set für Mammakarzinome
| Name_LL | IHE_PAT_elementTemplates _Element_template_ID | IHE_PAT_Element_name | ValueSetID |
|---|---|---|---|
| DCIS_CRM_DIST | 1.3.6.1.4.1.19376.1.8.1.4.419 | Breast-In situ neoplasm-Distance of lesion from closest uninvolved margin | PQ |
| DCIS_GRAD | 1.3.6.1.4.1.19376.1.8.1.4.444 | Breast-In situ neoplasm-Histologic grade of ductal carcinoma in situ (DCIS) | 1.3.6.1.4.1.19376.1.8.5.23 |
| DCIS_Typ | 1.3.6.1.4.1.19376.1.8.1.4.446 | Breast-In situ neoplasm-Histologic type | 1.3.6.1.4.1.19376.1.8.5.254 |
| DCIS_DIMEN_LARG | 1.3.6.1.4.1.19376.1.8.1.4.442 | Breast-In situ neoplasm-Lesion size, largest dimension | PQ |
| DCIS_MARG_INVOLV | 1.3.6.1.4.1.19376.1.8.1.4.414 | Breast-In situ neoplasm-Lesion size, largest dimension | BL |
| DCIS_NECROSIS | 1.3.6.1.4.1.19376.1.8.1.4.429 | Breast-In situ neoplasm-Necrosis of ductal carcinoma in situ (DCIS) | 1.3.6.1.4.1.19376.1.8.5.253 |
| INV_CRM_DIST | 1.3.6.1.4.1.19376.1.8.1.4.418 | Breast-Infiltrating malignant neoplasm-Distance of lesion from closest uninvolved margin | PQ |
| INV_ER | 1.3.6.1.4.1.19376.1.8.1.4.439 | Breast-Infiltrating malignant neoplasm-Estrogen receptor | 1.3.6.1.4.1.19376.1.8.5.31 |
| INV_PgR | 1.3.6.1.4.1.19376.1.8.1.4.438 | Breast-Infiltrating malignant neoplasm-Progesterone receptor | 1.3.6.1.4.1.19376.1.8.5.34 |
| INV_HER2_ISH | 1.3.6.1.4.1.19376.1.8.1.4.416 | Breast-Infiltrating malignant neoplasm-HER2/neu (FISH method) | 1.3.6.1.4.1.19376.1.8.5.32 |
| etc. | 1.3.6.1.4.1.19376.1.8.5.x |
Tabelle 30: Codeliste für Cancer Check List Mammakarzinom(OID 1.3.6.1.4.1.19376.1.8.1.1.2.1)
| ValueSetID | PathLex_Code | PathLex_Value |
|---|---|---|
| 1.3.6.1.4.1.19376.1.8.5.23 | 743 | low grade |
| 1.3.6.1.4.1.19376.1.8.5.23 | 744 | intermediate grade |
| 1.3.6.1.4.1.19376.1.8.5.23 | 745 | high grade |
| 1.3.6.1.4.1.19376.1.8.5.254 | 2308 | Ductal carcinoma in situ with microinvasion |
| 1.3.6.1.4.1.19376.1.8.5.254 | 2309 | Lobular carcinoma in situ with microinvasion |
| 1.3.6.1.4.1.19376.1.8.5.254 | 2557 | DCIS Comedo |
| 1.3.6.1.4.1.19376.1.8.5.254 | 2558 | DCIS Paget disease (DCIS involving nipple skin) |
| 1.3.6.1.4.1.19376.1.8.5.254 | 2559 | DCIS Cribriform |
| 1.3.6.1.4.1.19376.1.8.5.254 | 2560 | DCIS Micropapillary |
| 1.3.6.1.4.1.19376.1.8.5.254 | 2561 | DCIS Papillary |
| 1.3.6.1.4.1.19376.1.8.5.254 | 2562 | DCIS Solid |
| 1.3.6.1.4.1.19376.1.8.5.253 | 2168 | Not identified |
| 1.3.6.1.4.1.19376.1.8.5.253 | 2169 | Present, focal (small foci or single cell necrosis) |
| 1.3.6.1.4.1.19376.1.8.5.253 | 2170 | Present, central (expansive “comedo” necrosis) |
| 1.3.6.1.4.1.19376.1.8.5.31 | 1602 | No immunoreactive tumor cells present |
| 1.3.6.1.4.1.19376.1.8.5.31 | 2269 | Immunoreactive tumor cells present (> = 1%) (Specify Quantitation) |
| 1.3.6.1.4.1.19376.1.8.5.31 | 2270 | Less than 1% immunoreactive cells present |
| 1.3.6.1.4.1.19376.1.8.5.34 | 740 | No immunoreactive tumor cells present |
| 1.3.6.1.4.1.19376.1.8.5.34 | 2267 | Immunoreactive tumor cells present (> = 1%) (Specify Quantitation) |
| 1.3.6.1.4.1.19376.1.8.5.34 | 2268 | Less than 1% immunoreactive cells present |
| 1.3.6.1.4.1.19376.1.8.5.32 | 741 | Amplified (HER2 gene copy >6.0 or ratio >2.2) |
| 1.3.6.1.4.1.19376.1.8.5.32 | 742 | Not amplified (HER2 gene copy <4.0 or ratio <1.8) |
| 1.3.6.1.4.1.19376.1.8.5.32 | 1925 | Equivocal (HER2 gene copy 4.0 to 6.0 or ratio 1.8 to 2.2) |
| 1.3.6.1.4.1.19376.1.8.5.x | yy | etc. |
Tabelle 31: Value sets für Cancer Check List Mammakarzinom(OID 1.3.6.1.4.1.19376.1.8.2.1)
Cancer Check List und Value set für kolorektale Karzinome
| Name_LL | IHE_PAT_elementTemplates _Element_template_ID | IHE_PAT_Element_name | ValueSetID |
|---|
TBD
Tabelle 32: Codeliste für Cancer Check List kolorektale Karzinome(OID 1.3.6.1.4.1.19376.1.8.1.1.2.2)
| ValueSetID | PathLex_Code | PathLex_Value |
|---|
TBD
Tabelle 33: Value sets für Cancer Check List kolorektale Karzinome(OID 1.3.6.1.4.1.19376.1.8.2.2)
Cancer Check List und Value set für Prostatakarzinome
| Name_LL | IHE_PAT_elementTemplates _Element_template_ID | IHE_PAT_Element_name | ValueSetID |
|---|
TBD
Tabelle 34: Codeliste für Cancer Check List Prostatakarzinome(OID 1.3.6.1.4.1.19376.1.8.1.1.2.3)
| ValueSetID | PathLex_Code | PathLex_Value |
|---|
TBD
Tabelle 35: Value sets für Cancer Check List Prostatakarzinome(OID 1.3.6.1.4.1.19376.1.8.2.3)
Dieses Material ist Teil des Leitfadens Implementierungsleitfaden.
|
Anhang A: Diverses
Dokumentenhistorie
| Version | Stand | Bearbeiter | Beschreibung | Dok.-OID |
|---|---|---|---|---|
| 2014 | GH + FO | Layout, Tabellendarstellung, Umstrukturierung, Sections, Entries, Vokabularien, Dokumenttypen, Abgleich mit IHE-AP und HL7-AP | n.a. | |
| 19.10.12 | FO | Layout, Tabellendarstellung, Umstrukturierung | n.a. | |
| 18.10.12 | GH | Detailinfos | n.a. | |
| 06 | 13.04.12 | FO et.al. | Wikifizierung + Überarbeitung: Neustrukturierung | n.a. |
| 05 | 23.02.10 | FO | Überarbeitung: Neustrukturierung | n.a. |
| 04 | 16.11.09 | FO | Überarbeitung: Neustrukturierung | n.a. |
| 03 | 15.09.09 | FO | Überarbeitung | n.a. |
| 02 | 08.07.09 | FO | Überarbeitung | n.a. |
| 01 | 23.06.09 | IR | Dokument erstellt | n.a. |
Kollaboratives Werkzeug von IHE APSR
Durch IHE AP wurde für die APSR-Entwicklung, speziell die weitere Entwicklung der Interface-Terminologie PathLex, folgendes Werkzeug in einer beta Version zur verfügung gestellt:
http://termapp.davidouagne.com
Organspezifischer Pathologiebefundbericht
| Template ID | 1.3.6.1.4.1.19376.1.8.1.1.2.1 - 20 (AP APSR 6.2.3.2) | |
| General Description | In diesem Template werden die Daten zum organspezifischen Pathologiebefundbericht übermittelt. | |
| Status | identisch zu IHE | |
| LOINC Code | Opt. | Description |
| 11526-1 | R2 | Pathology Study |
Definition und Zweck
Dieses Template definiert bisher die Grundlage für die organspezifischen Einschränkungen von strukturierten Pathologiebefundberichten mit Tumordiagnosen. Das Parent Template ist 1.3.6.1.4.1.19376.1.8.1.1.1 (Pathologiebefundbericht), von dem es alle Eigenschaften erbt. Zukünftig wird es nur ein generisches Parenttemplate geben, in das durch verschiedene Problem organizer Templates generische oder läsionsspezifische AP Observation Templates eingebunden werden können. Diese sind durch eine Ontologie (OntoPathLex) definiert.
Beispiel
<ClinicalDocument xmlns='urn:hl7-org:v3'>
<typeId extension="POCD_HD000040" root="2.16.840.1.113883.1.3"/>
<!-- conformance to a generic APSR content module -->
<templateId root='1.3.6.1.4.1.19376.1.8.1.1.1'/>
<!-- conformance to a breast content module -->
<templateId root='1.3.6.1.4.1.19376.1.8.1.1.2.1'/>
<!-- remainder of the header not shown, similar to the header content of a generic APSR -->
<component>
<structuredBody>
<!—Same hierarchy of sections and entries as in generic APSR -->
</structuredBody>
</component>
</ClinicalDocument>
Spezifikation
Die organspezifischen Document Content Module berücksichtigen die Hierarchie von <section> und <entry> Elementen des generischen Document Content Moduls sowie die Beschränkungen ihrer Parent-Templates.
Jedes organspezifische Document Content Modul besteht aus einer Vokabularsatzbeschränkung für die Content Module "AP Observation" und "Specimen Collection Procedure", welche in jedem der <entry> Elemente dieses Document Content Moduls verschachtelt sein können.
Insbesondere werden die organspezifischen Observations bezüglich invasiver und nichtinvasiver maligner Tumoren anhand von sog. Cancer Checklists (z.B. CAP, deutsche S3-Leitlinien, Empfehlungen der Dt.Ges.f.Pathologie, etc.) spezifiziert. Die dazugehörigen Value sets sind im APSR Value Sets Appendix aufgeführt. Für den deutschsprachigen Raum werden diese weiter ergänzt bzw. ihre Verwendung begrenzt (National Extension).
Offene Punkte
- Codesysteme vervollständigen
- fehlende Abschnitte:
- n.n.
- Elemente und Attribute vollständig auflisten (inkl. Wikifizierung)
- Abgleich mit IHE Anatomic Pathology APSR und HL7 Anatomic Pathology
- ..
Referenzen/Literatur
| DIMDI, Alpha_Id: | Alpha-ID - Die Identifikationsnummer, http://www.dimdi.de/static/de/ehealth/alpha-id/index.htm |
| DIMDI, Verschl: | Anleitung zur Verschlüsselung, http://www.dimdi.de/static/de/klassi/diagnosen/icd10/icdsgbv20.htm |
| DIMDI, Basis: | Basiswissen Codieren, DIMDI 2004 |
| BMGS, 2004: | ICD-10-Bekanntmachung des BMGS, http://www.zi-berlin.de/Zi_ICD10Browser/zi_icd_10_browser.htm |
| InEK, Codierrichtlinien: | Deutsche Codierrichtlinien – Version 2005, Institut für Entgeltsystem im Krankenhaus (InEK gGmbH) 2004, http://www.g-drg.de/service/download/veroeff_2005/DKR2005_Endversion_PDF30_040916_1500.pdf |
| HL7 Datentypen: | HL7 Version 3 Datentypen und CMETs für das Deutsche Gesundheitswesen, www.hl7.de (Publikationen) |
| CDAr2Arztbrief: | Arztbrief auf Basis der HL7 Clinical Document Architecture Release 2 für das deutsche Gesundheitswesen, Version 1.50 vom 12.05.2006, herausgegeben vom VHitG, HL7 Deutschland und der Arbeitsgemeinschaft Sciphox, www.hl7.de (Publikationen)
http://www.hl7.de/download/documents/cdar2-arztbrief/Leitfaden-VHitG-Arztbrief-v150.pdf |
| Wiley: | TNM-System: Wiley Interscience |
Zeitangaben
In einem Bericht tauchen mehrere Zeitangaben auf, die hier einmal in Form einer Übersicht dargestellt werden sollen. Die Angaben in der Bedingung beziehen sich auf die Nummern aus der ersten Spalte, d.h. hierüber wird eine Reihenfolge etabliert:
| # | Datum | Art | Bedingung (bezogen auf #) |
im Krankenhaus | im Labor | HL7 V3 (CDA später) |
|---|---|---|---|---|---|---|
| 1 | Auftragserfassung | Beginn | x | Order | ||
| 2 | Auftragserfassung | Ende | 1 < 2 | x | ||
| 3 | Auftragsfreigabe | TS | 2 < 3 | x | ||
| 4 | Auftragsübermittlung | TS | 3 < 4 | x | ||
| 5 | Probenentnahme | von/bis | x | Specimen Specimen Collection Process | ||
| 6 | Probenversand (Ausgang) | TS | 5 < 6 | x | ||
| 7 | Auftragseingang | TS | 4 < 7 | x | Acknowledgement | |
| 8 | Auftragsbestätigung | TS | 7 < 8 | x | Promise | |
| 9 | Probeneingang | TS | 6 < 9 | x | Specimen-> Specimen Process Step | |
| 10 | Probenuntersuchung | Beginn | 9 < 10 7 < 10 |
x | documentationOf ServiceEvent->
Specimen | |
| 11 | Probenuntersuchung | Ende | 10 < 11 | x | ||
| 12 | Befundung | Beginn | 10 < 12 | x | Specimen-> ObservationEvent
author.time | |
| 13 | Befundung | Ende | 12 < 13 | x | ||
| 14 | Niederschrift Befund | Beginn | 13 < 14 | x | dataEnterer.time | |
| 15 | Niederschrift Befund | Ende | 14 < 15 | x | ||
| 16 | Freigabe Befund | TS | 15 < 16 | x | legalAuthenticator .time | |
| 17 | Übermittlung Befund | TS | 16 < 17 | x | Wrapper | |
| 18 | Befundeingang | TS | 17 < 18 | x | Wrapper | |
| 19 | Befund gelesen | TS | 18 < 19 | x | - |
Freigaben können mehrstufig erfolgen.
Mehrere Berichte in Abhängigkeit des Prozessschrittes
Attribut-Wert-Paare
Die Attribut-Wert-Paare werden textuell aus den codierten Informationen abgeleitet (derived).
Hierzu gehören auch die Cancer Checklists.
Die zu verwendenden Vokabularien sind im Anhang detailliert aufgelistet.
Die Informationen werden als Attribut-Wert-Paare in Form einer Tabelle dargestellt, die wie folgt aussieht.
| Entnahme | Resektat |
|---|---|
| Kalk histologisch | Ja |
| Kalk (mm) | 0,2 |
Oder in XML:
<section>
<!-- Darstellung als Tabelle -->
<text>
<tbody>
<tr>
<td><content ID="d1">Entnahme</content></td>
<td>Resektat</td>
</tr>
<tr>
<td><content ID="d2">Kalk Histologisch</content></td>
<td>Ja</td>
</tr>
<tr>
<td><content ID="d3">Kalk (mm)</content></td>
<td>0,2</td>
</tr>
...
</tbody>
</text>
<!-— erste Information -->
<entry typeCode="DRIV">
<observation classCode="OBS" moodCode="EVN">
<code code"Mamma.Entnahme" codeSystem="??????" />
<value xsi:type="CD" code="Resektat" codeSystem="????">
<originalText><reference value="#d1"/></originalText>
</value>
</observation>
</entry>
<!-— zweite Information -->
<entry typeCode="DRIV">
<observation>
<code code="Mamma.Kalk Histologisch" codeSystem="??????" />
<value xsi:type="BL" code="true">
<originalText><reference value="#d2"/></originalText>
</value>
</observation>
</entry>
<!-— dritte Information -->
<entry typeCode="DRIV">
<observation classCode="OBS" moodCode="EVN">
<code code="Mamma.Kalk"
codeSystem="a.b.c.dx.y.z"
displayName="Kalk" />
<value xsi:type="PQ" value="0,2" unit="mm" >
<originalText><reference value="#d3"/></originalText>
</value>
</observation>
</entry>
<!—weitere Information -->
...
</section>
Anlagen
Digitale Bilder
Makroskopische sowie mikroskopische Bilder und Virtual Slides müssen eingebunden werden können. Lösungen über DICOM Supplement 122: Specimen Module and Revised Pathology SOP Classes