Einleitung
Foemig (Diskussion | Beiträge) |
Foemig (Diskussion | Beiträge) K |
||
| Zeile 71: | Zeile 71: | ||
====Fokus==== | ====Fokus==== | ||
Dieser Leitfaden konzentriert sich auf die Umsetzung des Analysemodells (s.u.) in HL7 V3 CDA R2. | Dieser Leitfaden konzentriert sich auf die Umsetzung des Analysemodells (s.u.) in HL7 V3 CDA R2. | ||
| + | |||
| + | [[Kategorie:cdaonk|Einleitung]] | ||
Aktuelle Version vom 10. März 2013, 15:37 Uhr
Dieses Material ist Teil des Leitfadens Übermittlung onkologischer Daten.
|
Inhaltsverzeichnis
Einleitung
Hintergrundinformationen
Im Sommer 2008 traf sich eine kleine Arbeitsgruppe (DOC, ADT, Agfa, Siemens, megapharm, alcedis) bei der DOC Holding in Düsseldorf, um die Optimierung der Datenübertragung in der onkologischen Versorgung zu besprechen. Hintergrund ist die in jedem Bundesland unterschiedliche Spezifikation der epidemiologischen Krebs¬register sowie die verschiedenen Anforderungen der Daten¬übermittlung an klinische Krebsregister und Dienstleister aus der Qualitätssicherung, die zu Problemen und hohen Aufwendungen bei der Implementierung führen. Eine weitere Fragestellung ist die Vereinfachung der Datenübermittlung in der onkologischen Forschung.
Projekthistorie
Die Historie dieses Projektes ist im Anhang aufgeführt.
Scope
Ziel soll es sein, eine modellbasierte Grundlage zu schaffen, um die Daten¬übertragung innerhalb der Onkologie auf einen Standard abzubilden. Daten werden auf verschiedenen Ebenen gesammelt und kommuniziert. Die klinische Betreuung erfolgt noch weitestgehend gestützt auf Papier- oder unstrukturierten elektronischen Dokumenten. In strukturierter Form werden Daten jedoch in der Qualitätssicherung, den klinischen und epidemiologischen Krebsregistern sowie den Organzentren benötigt. Es existiert eine Reihe von Spezifikationen – teilweise bedingt durch gesetzliche Vorgaben auf Landes- und Bundesebene , die einen unterschiedlichen organisatorischen Kontext besitzen und bei ähnlichen Inhalten nicht unbedingt deckungsgleich sind. Praktisch führt dies zu Mehrfachdokumentation und / oder aufwendigen technischen Systemen.
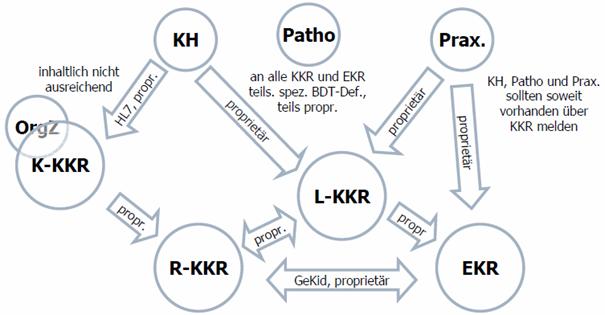
Abbildung 1: gegenwärtige Situation
Die vorhergehende Abbildung verdeutlicht die gegenwärtige Situation. Demnach gilt es folgende Spezifikationen abzubilden/abzulösen:
- Gemeinsamer onkologischer Basisdatensatz (ADT, GeKiD, DKG, KoQK, CCC Forum) auf Basis von XML
- Schnittstellen KKR / EKR (GeKiD)
- ONkeyLINE
- Datensatz Onkologie
- Spezielle Programme: BQS, DMP Mamma, QS / Benchmarking, ..
Als Grundproblem dieser Spezifikationen kann festgehalten werden, dass Prozesse und Rahmenbedingungen nicht ausreichend beachtet bzw. definiert worden sind: Anlass und Frequenz für Informationsfluss (Meldung), sinnvolle Teilmengen/Optionalitäten, lokale Besonderheiten (z.B. Landesgesetze), Verschlüsselung, technischer Transport, Verar¬beitungs¬status, Rückmeldung.
Als Idealfall ist anzustreben, dass jeder an der Behandlung beteiligte seinen inhaltlichen Anteil am Standard einmalig dokumentiert und dabei allenfalls Zusatzinformationen hinterlegt (wie die Zuordnung zu einer bestimmten Erkrankung oder therapeutischen Situation), die es erlauben, diese Informationen in übergeordnetem Kontext (Register, QS, nachbetreuende Behandler) korrekt einzuordnen.
Dieser Leitfaden zielt darauf ab, geeignete Dokumentenstrukturen für die einzelnen Meldungen auf Basis von Komponenten (Content Modules) zu spezifizieren, die die genannten Anforderungen abbilden können.
Basisdokumente
Dieses Dokument versucht die in den nachfolgenden Dokumenten aufgeführten Anforderungen umzusetzen:
- VHitG-Arztbrief (OID ???????)
- Header-Information
- Darstellung von Ärzten, Adressen, Namen, ..
- Grundlagen von Diagnosen und Prozeduren
- Diagnoseleitfaden (OID ??????)
- verfeinerte Diagnose-Information, ICD-O, TNM
- Liste der bisherigen Spezifikationen:
- XML-/CSV-Datensatz des Kooperationsverbundes Qualitätssicherung durch Klinische Krebsregister (KoQK) bzw. der Arbeitsgemeinschaft Deutscher Tumorzentren e.V. (ADT)
- XML-/CSV-Datensatz der Gesellschaft der epidemiologischen Krebsregister in Deutschland e.V. (GEKID)
- CSV-Datensatz des Instituts für angewandte Qualitätsförderung und Forschung im Gesundheitswesen GmbH (AQUA)
- XML-Datensätze der Deutsche Onkologie Centrum Holding GmbH
- Liste der noch nicht berücksichtigten Spezifikationen:
- CSV-Datensatz des GEKID-Datensatzes der Bundesgeschäftsstelle Qualitäts¬sicherung gGmbH (BQS)
- CSV-Datensatz der Gesellschaft für Pädiatrische Onkologie und Häma¬tologie (GPOH)
- BDT-Datensatz des Zentralinstituts für Kassenärztliche Versorgung (ZI)
- XML-Datensatz der Kassenärztlichen Bundesvereinigung (KBV)
- CDA2-Datensatz der Ärztekammer Westfalen-Lippe in Kooperation mit der Bundesgeschäftsstelle Qualitätssicherung gGmbH (BQS)
- XML-Datensatz der Arbeitsgruppe Tumordatenschnittstellen Niedersachsen
Aufbau
Nach dieser Einleitung werden die abzuhandelnden Szenarien beschrieben. Danach erfolgt eine Erläuterung des Analysemodells, das anhand der bisherigen Datensatzbeschreibungen und der dahinter liegenden medizinischen Informationen erarbeitet wurde. Daran schließen sich das dynamische Modell sowie das statische Modell mit der Definition der CDA-Strukturen (Header + Body) an. Im Anhang befinden sich Referenzen, eine offene Punkteliste, Verzeichnisse etc.
HL7 und Referenz-Modelle
Wie später noch weiter ausgeführt hat die Gruppe entschieden, die Umsetzung auf Basis von HL7 CDA zu realisieren. Daher sind die Basisspezifikationen des VHitG-Arztbriefes sowie des Diagnoseleitfadens relevant. Für hilfreiche Erläuterungen sei auf die entsprechenden Dokumente verwiesen.
Konzept und Begründung
Hier folgt das Konzept mit der dazu passenden Begründung des Leitfadens. Inhalt sollten der Zweck, die beteiligten Organisationen, ihre Rollen bei der Erstellung des Leitfadens und der Fokus des Dokuments sein.
Zweck
Dieser Leitfaden soll primär die Erst- und Folgemeldungen an die klin. und epid. Krebs¬register spezifizieren.
Fokus
Dieser Leitfaden konzentriert sich auf die Umsetzung des Analysemodells (s.u.) in HL7 V3 CDA R2.