Remmele-Score Entry (Template)
Foemig (Diskussion | Beiträge) K |
Foemig (Diskussion | Beiträge) K |
||
| Zeile 34: | Zeile 34: | ||
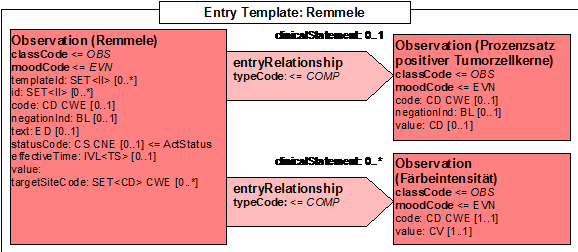
Als Grundlage dient das generische Modell, das der Einfachheit halber hier noch einmal wiederholt wird: | Als Grundlage dient das generische Modell, das der Einfachheit halber hier noch einmal wiederholt wird: | ||
| − | [[file: | + | [[file:cdaab2_remmele.gif]] |
===Attribute=== | ===Attribute=== | ||
Aktuelle Version vom 28. November 2013, 14:37 Uhr
Dieses Material ist Teil des Leitfadens Übermittlung onkologischer Daten.
|
Inhaltsverzeichnis
Entry: Remmele-Score
| Template-Metadaten | |
| Template-Typ | Entry |
| Template ID | 1.2.276.0.76.3.1.131.1.10.3.9xxxxx |
| generischeres Template | Assessment Scales |
| genutztes Template | |
| nutzende Templates | |
| abgeleitete Templates | |
| Schwester-Templates | |
| Generelle Beschreibung | Remmele-Score |
| allg. Erläuterung | Scores_und_Assessments |
| Ballotierungsstatus | in Arbeit |
| Erweiterbarkeit | geschlossen |
Modell
Als Grundlage dient das generische Modell, das der Einfachheit halber hier noch einmal wiederholt wird:
Attribute
| Lvl | RIM | Name | DT | Kard | Conf | Beschreibung |
|---|---|---|---|---|---|---|
| 3 | act | observation | 1..1 | M | Remmele-Score fix: @classCode="OBS" | |
| 4 | act | templateId | II | 1..1 | F | @root="1.2.276.0.76.3.1.131.1.10.3.xxxx" |
| 4 | act | id | II | 0..1 | O | ID der diagnostischen Angabe @root=OID des sendenden System, um diagnostische Angaben eindeutig zu identifizieren |
| 4 | act | code | CD CWE | 1..1 | F | @code="tbd" @codeSystem=LOINC: "tbd" |
| 4 | act | text | ED | 1..1 | R | textliche Beschreibung des codierten Inhalts der Observation; wird i.d.R. vom sendenden System automatisch generiert |
| 4 | act | effectiveTime | TS | 0..1 | O | Das Datum, an dem die Diagnose gestellt wurde |
| 4 | act | value | CD | 1..1 | R | Remmele-Score Value Set: |
| 4 | act | interpretationCode | CE | 1..1 | R | |
| 5 | act | entryRelationship | 0..1 | O | @typeCode="COMP" | |
| 6 | act | observation | 1..1 | M | Prozentsatz positiver Tumorzellkerne | |
| 7 | act | code | 1..1 | F | @code=LOINC: "444901007"
@codeSystem=LOINC: "2.16.840.1.113883.6.96" | |
| 6 | act | value | CV | 1..1 | R | Value Set: |
| 5 | act | entryRelationship | 0..1 | O | @typeCode="COMP" | |
| 6 | act | observation | 1..1 | M | Färbeintensität | |
| 6 | act | code | CV | 1..1 | F | @code=LOINC: " 444775005" @codeSystem=LOINC: "2.16.840.1.113883.6.96" |
| 6 | act | value | CV | 1..1 | R | Value Set: |
Beispiel
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.276.0.76.3.1.131.1.10.3.xxxx"/>
...
</observation>
Vokabular
Hormonrezeptorexpression nach Remmele-Stegner (Remmele-Score)
Code:
@code="TBD"
@codeSystem="TBD"
Value Set (OID TODO - Code System ?)
Die immunhistochemisch nachweisbare Rezeptorexpression für Östrogen- oder Progesteronrezeptoren (ER und PgR) in Tumorzellen wird als Produkt des Prozentsatzes positiver Zellkerne und der mittleren Färbeintensität als Scorewert angegeben. Die jeweiligen Werte werden in vier bzw. drei Klassen sortiert, so dass der Score Werte zwischen 0 und 12 annehmen kann.
Prozentsatz positiver Tumorzellkerne
Code=444775005 Codesystem=2.16.840.1.113883.6.96
| Code | Kriterien |
|---|---|
| 0 | 0% |
| 1 | 1-10% |
| 2 | 11-50% |
| 3 | 51-80% |
| 4 | >80% |
Färbeintensität
Code=444775005 Codesystem=2.16.840.1.113883.6.96
| Code | Kriterien |
|---|---|
| 0 | keine |
| 1 | schwach |
| 2 | mäßig |
| 3 | stark |
Interpretation
Code: @codeSystem="2.16.840.1.113883.5.83"
Value Set 2.16.840.1.113883.1.11.78
| ObservationInterpretationSusceptibility | ObservationRange | Interpretation |
|---|---|---|
| R | 0 | nicht hormonsensitiv |
| MS | 2 | fraglich hormonsensitiv |
| S | 3-12 | hormonsensitiv |