Residualtumor-Entry (Template)
Foemig (Diskussion | Beiträge) K |
(→Modell) |
||
| (27 dazwischenliegende Versionen von 4 Benutzern werden nicht angezeigt) | |||
| Zeile 2: | Zeile 2: | ||
==Entry: R-Klassifikation== | ==Entry: R-Klassifikation== | ||
| + | |||
{| class="hl7table" | {| class="hl7table" | ||
| − | |bgcolor="ddddff"|Template | + | |bgcolor="ddddff"| || bgcolor="ddddff"| Template-Metadaten |
| + | |- | ||
| + | |bgcolor="ddddff"|Template-Typ|| Entry | ||
| + | |- | ||
| + | |bgcolor="ddddff"|Template ID|| 1.2.276.0.76.3.1.131.1.10.3.7 | ||
| + | |- | ||
| + | |bgcolor="ddddff"|generischeres Template || | ||
| + | |- | ||
| + | |bgcolor="ddddff"|genutztes Template || | ||
| + | |- | ||
| + | |bgcolor="ddddff"|nutzende Templates || | ||
| + | |- | ||
| + | |bgcolor="ddddff"|abgeleitete Templates || | ||
| + | |- | ||
| + | |bgcolor="ddddff"|Schwester-Templates || | ||
| + | |- | ||
| + | |bgcolor="ddddff"|generelle Beschreibung|| R-Klassifikation (Residualtumor) | ||
| + | |- | ||
| + | |bgcolor="ddddff"|allg. Erläuterung|| | ||
|- | |- | ||
| − | |bgcolor="ddddff"| | + | |bgcolor="ddddff"|Verhältnis zu IHE|| neu |
| + | |- | ||
| + | |bgcolor="ddddff"|Ballotierungsstatus|| in Arbeit | ||
| + | |- | ||
| + | |bgcolor="ddddff"|Erweiterbarkeit|| geschlossen | ||
|} | |} | ||
| − | + | ===Beschreibung=== | |
| − | + | ||
| − | + | Die R-Klassifikation beschreibt den nach therapeutischen Maßnahmen (insbesondere Operationen) im Körper verbliebenen Residualtumor. Sie bezieht sich aber nicht nur auf Operationen, in deren Kontext sie allerdings die praktisch größte Bedeutung hat, sondern berücksichtigt teilweise auch klinische Angaben. | |
| − | + | ||
| − | < | + | Die sogenannte globale R-Klassifikation betrachtet die gesamte Erkrankung, also sowohl das lokale Geschehen als auch etwaige Fernmetastasen. Diese Klassifikation führt der betreuende Kliniker unter Berücksichtigung etwaiger Befunde der Pathologen durch. |
| − | + | ||
| − | }} | + | Die lokale Radikalität einer Operation wird gleichartig codiert, betrachtet aber nur das Resektat und wird durch den Pathologen bestimmt. |
| + | |||
| + | <b>Hinweis:</b> Es existieren zwei semantisch widersprüchliche Definitionen für die Codierung R2a/R2b bzw. R2a/R2b/R2c. Hier findet die erstgenannte Verwendung (vgl. Value Set). Die letztgenannte Definition, entstammend dem TNM-Supplement (4. Auflage), lässt sich wie folgt übersetzen: | ||
| + | |||
| + | * R2a: R2 mit Qualifier 'Vorhandensein Residualtumor' = "L" | ||
| + | * R2b: R2 mit Qualifier 'Vorhandensein Residualtumor' = "M" | ||
| + | * R2c: R2 mit Qualifier 'Vorhandensein Residualtumor' = "B" | ||
| + | |||
| + | {{WorkBox|Die vorgenannte Darstellungsweise zur Auflösung der widersprüchlichen Definition befindet sich noch in Diskussion und ist daher als vorläufig zu betrachten. }} | ||
| + | |||
| + | ===Modell=== | ||
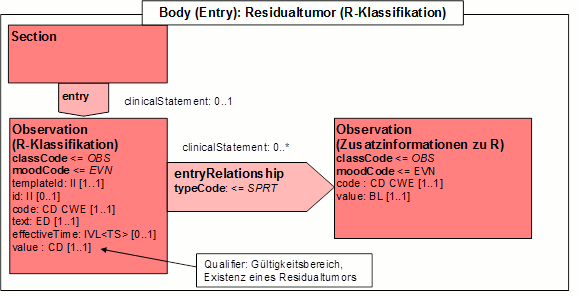
[[file:Cdaonk_r_klassifikation.gif|R-Klassifikation]] | [[file:Cdaonk_r_klassifikation.gif|R-Klassifikation]] | ||
| + | |||
| + | {{AlertBox| | ||
| + | TODO: R-Status ist Teil von TNM. Abgleich mit TNM-Template aus APSR 2.0 erforderlich! | ||
| + | }} | ||
Abbildung: R-Klassifikation | Abbildung: R-Klassifikation | ||
| + | ===Attribute=== | ||
{| class="hl7table" | {| class="hl7table" | ||
| Zeile 27: | Zeile 65: | ||
! RIM | ! RIM | ||
! Name | ! Name | ||
| − | |||
! DT | ! DT | ||
! Kard | ! Kard | ||
| Zeile 38: | Zeile 75: | ||
| observation | | observation | ||
| | | | ||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | | | + | | @classCode="OBS", @moodCode="EVN" |
|- | |- | ||
| Zeile 67: | Zeile 83: | ||
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| templateId | | templateId | ||
| − | |||
| II | | II | ||
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | + | | @root="1.2.276.0.76.3.1.131.1.10.3.7" | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | | @root | ||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
|- | |- | ||
| Zeile 87: | Zeile 92: | ||
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| id | | id | ||
| − | |||
| II | | II | ||
| 0..1 | | 0..1 | ||
| − | | | + | | O |
| − | | | + | | ID der diagnostischen Angabe |
|- | |- | ||
| Zeile 97: | Zeile 101: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|@root | |@root | ||
| − | |||
| | | | ||
|1..1 | |1..1 | ||
| − | | | + | |O |
|OID des sendenden System, um diagnostische Angaben eindeutig zu identifizieren | |OID des sendenden System, um diagnostische Angaben eindeutig zu identifizieren | ||
| Zeile 107: | Zeile 110: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|@extension | |@extension | ||
| − | |||
| | | | ||
|1..1 | |1..1 | ||
| − | | | + | |O |
| − | | | + | |ID der diagnostischen Angabe im sendenden System |
|- | |- | ||
| 4 | | 4 | ||
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| − | | code | + | | code |
| − | |||
| CD CWE | | CD CWE | ||
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | + | | @code="tnmR", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |@code | ||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
|- | |- | ||
| Zeile 147: | Zeile 128: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|text | |text | ||
| − | |||
|ED | |ED | ||
|1..1 | |1..1 | ||
| − | | | + | |R |
| − | |i.d.R. vom sendenden System automatisch generiert | + | |textliche Beschreibung des codierten Inhalts der Observation; i.d.R. vom sendenden System automatisch generiert |
|- | |- | ||
| Zeile 157: | Zeile 137: | ||
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| effectiveTime | | effectiveTime | ||
| − | |||
| TS | | TS | ||
| 0..1 | | 0..1 | ||
| − | | | + | | O |
| − | | Das Datum, an dem die Diagnose gestellt wurde | + | | Das Datum, an dem die Diagnose (fest)gestellt wurde |
|- | |- | ||
| Zeile 167: | Zeile 146: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|value | |value | ||
| − | |||
|CD | |CD | ||
|1..1 | |1..1 | ||
| − | | | + | |R |
| − | | | + | |Value Set 1.2.276.0.76.11.4 |
|- | |- | ||
|5 | |5 | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
| − | | | + | |qualifier |
| − | | | + | |CR |
| − | | | + | |0..1 |
| − | |1..1 | + | |O |
| − | | | + | |Gültigkeitsbereich. |
| − | | | + | Wird dieser Qualifier nicht übermittelt, so gilt implizit der Code "G" (gültig für die Gesamtsituation der Erkrankung). |
| + | |||
| + | |- | ||
| + | | 6 | ||
| + | |bgcolor="ff8888"| act | ||
| + | | name | ||
| + | | CV | ||
| + | | 1..1 | ||
| + | | F | ||
| + | |@code="tnmRDomain", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" | ||
|- | |- | ||
| − | | | + | |6 |
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
| − | | | + | |value |
| − | + | |CV | |
| − | | | ||
|1..1 | |1..1 | ||
| − | | | + | |R |
| − | |fix | + | |TODO: Value Set OID definieren: fix: @codeSystem="1.2.276.0.76.5.???" |
| + | {{WorkBox|TODO: die OID .414 für das Codesystem wurde doppelt vergeben (auch für OPS 2013!) }} | ||
|- | |- | ||
| Zeile 197: | Zeile 184: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|qualifier | |qualifier | ||
| − | |||
|CR | |CR | ||
|0..1 | |0..1 | ||
| − | | | + | |O |
| − | | | + | |Existenz eines Residualtumors. |
| + | Dieser Qualifier darf nur bei globaler Gültigkeit der R-Klassifikation übermittelt werden | ||
|- | |- | ||
| Zeile 207: | Zeile 194: | ||
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| name | | name | ||
| − | |||
| CV | | CV | ||
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | | | + | |@code="tnmRLocation", @codeSystem="1.2.276.0.76.3.1.131.1.5.1" |
| − | |||
|- | |- | ||
| − | | | + | |6 |
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
| − | | | + | |value |
| − | + | |CV | |
| − | | | ||
|1..1 | |1..1 | ||
| − | | | + | |R |
| − | |fix | + | |TODO: Value Set OID vergeben. fix: @codeSystem="1.2.276.0.76.5.???" |
| + | Die explizite Angabe für unbekannt wird durch @nullFlavor="UNK" ausgedrückt. | ||
| + | {{WorkBox|TODO: die OID .415 für das Codesystem wurde doppelt vergeben (auch für Alpha-ID 2013!) }} | ||
| + | |||
| + | |- | ||
| + | | 4 | ||
| + | |bgcolor="ffaaaa"| rel | ||
| + | | entryRelationship | ||
| + | | | ||
| + | | 0..n | ||
| + | | F | ||
| + | | @typeCode="SPRT" | ||
| + | |||
| + | |- | ||
| + | | 5 | ||
| + | |bgcolor="ff8888"| act | ||
| + | | observation | ||
| + | | | ||
| + | | 1..1 | ||
| + | | R | ||
| + | | Zusatzinformationen zur R-Klassifikation<br> | ||
| + | Jeder Zusatzinformation (Code) darf maximal ein Mal übermittelt werden. Wird die entsprechende Angabe nicht übermittelt, gilt implizit der Wert "false"<br> | ||
| + | fix: @classCode="OBS", @moodCode="EVN" | ||
|- | |- | ||
| − | | | + | | 6 |
| − | |bgcolor="ff8888"|act | + | |bgcolor="ff8888"| act |
| − | | | + | | code |
| − | + | | CD CWE | |
| − | | | + | | 1..1 |
| − | |1..1 | + | | R |
| − | | | + | | vgl. Tabelle "Zusatzinformationen zur R-Klassifikation". |
| − | | | ||
|- | |- | ||
| Zeile 238: | Zeile 243: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|value | |value | ||
| − | | | + | |BL |
| − | |||
|1..1 | |1..1 | ||
| − | | | + | |R |
| | | | ||
| + | |||
| + | |} | ||
| + | |||
| + | |||
| + | ===Verwendete Terminologie=== | ||
| + | |||
| + | ====Zusatzinformationen ==== | ||
| + | |||
| + | {| class="hl7table" | ||
| + | ! Code | ||
| + | ! Suffix-Symbol | ||
| + | ! Bedeutung | ||
|- | |- | ||
| − | | | + | | residualIs |
| − | | | + | | (is) |
| − | + | | invasives Karzinom komplett entfernt, aber in-situ Komponente am Schnittrand | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | | | ||
|- | |- | ||
| − | | | + | | residualCy+ |
| − | | | + | | (cy+) |
| − | + | | bei Spültechniken zytologisch entdeckte Tumorzellen | |
| − | |||
| − | |||
| − | |||
| − | |||
| − | | | ||
|- | |- | ||
| − | | | + | | residualUn |
| − | | | + | | (un) |
| − | + | | unsichere R0 Resektion (TNM-Supplement, 4. Auflage, Seite 25) | |
| − | |||
| − | |||
| − | | | ||
| − | |||
| − | |||
|- | |- | ||
| − | | | + | | residualGt1mm |
| − | | | + | | >1mm |
| − | | | + | | R0, Abstand Resektionsrand > 1mm, entspricht CRM negative (TNM-Supplement, 4. Auflage, Seite 25(??) |
| − | |||
| − | |||
| − | |||
| − | |||
| − | |||
|- | |- | ||
| − | | | + | | residualLt1mm |
| − | + | | <1mm | |
| − | + | | R0, Abstand Resektionsrand < 1mm, entspricht CRM positive (TNM-Supplement, 4. Auflage, Seite 25(??) | |
| − | |||
| − | | | ||
| − | | | ||
| − | |||
| − | |||
|- | |- | ||
| − | | | + | | residualDir |
| − | | | + | | -dir |
| − | | | + | | Tumor direkt am Resektionsrand durchschnitten |
| − | + | ||
| − | + | |} | |
| − | + | Tabelle: Zusatzinformationen zur R-Klassifikation<br> | |
| − | + | Value Set (OID TODO)<br> | |
| − | + | Codesystem: 1.2.276.0.76.3.1.131.1.5.1 | |
| + | |||
| + | |||
| + | |||
| + | ====Residualtumor==== | ||
| + | {| class="hl7table" | ||
| + | !Code!!Description!!Bedeutung | ||
|- | |- | ||
| − | | | + | |R0||No residual tumour||kein Residualtumor |
| − | | | + | |- |
| − | | | + | |R0a||||kein Residualtumor, Tumormarker normal |
| − | | | + | |- |
| − | | | + | |R0b||||kein Residualtumor, Tumormarker erhöht |
| − | |1..1 | + | |- |
| − | + | |R1||Microscopic residual tumour||nur mikroskopisch nachweisbarer Residualtumor | |
| − | + | |- | |
| + | |R2||Macroscopic residual tumour||makroskopischer Residualtumor | ||
| + | |- | ||
| + | |R2a||||makroskopischer Residualtumor, mikroskopisch nicht bestätigt | ||
| + | |- | ||
| + | |R2b||||makroskopischer Residualtumor, mikroskopisch bestätigt | ||
| + | |- | ||
| + | |RX||Presence of residual tumour cannot be assessed||Unbekannt | ||
| + | |- | ||
| + | |} | ||
| + | Tabelle: Residualtumor-Codes <br> | ||
| + | Value Set (OID 1.2.276.0.76.11.4)<br> | ||
| + | UICC 5. Auflage (OID 2.16.840.1.113883.15.8)<br> | ||
| + | UICC 6. Auflage (OID 2.16.840.1.113883.15.7)<br> | ||
| + | UICC 7. Auflage (OID 2.16.840.1.113883.15.6) | ||
| + | |||
| + | ====Gültigkeitsbereich der Beurteilung des Residualtumors==== | ||
| + | Auf welchen Bereich bezieht sich die Beurteilung? | ||
| + | {| class="hl7table" | ||
| + | !Code!!Bedeutung | ||
|- | |- | ||
| − | | | + | |L||Lokoregionärer Tumor |
| − | | | + | |- |
| − | | | + | |M||Fernmetastasen |
| − | | | + | |- |
| − | | | + | |G||Gesamtsituation der Erkrankung |
| − | | | + | |} |
| − | | | + | Tabelle: Gültigkeitsbereich-Codes <br> |
| − | + | Value Set (OID TODO)<br> | |
| + | Codesystem: "1.2.276.0.76.5.???" (TODO: vorläufig, die OID .414 wurde doppelt vergeben!) | ||
| + | |||
| + | |||
| + | |||
| + | ====Vorhandensein Residualtumor==== | ||
| + | Wo ist der Residualtumor lokalisiert? | ||
| + | {| class="hl7table" | ||
| + | !Code!!Bedeutung | ||
| + | |- | ||
| + | |L||Lokoregionärer Tumor | ||
|- | |- | ||
| − | | | + | |M||Fernmetastasen |
| − | | | + | |- |
| − | | | + | |B||beides |
| − | + | |} | |
| − | + | Tabelle: Gültigkeitsbereich-Codes <br> | |
| − | + | Value Set (OID TODO)<br> | |
| − | + | Codesystem: "1.2.276.0.76.5.???" (TODO: vorläufig, die OID .415 wurde doppelt vergeben!) | |
| − | + | ||
| − | + | ===Beispiel=== | |
<syntaxhighlight lang="XML"> | <syntaxhighlight lang="XML"> | ||
| Zeile 356: | Zeile 382: | ||
<name code="tnmRLocation" | <name code="tnmRLocation" | ||
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | codeSystem="1.2.276.0.76.3.1.131.1.5.1"/> | ||
| − | <value xsi:type="CD" code=" | + | <value xsi:type="CD" code="L" |
codeSystem="1.2.276.0.76.5.415"/> | codeSystem="1.2.276.0.76.5.415"/> | ||
</qualifier> | </qualifier> | ||
</value> | </value> | ||
| + | <entryRelationship typeCode="SPRT"> | ||
| + | <observation classCode="OBS" moodCode="EVN"> | ||
| + | <code code="residualIs" | ||
| + | codeSystem="1.2.276.0.76.3.1.131.1.5.1 "/> | ||
| + | <value xsi:type="BL" value="true"/> | ||
| + | </observation> | ||
| + | </entryRelationship> | ||
| + | <entryRelationship typeCode="SPRT"> | ||
| + | <observation classCode="OBS" moodCode="EVN"> | ||
| + | <code code="residualGt1mm" | ||
| + | codeSystem="1.2.276.0.76.3.1.131.1.5.1 "/> | ||
| + | <value xsi:type="BL" value="true"/> | ||
| + | </observation> | ||
| + | </entryRelationship> | ||
| + | <entryRelationship typeCode="SPRT"> | ||
| + | <observation classCode="OBS" moodCode="EVN"> | ||
| + | <code code="residualCy+" | ||
| + | codeSystem="1.2.276.0.76.3.1.131.1.5.1 "/> | ||
| + | <value xsi:type="BL" value="false"/> | ||
| + | </observation> | ||
| + | </entryRelationship> | ||
</observation> | </observation> | ||
</syntaxhighlight> | </syntaxhighlight> | ||
| + | [[Kategorie:cdaonk|R-Klassfikation]] | ||
[[Kategorie:CDA Entry Level Template|R-Klassfikation]] | [[Kategorie:CDA Entry Level Template|R-Klassfikation]] | ||
Aktuelle Version vom 28. September 2017, 13:15 Uhr
Dieses Material ist Teil des Leitfadens Arztbrief 2.x.
|
Inhaltsverzeichnis
Entry: R-Klassifikation
| Template-Metadaten | |
| Template-Typ | Entry |
| Template ID | 1.2.276.0.76.3.1.131.1.10.3.7 |
| generischeres Template | |
| genutztes Template | |
| nutzende Templates | |
| abgeleitete Templates | |
| Schwester-Templates | |
| generelle Beschreibung | R-Klassifikation (Residualtumor) |
| allg. Erläuterung | |
| Verhältnis zu IHE | neu |
| Ballotierungsstatus | in Arbeit |
| Erweiterbarkeit | geschlossen |
Beschreibung
Die R-Klassifikation beschreibt den nach therapeutischen Maßnahmen (insbesondere Operationen) im Körper verbliebenen Residualtumor. Sie bezieht sich aber nicht nur auf Operationen, in deren Kontext sie allerdings die praktisch größte Bedeutung hat, sondern berücksichtigt teilweise auch klinische Angaben.
Die sogenannte globale R-Klassifikation betrachtet die gesamte Erkrankung, also sowohl das lokale Geschehen als auch etwaige Fernmetastasen. Diese Klassifikation führt der betreuende Kliniker unter Berücksichtigung etwaiger Befunde der Pathologen durch.
Die lokale Radikalität einer Operation wird gleichartig codiert, betrachtet aber nur das Resektat und wird durch den Pathologen bestimmt.
Hinweis: Es existieren zwei semantisch widersprüchliche Definitionen für die Codierung R2a/R2b bzw. R2a/R2b/R2c. Hier findet die erstgenannte Verwendung (vgl. Value Set). Die letztgenannte Definition, entstammend dem TNM-Supplement (4. Auflage), lässt sich wie folgt übersetzen:
- R2a: R2 mit Qualifier 'Vorhandensein Residualtumor' = "L"
- R2b: R2 mit Qualifier 'Vorhandensein Residualtumor' = "M"
- R2c: R2 mit Qualifier 'Vorhandensein Residualtumor' = "B"
|
|
Die vorgenannte Darstellungsweise zur Auflösung der widersprüchlichen Definition befindet sich noch in Diskussion und ist daher als vorläufig zu betrachten. |
Modell
|
|
TODO: R-Status ist Teil von TNM. Abgleich mit TNM-Template aus APSR 2.0 erforderlich! |
Abbildung: R-Klassifikation
Attribute
Verwendete Terminologie
Zusatzinformationen
| Code | Suffix-Symbol | Bedeutung |
|---|---|---|
| residualIs | (is) | invasives Karzinom komplett entfernt, aber in-situ Komponente am Schnittrand |
| residualCy+ | (cy+) | bei Spültechniken zytologisch entdeckte Tumorzellen |
| residualUn | (un) | unsichere R0 Resektion (TNM-Supplement, 4. Auflage, Seite 25) |
| residualGt1mm | >1mm | R0, Abstand Resektionsrand > 1mm, entspricht CRM negative (TNM-Supplement, 4. Auflage, Seite 25(??) |
| residualLt1mm | <1mm | R0, Abstand Resektionsrand < 1mm, entspricht CRM positive (TNM-Supplement, 4. Auflage, Seite 25(??) |
| residualDir | -dir | Tumor direkt am Resektionsrand durchschnitten |
Tabelle: Zusatzinformationen zur R-Klassifikation
Value Set (OID TODO)
Codesystem: 1.2.276.0.76.3.1.131.1.5.1
Residualtumor
| Code | Description | Bedeutung |
|---|---|---|
| R0 | No residual tumour | kein Residualtumor |
| R0a | kein Residualtumor, Tumormarker normal | |
| R0b | kein Residualtumor, Tumormarker erhöht | |
| R1 | Microscopic residual tumour | nur mikroskopisch nachweisbarer Residualtumor |
| R2 | Macroscopic residual tumour | makroskopischer Residualtumor |
| R2a | makroskopischer Residualtumor, mikroskopisch nicht bestätigt | |
| R2b | makroskopischer Residualtumor, mikroskopisch bestätigt | |
| RX | Presence of residual tumour cannot be assessed | Unbekannt |
Tabelle: Residualtumor-Codes
Value Set (OID 1.2.276.0.76.11.4)
UICC 5. Auflage (OID 2.16.840.1.113883.15.8)
UICC 6. Auflage (OID 2.16.840.1.113883.15.7)
UICC 7. Auflage (OID 2.16.840.1.113883.15.6)
Gültigkeitsbereich der Beurteilung des Residualtumors
Auf welchen Bereich bezieht sich die Beurteilung?
| Code | Bedeutung |
|---|---|
| L | Lokoregionärer Tumor |
| M | Fernmetastasen |
| G | Gesamtsituation der Erkrankung |
Tabelle: Gültigkeitsbereich-Codes
Value Set (OID TODO)
Codesystem: "1.2.276.0.76.5.???" (TODO: vorläufig, die OID .414 wurde doppelt vergeben!)
Vorhandensein Residualtumor
Wo ist der Residualtumor lokalisiert?
| Code | Bedeutung |
|---|---|
| L | Lokoregionärer Tumor |
| M | Fernmetastasen |
| B | beides |
Tabelle: Gültigkeitsbereich-Codes
Value Set (OID TODO)
Codesystem: "1.2.276.0.76.5.???" (TODO: vorläufig, die OID .415 wurde doppelt vergeben!)
Beispiel
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.276.0.76.3.1.131.1.10.3.7"/>
<id extension="1234041"
root="1.2.276.0.76.3.1.131.1.4.3.9999.9999.999919"/>
<code code="tnmR" codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<text>
gesamt R1, Fernmetastasen vorhanden
</text>
<effectiveTime value="20110326"/>
<value xsi:type="CD" code="R1"
codeSystem="2.16.840.1.113883.15.6">
<qualifier>
<name code="tnmRDomain"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="G" codeSystem="1.2.276.0.76.5.414"/>
</qualifier>
<qualifier>
<name code="tnmRLocation"
codeSystem="1.2.276.0.76.3.1.131.1.5.1"/>
<value xsi:type="CD" code="L"
codeSystem="1.2.276.0.76.5.415"/>
</qualifier>
</value>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="residualIs"
codeSystem="1.2.276.0.76.3.1.131.1.5.1 "/>
<value xsi:type="BL" value="true"/>
</observation>
</entryRelationship>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="residualGt1mm"
codeSystem="1.2.276.0.76.3.1.131.1.5.1 "/>
<value xsi:type="BL" value="true"/>
</observation>
</entryRelationship>
<entryRelationship typeCode="SPRT">
<observation classCode="OBS" moodCode="EVN">
<code code="residualCy+"
codeSystem="1.2.276.0.76.3.1.131.1.5.1 "/>
<value xsi:type="BL" value="false"/>
</observation>
</entryRelationship>
</observation>