Gleason-Score-Entry (Template)
Foemig (Diskussion | Beiträge) K (hat „cdaonk:Gleason-Score-Entry (Template)“ nach „cdaab2:Gleason-Score-Entry (Template)“ verschoben) |
Foemig (Diskussion | Beiträge) K |
||
| Zeile 1: | Zeile 1: | ||
{{DocumentPart}} | {{DocumentPart}} | ||
| − | |||
| − | |||
| − | |||
| − | |||
==Entry: Gleason-Score== | ==Entry: Gleason-Score== | ||
{| class="hl7table" | {| class="hl7table" | ||
| − | |bgcolor="ddddff"|Template | + | |bgcolor="ddddff"| || bgcolor="ddddff"| Template-Metadaten |
| + | |- | ||
| + | |bgcolor="ddddff"|Template-Typ|| Entry | ||
| + | |- | ||
| + | |bgcolor="ddddff"|Template ID|| 1.2.276.0.76.3.1.131.1.10.3.9 | ||
| + | |- | ||
| + | |bgcolor="ddddff"|genutztes Template || [[cdaab2:Assessment_Scales_(Entry)]] | ||
| + | |- | ||
| + | |bgcolor="ddddff"|abgeleitete Templates || <Verweis auf das Template> | ||
|- | |- | ||
| − | |bgcolor="ddddff"| | + | |bgcolor="ddddff"|Generelle Beschreibung|| Gleason-Score (Prostatakarzinom) |
| + | |- | ||
| + | |bgcolor="ddddff"|allg. Erläuterung|| < Verweis auf eine allgemeine Erläuterungsseite > | ||
| + | |- | ||
| + | |bgcolor="ddddff"|Ballotierungsstatus|| < Status der Verabschiedung > | ||
| + | |- | ||
| + | |bgcolor="ddddff"|Erweiterbarkeit|| offen oder geschlossen | ||
|} | |} | ||
| + | |||
{{AlertBox| | {{AlertBox| | ||
| Zeile 29: | Zeile 40: | ||
gebildet. Es gibt die primären Gleason-Muster 1-5 (aufsteigende Malignität). | gebildet. Es gibt die primären Gleason-Muster 1-5 (aufsteigende Malignität). | ||
| − | [[ | + | [[file:GleasonMuster.jpg]] |
Der Gleason Score setzt sich beim Resektionspräparat aus der Summe des häufigsten und des zweithäufigsten, bei der Stanzbiopsie des schlechtesten und des zweitschlechtesten Gleason-Musters zusammen. Ist nur ein Muster erkennbar (häufig in Stanzbiopsien), so wird der Score aus dem verdoppelten Musterwert gebildet. An der Stanze sind Gleason-Muster <3 nicht zugelassen. | Der Gleason Score setzt sich beim Resektionspräparat aus der Summe des häufigsten und des zweithäufigsten, bei der Stanzbiopsie des schlechtesten und des zweitschlechtesten Gleason-Musters zusammen. Ist nur ein Muster erkennbar (häufig in Stanzbiopsien), so wird der Score aus dem verdoppelten Musterwert gebildet. An der Stanze sind Gleason-Muster <3 nicht zugelassen. | ||
| Zeile 36: | Zeile 47: | ||
Seit 2005 gilt die leicht modifizierte Gleason-Klassifizierung (nach Epstein \[Helpap2007\]), d.h. es ist hier die Angabe des verwendeten Verfahrens – genauer die zu verwendenden Codes - erforderlich. | Seit 2005 gilt die leicht modifizierte Gleason-Klassifizierung (nach Epstein \[Helpap2007\]), d.h. es ist hier die Angabe des verwendeten Verfahrens – genauer die zu verwendenden Codes - erforderlich. | ||
| − | + | ===Attribute=== | |
{| class="hl7table" | {| class="hl7table" | ||
| Zeile 42: | Zeile 53: | ||
! RIM | ! RIM | ||
! Name | ! Name | ||
| − | |||
! DT | ! DT | ||
! Kard | ! Kard | ||
| Zeile 52: | Zeile 62: | ||
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| observation | | observation | ||
| − | |||
| | | | ||
| 1..1 | | 1..1 | ||
| Zeile 62: | Zeile 71: | ||
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| @classCode | | @classCode | ||
| − | |||
| CS CNE | | CS CNE | ||
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | | fix | + | | fix: "OBS" |
|- | |- | ||
| Zeile 72: | Zeile 80: | ||
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| @moodCode | | @moodCode | ||
| − | |||
| CS CNE | | CS CNE | ||
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | | fix | + | | fix: "EVN" |
|- | |- | ||
| Zeile 82: | Zeile 89: | ||
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| templateId | | templateId | ||
| − | |||
| II | | II | ||
| 1..1 | | 1..1 | ||
| required | | required | ||
| − | | | + | | Gleason-Score |
|- | |- | ||
| Zeile 92: | Zeile 98: | ||
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| @root | | @root | ||
| − | |||
| | | | ||
| 1..1 | | 1..1 | ||
| − | | | + | | F |
| − | | fix | + | | fix: "1.2.276.0.76.3.1.131.1.10.3.9" |
|- | |- | ||
| Zeile 102: | Zeile 107: | ||
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| id | | id | ||
| − | |||
| II | | II | ||
| 0..1 | | 0..1 | ||
| optional | | optional | ||
| − | | | + | | ID der diagnostischen Angabe |
|- | |- | ||
| Zeile 112: | Zeile 116: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|@root | |@root | ||
| − | |||
| | | | ||
|1..1 | |1..1 | ||
| Zeile 122: | Zeile 125: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|@extension | |@extension | ||
| − | |||
| | | | ||
|1..1 | |1..1 | ||
|optional | |optional | ||
| − | | | + | |ID der diagnostischen Angabe im sendenden System |
|- | |- | ||
| Zeile 132: | Zeile 134: | ||
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| code | | code | ||
| − | |||
| CD CWE | | CD CWE | ||
| 1..1 | | 1..1 | ||
| Zeile 142: | Zeile 143: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|@code | |@code | ||
| − | |||
| | | | ||
|1..1 | |1..1 | ||
| − | | | + | |F |
| − | |fix | + | |fix: "35266-6" |
|- | |- | ||
| Zeile 152: | Zeile 152: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|@codeSystem | |@codeSystem | ||
| − | |||
| | | | ||
|1..1 | |1..1 | ||
| − | | | + | |F |
| − | |fix: LOINC | + | |fix: LOINC: "2.16.840.1.113883.6.1" |
|- | |- | ||
| Zeile 162: | Zeile 161: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|text | |text | ||
| − | |||
|ED | |ED | ||
|1..1 | |1..1 | ||
|required | |required | ||
| − | |i.d.R. vom sendenden System automatisch generiert | + | |textliche Beschreibung des codierten Inhalts der Observation; wird i.d.R. vom sendenden System automatisch generiert |
|- | |- | ||
| Zeile 172: | Zeile 170: | ||
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| effectiveTime | | effectiveTime | ||
| − | |||
| TS | | TS | ||
| 0..1 | | 0..1 | ||
| Zeile 182: | Zeile 179: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|value | |value | ||
| − | |||
|CD | |CD | ||
|1..1 | |1..1 | ||
|required | |required | ||
| − | | | + | |Code für das Gleason-Score |
|- | |- | ||
| Zeile 192: | Zeile 188: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|@code | |@code | ||
| − | |||
| | | | ||
|1..1 | |1..1 | ||
| − | | | + | |F |
|Codesystem 1.2.276.0.76.5.404 | |Codesystem 1.2.276.0.76.5.404 | ||
| Zeile 202: | Zeile 197: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|@codeSystem | |@codeSystem | ||
| − | |||
| | | | ||
|1..1 | |1..1 | ||
| − | | | + | |F |
| − | |fix | + | |fix: "1.2.276.0.76.5.404" |
|- | |- | ||
| Zeile 212: | Zeile 206: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|qualifier | |qualifier | ||
| − | |||
|CR | |CR | ||
|0..1 | |0..1 | ||
| Zeile 222: | Zeile 215: | ||
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| name | | name | ||
| − | |||
| CV | | CV | ||
| 1..1 | | 1..1 | ||
| required | | required | ||
| − | | | + | | Gleason 1 (Entdifferenzierungsgrad) |
|- | |- | ||
| Zeile 232: | Zeile 224: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|@code | |@code | ||
| − | |||
| | | | ||
|1..1 | |1..1 | ||
| − | | | + | |F |
| − | |fix | + | |fix:_ LOINC: "44641-9" |
|- | |- | ||
| Zeile 242: | Zeile 233: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|@codeSystem | |@codeSystem | ||
| − | |||
| | | | ||
|1..1 | |1..1 | ||
| − | | | + | |F |
| − | |fix: LOINC | + | |fix: LOINC: "2.16.840.1.113883.6.1" |
|- | |- | ||
| Zeile 252: | Zeile 242: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|value | |value | ||
| − | |||
|CV | |CV | ||
|1..1 | |1..1 | ||
|required | |required | ||
| − | | | + | |Code für den Gleason-Entdifferenzierungsgrad |
|- | |- | ||
| Zeile 262: | Zeile 251: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|@code | |@code | ||
| − | |||
| | | | ||
|1..1 | |1..1 | ||
| − | | | + | |F |
| − | |Codesystem 1.2.276.0.76.5.402 | + | |eigentlicher Code: Codesystem 1.2.276.0.76.5.402 |
|- | |- | ||
| Zeile 272: | Zeile 260: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|@codeSystem | |@codeSystem | ||
| − | |||
| | | | ||
|1..1 | |1..1 | ||
| − | | | + | |F |
| − | |fix | + | |fix: "1.2.276.0.76.5.402" |
|- | |- | ||
| Zeile 282: | Zeile 269: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|qualifier | |qualifier | ||
| − | |||
|CR | |CR | ||
|0..1 | |0..1 | ||
| Zeile 292: | Zeile 278: | ||
|bgcolor="ff8888"| act | |bgcolor="ff8888"| act | ||
| name | | name | ||
| − | |||
| CV | | CV | ||
| 1..1 | | 1..1 | ||
| required | | required | ||
| − | | | + | | Gleason 2 (Wachstumsmuster) |
|- | |- | ||
| Zeile 302: | Zeile 287: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|@code | |@code | ||
| − | |||
| | | | ||
|1..1 | |1..1 | ||
| − | | | + | |F |
| − | |fix | + | |fix: LOICN: "44642-7" |
|- | |- | ||
| Zeile 312: | Zeile 296: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|@codeSystem | |@codeSystem | ||
| − | |||
| | | | ||
|1..1 | |1..1 | ||
|required | |required | ||
| − | |fix: LOINC | + | |fix: LOINC: "2.16.840.1.113883.6.1" |
|- | |- | ||
| Zeile 322: | Zeile 305: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|value | |value | ||
| − | |||
|CV | |CV | ||
|1..1 | |1..1 | ||
|required | |required | ||
| − | | | + | |Code für das Gleason-Wachstumsmuster |
|- | |- | ||
| Zeile 332: | Zeile 314: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|@code | |@code | ||
| − | |||
| | | | ||
|1..1 | |1..1 | ||
|required | |required | ||
| − | |Codesystem 1.2.276.0.76.5.403 | + | |eigentlicher Code: Codesystem 1.2.276.0.76.5.403 |
|- | |- | ||
| Zeile 342: | Zeile 323: | ||
|bgcolor="ff8888"|act | |bgcolor="ff8888"|act | ||
|@codeSystem | |@codeSystem | ||
| − | |||
| | | | ||
|1..1 | |1..1 | ||
| − | | | + | |F |
| − | |fix | + | |fix: "1.2.276.0.76.5.403" |
|} | |} | ||
| + | |||
| + | ===Beispiel=== | ||
<syntaxhighlight lang="XML"> | <syntaxhighlight lang="XML"> | ||
| Zeile 388: | Zeile 370: | ||
</syntaxhighlight> | </syntaxhighlight> | ||
| + | ===Vokabular=== | ||
| + | |||
| + | ====Entdifferenzierungsgrad==== | ||
{| class="hl7table" | {| class="hl7table" | ||
| Zeile 414: | Zeile 399: | ||
Tabelle 31: Entdifferenzierungsgrad nach Gleason-Score<sup>17</sup> (OID 1.2.276.0.76.5.402) | Tabelle 31: Entdifferenzierungsgrad nach Gleason-Score<sup>17</sup> (OID 1.2.276.0.76.5.402) | ||
| + | ====Wachstumsmuster==== | ||
| + | |||
| + | {{WorkBox| | ||
| + | muss noch eingefügt werden | ||
| + | }} | ||
| − | ===Gleason Grading=== | + | ====Gleason Grading==== |
{| class="hl7table" | {| class="hl7table" | ||
Version vom 8. Juli 2013, 13:33 Uhr
Dieses Material ist Teil des Leitfadens Arztbrief 2.x.
|
Inhaltsverzeichnis
Entry: Gleason-Score
| Template-Metadaten | |
| Template-Typ | Entry |
| Template ID | 1.2.276.0.76.3.1.131.1.10.3.9 |
| genutztes Template | cdaab2:Assessment_Scales_(Entry) |
| abgeleitete Templates | <Verweis auf das Template> |
| Generelle Beschreibung | Gleason-Score (Prostatakarzinom) |
| allg. Erläuterung | < Verweis auf eine allgemeine Erläuterungsseite > |
| Ballotierungsstatus | < Status der Verabschiedung > |
| Erweiterbarkeit | offen oder geschlossen |
|
|
TODO: Abgleich mit dem Assessment-Scale Leitfaden ist erforderlich! Danach wird der Gesamt-Score in der Hauptobservation übermittelt, die zusätzlichen Details hingegen als Komponenten (COMP). |
|
|
TODO: Diagramm ergänzen |
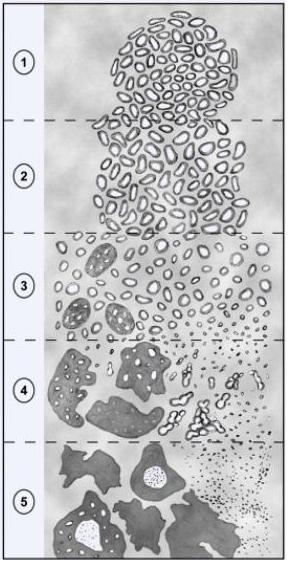
Der Gleason-Score dient zur Malignitätsbeurteilung des Prostatakarzinoms. Der Gesamtscore wird dabei aus der Addition zweier einzelner Beurteilungen (primäres Gleason-Muster) in einem komplizierten Verfahren der semiquantitativen Bewertung von
- Entdifferenzierungsgrad
- Wachstumsmuster
gebildet. Es gibt die primären Gleason-Muster 1-5 (aufsteigende Malignität).
Der Gleason Score setzt sich beim Resektionspräparat aus der Summe des häufigsten und des zweithäufigsten, bei der Stanzbiopsie des schlechtesten und des zweitschlechtesten Gleason-Musters zusammen. Ist nur ein Muster erkennbar (häufig in Stanzbiopsien), so wird der Score aus dem verdoppelten Musterwert gebildet. An der Stanze sind Gleason-Muster <3 nicht zugelassen.
Damit ergeben sich Gesamtwerte von 2 bis 10 (s. hierzu auch Scores & Assessment DSTU). Seit 2005 gilt die leicht modifizierte Gleason-Klassifizierung (nach Epstein \[Helpap2007\]), d.h. es ist hier die Angabe des verwendeten Verfahrens – genauer die zu verwendenden Codes - erforderlich.
Attribute
Beispiel
<observation classCode="OBS" moodCode="EVN">
<templateId root="1.2.276.0.76.3.1.131.1.10.3.9"/>
<id extension="1234009"
root="1.2.276.0.76.3.1.131.1.4.3.9999.9999.999919"/>
<code code="35266-6"
codeSystem="2.16.840.1.113883.6.1"/>
<text>
Gleason-Score: 4+5=9
</text>
<effectiveTime value="20110326"/>
<value xsi:type="CD" code="9" codeSystem="1.2.276.0.76.5.404">
</value>
<!-- Einzelwerte zum Score: 1. Wert -->
<entryRelationship typeCode='COMP' inversionInd='false'>
<observation moodCode='EVN' classCode='OBS'>
<code code='44641-9' displayName=''
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC' />
<statusCode code='completed'/>
<value xsi:type='CD' code='4' codeSystem='1.2.276.0.76.5.402'/>
</value>
</observation>
</entryRelationship>
<!-- Einzelwerte zum Score: 2. Wert -->
<entryRelationship typeCode='COMP' inversionInd='false'>
<observation moodCode='EVN' classCode='OBS'>
<code code='44642-7' displayName=''
codeSystem='2.16.840.1.113883.6.1' codeSystemName='LOINC' />
<statusCode code='completed'/>
<value xsi:type='CD' code='5' codeSystem='1.2.276.0.76.5.403'/>
</value>
</observation>
</entryRelationship>
</observation>
Vokabular
Entdifferenzierungsgrad
| Code | Description | Bedeutung |
|---|---|---|
| 1 | Round to oval equal individual glands, lying close to each other, sharply demarcated from the surrounding area | Runde bis ovale gleich große Einzeldrüsen, dicht nebeneinander liegend, scharf gegen die Umgebung abgegrenzt |
| 2 | Slightly less uniform single glands, separated by small amounts of stroma, less sharply defined tumor margin | Etwas weniger uniforme Einzeldrüsen, getrennt durch geringe Mengen von Stroma, weniger scharf begrenzter Tumorrand |
| 3 | a) Irregularly large and irregularly shaped glands, usually with abundant stroma, sometimes also stored tightly irregular and indistinct tumor border | Unregelmäßig große und unregelmäßig gestaltete Drüsen mit gewöhnlich reichlicherem Stroma, gelegentlich auch dicht gelagert, unregelmäßige und unscharfe Tumorgrenze |
| b) Papillary or cribriform structures, sometimes in large gang-like formations |
Papilläre oder kribriforme Strukturen, z.T. in großen gangähnlichen Bildungen | |
| 4 | a) Large irregular Epithelformationen by glandular fusion ("fused glands") and branched glands with irregular infiltration into the surrounding area | Große unregelmäßige Epithelformationen durch Drüsenverschmelzung („fused glands") sowie verzweigte Drüsen mit unregelmäßiger Infiltration in die Umgebung |
| b) Adenocarcinoma with prominent clear cytoplasm similar to clear cell adenocarcinomas of the kidney | Adenokarzinom mit ausgeprägt klarem Zytoplasma ähnlich hellzelligen Adenokarzinomen der Niere | |
| 5 | a) Circumscribed round epithelial clusters with mostly solid and cribriform construction, usually with central necrosis (comedo carcinoma-like) | Scharf begrenzte runde Epithelhaufen mit meist solidem und kribriformem Bau, gewöhnlich mit zentraler Nekrose
(komedo-karzinomähnlich) |
| b) Irregularly shaped formations of an undifferentiated carcinoma, which only just discernible glandular formation or is identified vacuoles (signet ring-like) than adenocarcinoma | Unregelmäßig begrenzte Formationen eines undifferenzierten Karzinoms, das nur durch gerade noch erkennbare Drüsenbildung
oder Vakuolen (siegelringähnlich) als Adenokarzinom zu identifizieren ist |
Tabelle 31: Entdifferenzierungsgrad nach Gleason-Score17 (OID 1.2.276.0.76.5.402)
Wachstumsmuster
|
|
muss noch eingefügt werden |
Gleason Grading
| Gleason-Score | Gleason Grading | Bedeutung |
|---|---|---|
| 2 | G1 | gute Prognose |
| 3 | G1 | gute Prognose |
| 4 | G1 | gute Prognose |
| 5 | G2 | gute Prognose |
| 6 | G2 | gute Prognose |
| 7 | G2-G3 | schlechte Prognose |
| 7a | G2-G3 (ergibt sich aus 3+4) | schlechte Prognose |
| 7b | G2-G3 (ergibt sich aus 4+3) | schlechte Prognose |
| 8 | G3 | schlechte Prognose |
| 9 | G3 | schlechte Prognose |
| 10 | G3 | schlechte Prognose |
Tabelle 32: Grading nach Gleason-Score (OID 1.2.276.0.76.5.404)